+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2gbh | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | NMR structure of stem region of helix-35 of 23S E.coli ribosomal RNA (residues 736-760) | ||||||||||||||||||
 要素 要素 | 5'-R(* キーワード キーワードRNA / RDC / rCSA | 機能・相同性 | RNA / RNA (> 10) |  機能・相同性情報 機能・相同性情報手法 | 溶液NMR / Cartesian simulated annealing |  データ登録者 データ登録者Bax, A. / Boisbouvier, J. / Bryce, D. / Grishaev, A. / Jaroniec, C. / Miclet, E. / Nikonovicz, E. / O'Neil-Cabello, E. / Ying, J. |  引用 引用 ジャーナル: J.Am.Chem.Soc. / 年: 2004 ジャーナル: J.Am.Chem.Soc. / 年: 2004タイトル: Measurement of five dipolar couplings from a single 3D NMR multiplet applied to the study of RNA dynamics. 著者: O'Neil-Cabello, E. / Bryce, D.L. / Nikonowicz, E.P. / Bax, A. 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2gbh.cif.gz 2gbh.cif.gz | 57.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2gbh.ent.gz pdb2gbh.ent.gz | 43.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2gbh.json.gz 2gbh.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2gbh_validation.pdf.gz 2gbh_validation.pdf.gz | 304.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2gbh_full_validation.pdf.gz 2gbh_full_validation.pdf.gz | 316.3 KB | 表示 | |
| XML形式データ |  2gbh_validation.xml.gz 2gbh_validation.xml.gz | 2 KB | 表示 | |
| CIF形式データ |  2gbh_validation.cif.gz 2gbh_validation.cif.gz | 2.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/gb/2gbh https://data.pdbj.org/pub/pdb/validation_reports/gb/2gbh ftp://data.pdbj.org/pub/pdb/validation_reports/gb/2gbh ftp://data.pdbj.org/pub/pdb/validation_reports/gb/2gbh | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 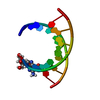
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 7637.666 Da / 分子数: 1 / 由来タイプ: 合成 詳細: The sequence of this RNA naturally exists in 23S Ribosomal RNA of E.Coli. |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR |
|---|---|
| NMR実験 | タイプ: RDC measurement |
- 試料調製
試料調製
| 詳細 | 内容: 1.5mM helix-35psi U-15N,13C; 17mM NaCl, 17mM phosphate buffer; 99% D2O 溶媒系: 99% D2O |
|---|---|
| 試料状態 | イオン強度: 17 mM NaCl, 17 mM phosphate / pH: 6.8 / 圧: ambient / 温度: 298 K |
-NMR測定
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射波長 | 相対比: 1 | |||||||||||||||
| NMRスペクトロメーター |
|
- 解析
解析
| NMR software | 名称:  Xplor-NIH / バージョン: 2.9.4 / 開発者: Schwieters, Kuszewski, Tjandra, Clore / 分類: 精密化 Xplor-NIH / バージョン: 2.9.4 / 開発者: Schwieters, Kuszewski, Tjandra, Clore / 分類: 精密化 |
|---|---|
| 精密化 | 手法: Cartesian simulated annealing / ソフトェア番号: 1 詳細: Experimental restraints for the helix-35 stem are 277 RDCs, 13 31P anisotropic shifts, 41 dihedral angles and 188 NOEs. During the refinement the geometries of selected ribose rings were kept ...詳細: Experimental restraints for the helix-35 stem are 277 RDCs, 13 31P anisotropic shifts, 41 dihedral angles and 188 NOEs. During the refinement the geometries of selected ribose rings were kept identical with the NCS restraint terms. Attractive non-bonded potentials were employed. No database-derived terms were used. The parameter file was modified with nucleotide type-specific values for distances, angles and improper torsions. Cross-validation statistics on the groups of 1/4 of ribose 1-bond C-H RDCs corresponds to an average Q-factor of 0.135. |
| 代表構造 | 選択基準: lowest energy |
| NMRアンサンブル | コンフォーマー選択の基準: all calculated structures submitted 計算したコンフォーマーの数: 5 / 登録したコンフォーマーの数: 5 |
 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj





























