+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2fzp | ||||||
|---|---|---|---|---|---|---|---|
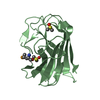
| タイトル | Crystal structure of the USP8 interaction domain of human NRDP1 | ||||||
 要素 要素 | ring finger protein 41 isoform 1 | ||||||
 キーワード キーワード | LIGASE / E3 Ligase / protein ubiquitination / Structural Genomics / Structural Genomics Consortium / SGC | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of lymphocyte differentiation / interleukin-3 receptor binding / erythropoietin receptor binding / regulation of myeloid cell differentiation / endoplasmic reticulum tubular network / negative regulation of mitophagy / regulation of reactive oxygen species metabolic process / regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / regulation of MAPK cascade / protein autoubiquitination ...regulation of lymphocyte differentiation / interleukin-3 receptor binding / erythropoietin receptor binding / regulation of myeloid cell differentiation / endoplasmic reticulum tubular network / negative regulation of mitophagy / regulation of reactive oxygen species metabolic process / regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / regulation of MAPK cascade / protein autoubiquitination / proteasomal protein catabolic process / extrinsic apoptotic signaling pathway / Downregulation of ERBB2:ERBB3 signaling / negative regulation of cell migration / RING-type E3 ubiquitin transferase / receptor tyrosine kinase binding / small GTPase binding / autophagy / protein polyubiquitination / ubiquitin-protein transferase activity / positive regulation of protein catabolic process / positive regulation of reactive oxygen species metabolic process / ubiquitin protein ligase activity / Antigen processing: Ubiquitination & Proteasome degradation / protein ubiquitination / protein domain specific binding / negative regulation of cell population proliferation / perinuclear region of cytoplasm / zinc ion binding / identical protein binding / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.87 Å 分子置換 / 解像度: 1.87 Å | ||||||
 データ登録者 データ登録者 | Walker, J.R. / Avvakumov, G.V. / Xue, S. / Newman, E.M. / Butler-Cole, C. / Finerty Jr., P.J. / Weigelt, J. / Sundstrom, M. / Arrowsmith, C. / Edwards, A. ...Walker, J.R. / Avvakumov, G.V. / Xue, S. / Newman, E.M. / Butler-Cole, C. / Finerty Jr., P.J. / Weigelt, J. / Sundstrom, M. / Arrowsmith, C. / Edwards, A. / Bochkarev, A. / Dhe-Paganon, S. / Structural Genomics Consortium (SGC) | ||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2006 ジャーナル: J.Biol.Chem. / 年: 2006タイトル: Amino-terminal Dimerization, NRDP1-Rhodanese Interaction, and Inhibited Catalytic Domain Conformation of the Ubiquitin-specific Protease 8 (USP8). 著者: Avvakumov, G.V. / Walker, J.R. / Xue, S. / Finerty Jr., P.J. / Mackenzie, F. / Newman, E.M. / Dhe-Paganon, S. #1: ジャーナル: Mol.Cell.Biol. / 年: 2004 タイトル: Stabilization of the E3 ubiquitin ligase Nrdp1 by the deubiquitinating enzyme USP8. 著者: Wu, X. / Yen, L. / Irwin, L. / Sweeney, C. / Carraway, K.L. #2: ジャーナル: Embo J. / 年: 2004 タイトル: Nrdp1-mediated degradation of the gigantic IAP, BRUCE, is a novel pathway for triggering apoptosis. 著者: Qiu, X.B. / Markant, S.L. / Yuan, J. / Goldberg, A.L. #3: ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2002 タイトル: An RBCC protein implicated in maintenance of steady-state neuregulin receptor levels. 著者: Diamonti, A.J. / Guy, P.M. / Ivanof, C. / Wong, K. / Sweeney, C. / Carraway, K.L. #4: ジャーナル: Blood Cells Mol.Dis. / 年: 2001 タイトル: FLRF, a novel evolutionarily conserved RING finger gene, is differentially expressed in mouse fetal and adult hematopoietic stem cells and progenitors. 著者: Abdullah, J.M. / Li, X. / Nachtman, R.G. / Jurecic, R. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2fzp.cif.gz 2fzp.cif.gz | 43.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2fzp.ent.gz pdb2fzp.ent.gz | 31.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2fzp.json.gz 2fzp.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2fzp_validation.pdf.gz 2fzp_validation.pdf.gz | 420.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2fzp_full_validation.pdf.gz 2fzp_full_validation.pdf.gz | 422 KB | 表示 | |
| XML形式データ |  2fzp_validation.xml.gz 2fzp_validation.xml.gz | 8.2 KB | 表示 | |
| CIF形式データ |  2fzp_validation.cif.gz 2fzp_validation.cif.gz | 10.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/fz/2fzp https://data.pdbj.org/pub/pdb/validation_reports/fz/2fzp ftp://data.pdbj.org/pub/pdb/validation_reports/fz/2fzp ftp://data.pdbj.org/pub/pdb/validation_reports/fz/2fzp | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 16172.340 Da / 分子数: 1 / 断片: USP8 interaction Domain / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: RNF41 / プラスミド: PET28-LIC / 生物種 (発現宿主): Escherichia coli / 発現宿主: Homo sapiens (ヒト) / 遺伝子: RNF41 / プラスミド: PET28-LIC / 生物種 (発現宿主): Escherichia coli / 発現宿主:  参照: UniProt: Q9H4P4, 合成酵素; C-N結合を形成; 酸-D-アミノ酸リガーゼ(ペプチド合成) |
|---|---|
| #2: 水 | ChemComp-HOH / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.1 Å3/Da / 溶媒含有率: 41.49 % |
|---|---|
| 結晶化 | 温度: 298 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7 詳細: 1.8M (NH4)2SO4, 0.1M HEPES pH 7.0,0.2M NaCl, 0.001M DTT, VAPOR DIFFUSION, HANGING DROP, temperature 298.0K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 19-BM / 波長: 1.00769 Å / ビームライン: 19-BM / 波長: 1.00769 Å |
| 検出器 | タイプ: SBC-3 / 検出器: CCD / 日付: 2005年11月4日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.00769 Å / 相対比: 1 |
| 反射 | 解像度: 1.87→27.36 Å / Num. all: 12100 / Num. obs: 12100 / % possible obs: 99 % / Observed criterion σ(F): 0 / Observed criterion σ(I): -3 / 冗長度: 4.3 % / Rmerge(I) obs: 0.14 / Net I/σ(I): 8.97 |
| 反射 シェル | 解像度: 1.87→1.94 Å / 冗長度: 4.4 % / Rmerge(I) obs: 0.653 / Mean I/σ(I) obs: 1.52 / Num. unique all: 1173 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 / 解像度: 1.87→27.36 Å / Cor.coef. Fo:Fc: 0.953 / Cor.coef. Fo:Fc free: 0.918 / SU B: 5.882 / SU ML: 0.088 / TLS residual ADP flag: LIKELY RESIDUAL / 交差検証法: THROUGHOUT / ESU R: 0.137 / ESU R Free: 0.143 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS 分子置換 / 解像度: 1.87→27.36 Å / Cor.coef. Fo:Fc: 0.953 / Cor.coef. Fo:Fc free: 0.918 / SU B: 5.882 / SU ML: 0.088 / TLS residual ADP flag: LIKELY RESIDUAL / 交差検証法: THROUGHOUT / ESU R: 0.137 / ESU R Free: 0.143 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: BABINET MODEL WITH MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 15.627 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.87→27.36 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.873→1.922 Å / Total num. of bins used: 20
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj





