| 登録情報 | データベース: PDB / ID: 2exg
|
|---|
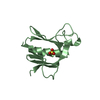
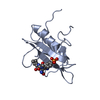
| タイトル | Making Protein-Protein Interactions Drugable: Discovery of Low-Molecular-Weight Ligands for the AF6 PDZ Domain |
|---|
 要素 要素 | Afadin |
|---|
 キーワード キーワード | CELL ADHESION / Inhibitors of protein-protein interactions / low-molecular-weight ligands / protein structure / NMR screening / PDZ domains |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
establishment of protein localization to plasma membrane / positive regulation of cell-cell adhesion mediated by cadherin / pore complex assembly / establishment of endothelial intestinal barrier / positive regulation of cell-cell adhesion / bicellular tight junction assembly / cell-cell contact zone / cell-cell adhesion mediated by cadherin / Adherens junctions interactions / tight junction ...establishment of protein localization to plasma membrane / positive regulation of cell-cell adhesion mediated by cadherin / pore complex assembly / establishment of endothelial intestinal barrier / positive regulation of cell-cell adhesion / bicellular tight junction assembly / cell-cell contact zone / cell-cell adhesion mediated by cadherin / Adherens junctions interactions / tight junction / pore complex / cell adhesion molecule binding / negative regulation of cell migration / adherens junction / small GTPase binding / actin filament binding / cell-cell junction / cell junction / cell-cell signaling / regulation of protein localization / cell adhesion / nuclear speck / cadherin binding / positive regulation of gene expression / signal transduction / nucleoplasm / plasma membrane / cytosol類似検索 - 分子機能 : / Afadin, cargo binding domain / Ras association (RalGDS/AF-6) domain / Ras association (RalGDS/AF-6) domain / Dilute domain / DIL domain / Dilute domain profile. / DIL / Ras-associating (RA) domain profile. / Ras-associating (RA) domain ...: / Afadin, cargo binding domain / Ras association (RalGDS/AF-6) domain / Ras association (RalGDS/AF-6) domain / Dilute domain / DIL domain / Dilute domain profile. / DIL / Ras-associating (RA) domain profile. / Ras-associating (RA) domain / Forkhead associated domain / FHA domain / Forkhead-associated (FHA) domain / SMAD/FHA domain superfamily / PDZ domain / Pdz3 Domain / PDZ domain / PDZ domain profile. / Domain present in PSD-95, Dlg, and ZO-1/2. / PDZ domain / PDZ superfamily / Ubiquitin-like domain superfamily / Roll / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR / Torsional angle dynamics |
|---|
 データ登録者 データ登録者 | Joshi, M. / Vargas, C. / Boisguerin, P. / Krause, G. / Schade, M. / Oschkinat, H. |
|---|
 引用 引用 |  ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2006 ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2006
タイトル: Discovery of low-molecular-weight ligands for the AF6 PDZ domain.
著者: Joshi, M. / Vargas, C. / Boisguerin, P. / Diehl, A. / Krause, G. / Schmieder, P. / Moelling, K. / Hagen, V. / Schade, M. / Oschkinat, H. |
|---|
| 履歴 | | 登録 | 2005年11月8日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2006年10月17日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年5月1日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2022年3月9日 | Group: Data collection / Database references / Derived calculations
カテゴリ: database_2 / pdbx_nmr_software ...database_2 / pdbx_nmr_software / pdbx_struct_assembly / pdbx_struct_oper_list / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
| 改定 1.4 | 2024年5月22日 | Group: Data collection / カテゴリ: chem_comp_atom / chem_comp_bond |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2006
ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2006 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2exg.cif.gz
2exg.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2exg.ent.gz
pdb2exg.ent.gz PDB形式
PDB形式 2exg.json.gz
2exg.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2exg_validation.pdf.gz
2exg_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2exg_full_validation.pdf.gz
2exg_full_validation.pdf.gz 2exg_validation.xml.gz
2exg_validation.xml.gz 2exg_validation.cif.gz
2exg_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/ex/2exg
https://data.pdbj.org/pub/pdb/validation_reports/ex/2exg ftp://data.pdbj.org/pub/pdb/validation_reports/ex/2exg
ftp://data.pdbj.org/pub/pdb/validation_reports/ex/2exg リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 発現宿主:
Homo sapiens (ヒト) / 発現宿主: 
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj



