+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2erl | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | PHEROMONE ER-1 FROM | |||||||||
 要素 要素 | MATING PHEROMONE ER-1 | |||||||||
 キーワード キーワード | PHEROMONE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Euplotes raikovi (真核生物) Euplotes raikovi (真核生物) | |||||||||
| 手法 |  X線回折 / 解像度: 1 Å X線回折 / 解像度: 1 Å | |||||||||
 データ登録者 データ登録者 | Anderson, D.H. / Weiss, M.S. / Eisenberg, D. | |||||||||
 引用 引用 |  ジャーナル: Acta Crystallogr.,Sect.D / 年: 1996 ジャーナル: Acta Crystallogr.,Sect.D / 年: 1996タイトル: A challenging case for protein crystal structure determination: the mating pheromone Er-1 from Euplotes raikovi. 著者: Anderson, D.H. / Weiss, M.S. / Eisenberg, D. #1:  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1995 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1995タイトル: A Cooperative Model for Receptor Recognition and Cell Adhesion: Evidence from the Molecular Packing in the 1.6-A Crystal Structure of the Pheromone Er-1 from the Ciliated Protozoan Euplotes Raikovi 著者: Weiss, M.S. / Anderson, D.H. / Raffioni, S. / Bradshaw, R.A. / Ortenzi, C. / Luporini, P. / Eisenberg, D. #2:  ジャーナル: Protein Sci. / 年: 1994 ジャーナル: Protein Sci. / 年: 1994タイトル: The NMR Solution Structure of the Pheromone Er-1 from the Ciliated Protozoan Euplotes Raikovi 著者: Mronga, S. / Luginbuhl, P. / Brown, L.R. / Ortenzi, C. / Luporini, P. / Bradshaw, R.A. / Wuthrich, K. #3:  ジャーナル: J.Mol.Biol. / 年: 1990 ジャーナル: J.Mol.Biol. / 年: 1990タイトル: Crystallization of the Euplotes Raikovi Mating Pheromone Er-1 著者: Anderson, D. / Raffioni, S. / Luporini, P. / Bradshaw, R.A. / Eisenberg, D. #4:  ジャーナル: J.Biol.Chem. / 年: 1988 ジャーナル: J.Biol.Chem. / 年: 1988タイトル: Primary Structure of the Mating Pheromone Er-1 of the Ciliate Euplotes Raikovi 著者: Raffioni, S. / Luporini, P. / Chait, B.T. / Disper, S.S. / Bradshaw, R.A. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2erl.cif.gz 2erl.cif.gz | 38.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2erl.ent.gz pdb2erl.ent.gz | 27 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2erl.json.gz 2erl.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2erl_validation.pdf.gz 2erl_validation.pdf.gz | 416.4 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2erl_full_validation.pdf.gz 2erl_full_validation.pdf.gz | 416.4 KB | 表示 | |
| XML形式データ |  2erl_validation.xml.gz 2erl_validation.xml.gz | 4.3 KB | 表示 | |
| CIF形式データ |  2erl_validation.cif.gz 2erl_validation.cif.gz | 5.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/er/2erl https://data.pdbj.org/pub/pdb/validation_reports/er/2erl ftp://data.pdbj.org/pub/pdb/validation_reports/er/2erl ftp://data.pdbj.org/pub/pdb/validation_reports/er/2erl | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 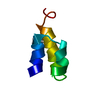
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 4417.881 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Euplotes raikovi (真核生物) / 参照: UniProt: P10774 Euplotes raikovi (真核生物) / 参照: UniProt: P10774 |
|---|---|
| #2: 化合物 | ChemComp-EOH / |
| #3: 水 | ChemComp-HOH / |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.53 Å3/Da / 溶媒含有率: 19.6 % | ||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶 | *PLUS 溶媒含有率: 20 % | ||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 298-301 K / pH: 3.5 / 手法: 蒸気拡散法, ハンギングドロップ法 | ||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 放射光源 | 波長: 0.7107 |
|---|---|
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1993年9月20日 |
| 放射 | 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.7107 Å / 相対比: 1 |
| 反射 | 解像度: 1→25.26 Å / Num. obs: 13659 / % possible obs: 93.8 % / Observed criterion σ(I): 0 / 冗長度: 7.5 % / Rmerge(I) obs: 0.054 |
| 反射 | *PLUS 最低解像度: 8 Å / Num. obs: 13461 / % possible obs: 92.4 % / Num. measured all: 101188 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 1→25.25 Å / Num. parameters: 3121 / Num. restraintsaints: 3883 / σ(F): 0 立体化学のターゲット値: ENGH AND HUBER (1991) ACTA CRYST. A47, 392-400 詳細: ANISOTROPIC TEMPERATURE FACTORS HAVE BEEN REFINED FOR THE NON-HYDROGEN ATOMS IN THIS ENTRY. HYDROGEN ATOMS HAVE ISOTROPIC U'S THAT ARE 1.2 TIMES THE ISOTROPIC U'S OF THE ATOMS ON WHICH THEY ...詳細: ANISOTROPIC TEMPERATURE FACTORS HAVE BEEN REFINED FOR THE NON-HYDROGEN ATOMS IN THIS ENTRY. HYDROGEN ATOMS HAVE ISOTROPIC U'S THAT ARE 1.2 TIMES THE ISOTROPIC U'S OF THE ATOMS ON WHICH THEY RIDE. THE QUANTITIES ON THE ANISOU RECORD FOR EACH HYDROGEN ATOM ARE THE EQUIVALENT-TO-ISOTROPIC U'S.
| |||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: DRIESSEN, ET AL., (1989) J.APPL.CRYST. 22, 510-516 | |||||||||||||||||||||||||||||||||
| Refine analyze | Num. disordered residues: 4 / Occupancy sum non hydrogen: 1 | |||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1→25.25 Å
| |||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: SHELXL / バージョン: 93 / 分類: refinement | |||||||||||||||||||||||||||||||||
| 精密化 | *PLUS Rfactor Rwork: 0.129 / 最低解像度: 26 Å / % reflection Rfree: 10 % / Rfactor obs: 0.1292 | |||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | |||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | |||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS タイプ: s_plane_restr / Dev ideal: 0.206 |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj







