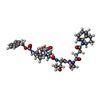
登録情報 データベース : PDB / ID : 2c2zタイトル Crystal structure of caspase-8 in complex with aza-peptide Michael acceptor inhibitor AZA-PEPTIDE INHIBITOR (5S, 8R, 11S)-8-(2-CARBOXYETHYL) -14-[4-(3,4-DIHYDROQUINOLIN-1(2H)-YL)-4-OXOBUTANOYL] -11-[(1R)-1-HYDROXYETHYL]-5-(2-METHYLPROPYL)-3,6,9,12-TETRAOXO -1-PHENYL-2-OXA-4,7,10,13,14-PENTAAZAHEXADECAN-16-OIC ACID CASPASE-8 P10 SUBUNIT CASPASE-8 P18 SUBUNIT キーワード / / / / / / / / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 HOMO SAPIENS (ヒト)SYNTHETIC CONSTRUCT (人工物) 手法 / / OTHER / 解像度 : 1.95 Å データ登録者 Ganesan, R. / Jelakovic, S. / Ekici, O.D. / Li, Z.Z. / James, K.E. / Asgian, J.L. / Campbell, A.J. / Mikolajczyk, J. / Salvesen, G.S. / Powers, J.C. / Gruetter, M.G. ジャーナル : J.Med.Chem. / 年 : 2006タイトル : Design, Synthesis, and Evaluation of Aza-Peptide Michael Acceptors as Selective and Potent Inhibitors of Caspases-2, -3, -6, -7, -8, -9, and - 10.著者 : Ekici, O.D. / Li, Z.Z. / Campbell, A.J. / James, K.E. / Asgian, J.L. / Mikolajczyk, J. / Salvesen, G.S. / Ganesan, R. / Jelakovic, S. / Grutter, M.G. / Powers, J.C. 履歴 登録 2005年10月2日 登録サイト / 処理サイト 改定 1.0 2006年9月20日 Provider / タイプ 改定 1.1 2011年7月13日 Group Atomic model / Database references ... Atomic model / Database references / Derived calculations / Non-polymer description / Structure summary / Version format compliance 改定 1.2 2011年7月20日 Group / Structure summary改定 1.3 2012年11月30日 Group 改定 1.4 2017年2月8日 Group
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 HOMO SAPIENS (ヒト)
HOMO SAPIENS (ヒト) X線回折 /
X線回折 /  シンクロトロン / OTHER / 解像度: 1.95 Å
シンクロトロン / OTHER / 解像度: 1.95 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Med.Chem. / 年: 2006
ジャーナル: J.Med.Chem. / 年: 2006 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2c2z.cif.gz
2c2z.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2c2z.ent.gz
pdb2c2z.ent.gz PDB形式
PDB形式 2c2z.json.gz
2c2z.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2c2z_validation.pdf.gz
2c2z_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2c2z_full_validation.pdf.gz
2c2z_full_validation.pdf.gz 2c2z_validation.xml.gz
2c2z_validation.xml.gz 2c2z_validation.cif.gz
2c2z_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/c2/2c2z
https://data.pdbj.org/pub/pdb/validation_reports/c2/2c2z ftp://data.pdbj.org/pub/pdb/validation_reports/c2/2c2z
ftp://data.pdbj.org/pub/pdb/validation_reports/c2/2c2z リンク
リンク 集合体
集合体

 要素
要素 HOMO SAPIENS (ヒト) / プラスミド: PET11D / 発現宿主:
HOMO SAPIENS (ヒト) / プラスミド: PET11D / 発現宿主: 
 HOMO SAPIENS (ヒト) / プラスミド: PET11D / 発現宿主:
HOMO SAPIENS (ヒト) / プラスミド: PET11D / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SLS
SLS  / ビームライン: X06SA / 波長: 0.90001
/ ビームライン: X06SA / 波長: 0.90001  解析
解析 ムービー
ムービー コントローラー
コントローラー






















 PDBj
PDBj