+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2abk | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
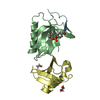
| タイトル | REFINEMENT OF THE NATIVE STRUCTURE OF ENDONUCLEASE III TO A RESOLUTION OF 1.85 ANGSTROM | |||||||||
 要素 要素 | ENDONUCLEASE III | |||||||||
 キーワード キーワード | ENDONUCLEASE / DNA-REPAIR / DNA GLYCOSYLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報oxidized pyrimidine nucleobase lesion DNA N-glycosylase activity / base-excision repair, AP site formation via deaminated base removal / base-excision repair, AP site formation / DNA N-glycosylase activity / uracil DNA N-glycosylase activity / DNA-(apurinic or apyrimidinic site) endonuclease activity / class I DNA-(apurinic or apyrimidinic site) endonuclease activity / DNA-(apurinic or apyrimidinic site) lyase / cellular response to UV / 4 iron, 4 sulfur cluster binding ...oxidized pyrimidine nucleobase lesion DNA N-glycosylase activity / base-excision repair, AP site formation via deaminated base removal / base-excision repair, AP site formation / DNA N-glycosylase activity / uracil DNA N-glycosylase activity / DNA-(apurinic or apyrimidinic site) endonuclease activity / class I DNA-(apurinic or apyrimidinic site) endonuclease activity / DNA-(apurinic or apyrimidinic site) lyase / cellular response to UV / 4 iron, 4 sulfur cluster binding / DNA binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 |  X線回折 / 解像度: 1.85 Å X線回折 / 解像度: 1.85 Å | |||||||||
 データ登録者 データ登録者 | Thayer, M.M. / Tainer, J.A. | |||||||||
 引用 引用 |  ジャーナル: EMBO J. / 年: 1995 ジャーナル: EMBO J. / 年: 1995タイトル: Novel DNA binding motifs in the DNA repair enzyme endonuclease III crystal structure. 著者: Thayer, M.M. / Ahern, H. / Xing, D. / Cunningham, R.P. / Tainer, J.A. #1:  ジャーナル: J.Mol.Biol. / 年: 1992 ジャーナル: J.Mol.Biol. / 年: 1992タイトル: Crystallization and Crystallographic Characterization of the Iron-Sulfur Containing DNA-Repair Enzyme Endonuclease III from Escherichia Coli Enzyme Endonuclease III 著者: Kuo, C.-F. / Mcree, D.E. / Cunningham, R.P. / Tainer, J.A. #2:  ジャーナル: Science / 年: 1992 ジャーナル: Science / 年: 1992タイトル: Atomic Structure of the DNA Repair [4Fe-4S] Enzyme Endonuclease III 著者: Kuo, C.-F. / Mcree, D.E. / Fisher, C.L. / Handley, S. / Cunningham, R.P. / Tainer, J.A. | |||||||||
| 履歴 |
| |||||||||
| Remark 650 | HELIX DETERMINATION METHOD: KABSCH AND SANDER AS IMPLEMENTED BY PROCHECK FOLLOWED BY VISUAL ...HELIX DETERMINATION METHOD: KABSCH AND SANDER AS IMPLEMENTED BY PROCHECK FOLLOWED BY VISUAL INSPECTION OF MODEL. TURN KABSCH AND SANDER IN PROCHECK TURN_ID: FG, HAIRPIN OF HELIX HAIRPIN HELIX MOTIF. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2abk.cif.gz 2abk.cif.gz | 55.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2abk.ent.gz pdb2abk.ent.gz | 40.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2abk.json.gz 2abk.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2abk_validation.pdf.gz 2abk_validation.pdf.gz | 381.4 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2abk_full_validation.pdf.gz 2abk_full_validation.pdf.gz | 382.7 KB | 表示 | |
| XML形式データ |  2abk_validation.xml.gz 2abk_validation.xml.gz | 5.8 KB | 表示 | |
| CIF形式データ |  2abk_validation.cif.gz 2abk_validation.cif.gz | 8.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ab/2abk https://data.pdbj.org/pub/pdb/validation_reports/ab/2abk ftp://data.pdbj.org/pub/pdb/validation_reports/ab/2abk ftp://data.pdbj.org/pub/pdb/validation_reports/ab/2abk | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 23595.398 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  参照: UniProt: P0AB83, DNA-(apurinic or apyrimidinic site) lyase |
|---|---|
| #2: 化合物 | ChemComp-SF4 / |
| #3: 水 | ChemComp-HOH / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.93 Å3/Da / 溶媒含有率: 58.07 % | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | *PLUS pH: 7 / 手法: unknown | ||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 放射光源 | 波長: 1.5418 Å |
|---|---|
| 検出器 | タイプ: XENTRONICS / 検出器: AREA DETECTOR / 日付: 1993年5月18日 |
| 放射 | 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 1.85→20 Å / Num. obs: 27787 / % possible obs: 99.8 % / Observed criterion σ(I): 0 / 冗長度: 10 % / Rmerge(I) obs: 0.07 |
| 反射 | *PLUS Rmerge(I) obs: 0.07 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 1.85→10 Å / σ(F): 2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 23.7 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.85→10 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / バージョン: 3 / 分類: refinement X-PLOR / バージョン: 3 / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj



