+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1zbn | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Solution structure of BIV TAR hairpin complexed to JDV Tat arginine-rich motif | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | RNA binding protein/RNA / RNA-peptide complex / RNA binding protein-RNA COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of viral transcription / host cell nucleolus / RNA-binding transcription regulator activity / RNA binding 類似検索 - 分子機能 | ||||||
| 手法 | 溶液NMR / distance geometry, restrained molecular dynamics | ||||||
 データ登録者 データ登録者 | Calabro, V. / Daugherty, M.D. / Frankel, A.D. | ||||||
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2005 ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2005タイトル: A single intermolecular contact mediates intramolecular stabilization of both RNA and protein. 著者: Calabro, V. / Daugherty, M.D. / Frankel, A.D. #1:  ジャーナル: Mol.Cell / 年: 2000 ジャーナル: Mol.Cell / 年: 2000タイトル: An RNA-binding chameleon 著者: Smith, C.A. / Calabro, V. / Frankel, A.D. #2:  ジャーナル: Science / 年: 1995 ジャーナル: Science / 年: 1995タイトル: Solution structure of a bovine immunodeficiency virus Tat-TAR peptide-RNA complex 著者: Puglisi, J.D. / Chen, L. / Blanchard, S. / Frankel, A.D. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1zbn.cif.gz 1zbn.cif.gz | 243.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1zbn.ent.gz pdb1zbn.ent.gz | 201.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1zbn.json.gz 1zbn.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1zbn_validation.pdf.gz 1zbn_validation.pdf.gz | 364.8 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1zbn_full_validation.pdf.gz 1zbn_full_validation.pdf.gz | 504.3 KB | 表示 | |
| XML形式データ |  1zbn_validation.xml.gz 1zbn_validation.xml.gz | 19.6 KB | 表示 | |
| CIF形式データ |  1zbn_validation.cif.gz 1zbn_validation.cif.gz | 30.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/zb/1zbn https://data.pdbj.org/pub/pdb/validation_reports/zb/1zbn ftp://data.pdbj.org/pub/pdb/validation_reports/zb/1zbn ftp://data.pdbj.org/pub/pdb/validation_reports/zb/1zbn | HTTPS FTP |
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 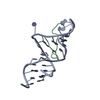
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 8923.310 Da / 分子数: 1 / 変異: A4G, U31C / 由来タイプ: 合成 詳細: This sequence occurs naturally in Bovine immunodeficiency virus (BIV). |
|---|---|
| #2: タンパク質・ペプチド | 分子量: 2064.493 Da / 分子数: 1 / Fragment: Arginine-rich domain / 由来タイプ: 合成 詳細: This sequence occurs naturally in Jembrana disease virus (JDV). 参照: UniProt: Q82854*PLUS |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||||||||||||||||||
| NMR実験の詳細 | Text: This structure was determined using standard 2D homonuclear techniques. |
- 試料調製
試料調製
| 詳細 |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料状態 |
|
-NMR測定
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M |
|---|---|
| 放射波長 | 相対比: 1 |
| NMRスペクトロメーター | タイプ: Varian UNITY / 製造業者: Varian / モデル: UNITY / 磁場強度: 600 MHz |
- 解析
解析
| NMR software |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: distance geometry, restrained molecular dynamics / ソフトェア番号: 1 詳細: Structures were calculated beginning with 1,000 random structures and incrementally adding distance constraints in four iterations. The final 30 structures with the lowest number of ...詳細: Structures were calculated beginning with 1,000 random structures and incrementally adding distance constraints in four iterations. The final 30 structures with the lowest number of violations were subjected to RMD. The structures are based on a total of 700 distance constraints, 61 dihedral angle restraints. | ||||||||||||||||||||
| 代表構造 | 選択基準: lowest energy | ||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the lowest energy 計算したコンフォーマーの数: 30 / 登録したコンフォーマーの数: 10 |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj





























 CYANA
CYANA