+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1xvn | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | echinomycin (ACGTACGT)2 complex | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | DNA/ANTIBIOTIC / BISINTERCALATOR / HOOGSTEEN BASEPAIR / DEPSIPEPTIDE / QUINOXALINE / THIOACETAL / ANTIBIOTIC / ANTITUMOR / DNA-ANTIBIOTIC COMPLEX | ||||||
| 機能・相同性 | Echinomycin / 2-CARBOXYQUINOXALINE / : / DNA 機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  STREPTOMYCES ECHINATUS (バクテリア) STREPTOMYCES ECHINATUS (バクテリア) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.5 Å 分子置換 / 解像度: 1.5 Å | ||||||
 データ登録者 データ登録者 | Cuesta-Seijo, J.A. / Sheldrick, G.M. | ||||||
 引用 引用 |  ジャーナル: Acta Crystallogr.,Sect.D / 年: 2005 ジャーナル: Acta Crystallogr.,Sect.D / 年: 2005タイトル: Structures of Complexes between Echinomycin and Duplex DNA. 著者: Cuesta-Seijo, J.A. / Sheldrick, G.M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1xvn.cif.gz 1xvn.cif.gz | 27.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1xvn.ent.gz pdb1xvn.ent.gz | 20.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1xvn.json.gz 1xvn.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1xvn_validation.pdf.gz 1xvn_validation.pdf.gz | 418 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1xvn_full_validation.pdf.gz 1xvn_full_validation.pdf.gz | 419.3 KB | 表示 | |
| XML形式データ |  1xvn_validation.xml.gz 1xvn_validation.xml.gz | 4.5 KB | 表示 | |
| CIF形式データ |  1xvn_validation.cif.gz 1xvn_validation.cif.gz | 5.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/xv/1xvn https://data.pdbj.org/pub/pdb/validation_reports/xv/1xvn ftp://data.pdbj.org/pub/pdb/validation_reports/xv/1xvn ftp://data.pdbj.org/pub/pdb/validation_reports/xv/1xvn | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 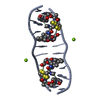
| ||||||||||||
| 単位格子 |
| ||||||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 2426.617 Da / 分子数: 1 / 由来タイプ: 合成 | ||||
|---|---|---|---|---|---|
| #2: タンパク質・ペプチド |   タイプ: Cyclic depsipeptide / クラス: 抗生剤 / 分子量: 809.008 Da / 分子数: 1 / 由来タイプ: 天然 タイプ: Cyclic depsipeptide / クラス: 抗生剤 / 分子量: 809.008 Da / 分子数: 1 / 由来タイプ: 天然詳細: ECHINOMYCIN IS A BICYCLIC OCTADEPSIPEPTIDE. BICYCLIZATION IS ACHIEVED BY LINKING THE N- AND THE C- TERMINI, AND A THIOACETAL BOND BETWEEN RESIDUES 3 AND 7. THE TWO QUINOXALINE CHROMOPHORES ...詳細: ECHINOMYCIN IS A BICYCLIC OCTADEPSIPEPTIDE. BICYCLIZATION IS ACHIEVED BY LINKING THE N- AND THE C- TERMINI, AND A THIOACETAL BOND BETWEEN RESIDUES 3 AND 7. THE TWO QUINOXALINE CHROMOPHORES ARE LINKED TO THE D-SERINE RESIDUES, RESIDUES 1 AND 5. 由来: (天然)  STREPTOMYCES ECHINATUS (バクテリア) / 参照: NOR: NOR01126, Echinomycin STREPTOMYCES ECHINATUS (バクテリア) / 参照: NOR: NOR01126, Echinomycin | ||||
| #3: 化合物 | ChemComp-MG / | ||||
| #4: 化合物 | 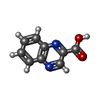  タイプ: Cyclic depsipeptide / クラス: 抗生剤 / 分子量: 174.156 Da / 分子数: 2 / 由来タイプ: 合成 / 式: C9H6N2O2 タイプ: Cyclic depsipeptide / クラス: 抗生剤 / 分子量: 174.156 Da / 分子数: 2 / 由来タイプ: 合成 / 式: C9H6N2O2詳細: ECHINOMYCIN IS A BICYCLIC OCTADEPSIPEPTIDE. BICYCLIZATION IS ACHIEVED BY LINKING THE N- AND THE C- TERMINI, AND A THIOACETAL BOND BETWEEN RESIDUES 3 AND 7. THE TWO QUINOXALINE CHROMOPHORES ...詳細: ECHINOMYCIN IS A BICYCLIC OCTADEPSIPEPTIDE. BICYCLIZATION IS ACHIEVED BY LINKING THE N- AND THE C- TERMINI, AND A THIOACETAL BOND BETWEEN RESIDUES 3 AND 7. THE TWO QUINOXALINE CHROMOPHORES ARE LINKED TO THE D-SERINE RESIDUES, RESIDUES 1 AND 5. 参照: Echinomycin #5: 水 | ChemComp-HOH / | 構成要素の詳細 | THE ECHINOMYCIN IS A BICYCLIC OCTADEPSIPEPTIDE, A MEMBER OF THE QUINOXALINE CLASS OF ANTIBIOTICS. ...THE ECHINOMYCI | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.19 Å3/Da / 溶媒含有率: 43.32 % | ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | pH: 6 詳細: PEG 200, PEG 3350, MGCL2, MES, SPERMINE-HCL, PH 6, VAPOR DIFFUSION, HANGING DROP, TEMPERATURE 293K | ||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 |
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  BESSY BESSY  / ビームライン: 14.1 / 波長: 0.9 / ビームライン: 14.1 / 波長: 0.9 |
| 検出器 | タイプ: MAR CCD 165 mm / 検出器: CCD / 日付: 2003年12月11日 |
| 放射 | モノクロメーター: SI-111 CRYSTAL / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.9 Å / 相対比: 1 |
| 反射 | 解像度: 1.5→26.2 Å / Num. obs: 6499 / % possible obs: 99 % / Observed criterion σ(I): 0 / 冗長度: 21.27 % / Rmerge(I) obs: 0.0424 / Rsym value: 0.0424 / Net I/σ(I): 39.11 |
| 反射 シェル | 解像度: 1.5→1.6 Å / 冗長度: 20.44 % / Rmerge(I) obs: 0.4225 / Mean I/σ(I) obs: 9.03 / Rsym value: 0.4225 / % possible all: 98.4 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 / 解像度: 1.5→26.2 Å / 交差検証法: FREE R / σ(F): 0 / 立体化学のターゲット値: ENGH AND HUBER 分子置換 / 解像度: 1.5→26.2 Å / 交差検証法: FREE R / σ(F): 0 / 立体化学のターゲット値: ENGH AND HUBER詳細: THE SKELETON OF ECHINOMYCIN IS NEARLY SYMMETRIC (NOT MODELLED TO DO SO), THE BRIDGE IS NOT. BOTH POSSIBLE ORIENTATIONS OF BINDING WERE OBSERVED AND ARE MODELLED AS DISORDERED/MICROHETE.
| ||||||||||||||||||||
| Refine analyze | Num. disordered residues: 7 | ||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.5→26.2 Å
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj


