+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1xst | ||||||
|---|---|---|---|---|---|---|---|
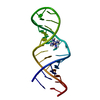
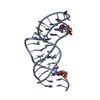
| タイトル | Solution structure of E.coli RNase P RNA P4 stem, U69A mutation, complexed with cobalt (III) hexammine. | ||||||
 要素 要素 | RNA (27-MER) | ||||||
 キーワード キーワード | RNA / Ribonuclease P RNA / Ribozyme / transfer RNA processing / P4 stem / U69A mutant / metal binding site / metal complex / cobalt (III) hexammine complex | ||||||
| 機能・相同性 | COBALT HEXAMMINE(III) / RNA / RNA (> 10) 機能・相同性情報 機能・相同性情報 | ||||||
| 手法 | 溶液NMR / restrained molecular dynamics, simulated annealing | ||||||
| Model type details | minimized average | ||||||
 データ登録者 データ登録者 | Schmitz, M. | ||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res. / 年: 2004 ジャーナル: Nucleic Acids Res. / 年: 2004タイトル: Change of RNase P RNA function by single base mutation correlates with perturbation of metal ion binding in P4 as determined by NMR spectroscopy 著者: Schmitz, M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1xst.cif.gz 1xst.cif.gz | 26.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1xst.ent.gz pdb1xst.ent.gz | 17.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1xst.json.gz 1xst.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1xst_validation.pdf.gz 1xst_validation.pdf.gz | 328 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1xst_full_validation.pdf.gz 1xst_full_validation.pdf.gz | 328 KB | 表示 | |
| XML形式データ |  1xst_validation.xml.gz 1xst_validation.xml.gz | 1.8 KB | 表示 | |
| CIF形式データ |  1xst_validation.cif.gz 1xst_validation.cif.gz | 2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/xs/1xst https://data.pdbj.org/pub/pdb/validation_reports/xs/1xst ftp://data.pdbj.org/pub/pdb/validation_reports/xs/1xst ftp://data.pdbj.org/pub/pdb/validation_reports/xs/1xst | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 8656.183 Da / 分子数: 1 / 変異: U69A / 由来タイプ: 合成 詳細: enzymatically synthesized from DNA oligonucleotide template ny T7 RNA polymerase |
|---|---|
| #2: 化合物 | ChemComp-NCO / |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||||||||||||||
| NMR実験の詳細 | Text: The structure was determined using standard 2D homonuclear techniques as well as 13C and 31P heteronuclear experiments performed at natural abundance. Intermolecular NOE crosspeaks between RNA ...Text: The structure was determined using standard 2D homonuclear techniques as well as 13C and 31P heteronuclear experiments performed at natural abundance. Intermolecular NOE crosspeaks between RNA protons and cobalt (III) hexammine protons and intermolecular distance constraints derived thereof were used to determine the site of cobalt (III) hexammine binding. |
- 試料調製
試料調製
| 詳細 |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料状態 |
|
-NMR測定
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M |
|---|---|
| 放射波長 | 相対比: 1 |
| NMRスペクトロメーター | タイプ: Bruker DMX / 製造業者: Bruker / モデル: DMX / 磁場強度: 600 MHz |
- 解析
解析
| NMR software |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: restrained molecular dynamics, simulated annealing / ソフトェア番号: 1 詳細: The average structure is based on the superposition of 14 structures after refinement. The average RMS deviation between the ensemble and the average structure is 1.85 Angstrom. A total of ...詳細: The average structure is based on the superposition of 14 structures after refinement. The average RMS deviation between the ensemble and the average structure is 1.85 Angstrom. A total of 290 NOE-derived distance constraints, 245 dihedral constraints and 48 distance constraints from hydrogen bonds were used in refinement. 15 NOE derived intermolecular distance constraints were used to localize the bound cobalt (III) hexammine. | ||||||||||||||||||||
| 代表構造 | 選択基準: minimized average structure | ||||||||||||||||||||
| NMRアンサンブル | 登録したコンフォーマーの数: 1 |
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj






























 NMRPipe
NMRPipe