+ データを開く
データを開く
- 基本情報
基本情報
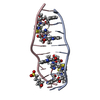
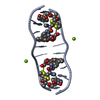
| 登録情報 | データベース: PDB / ID: 1vs2 | ||||||
|---|---|---|---|---|---|---|---|
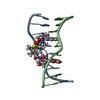
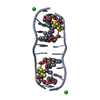
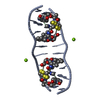
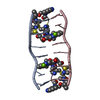
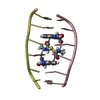
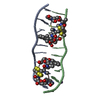
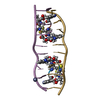
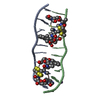
| タイトル | Interactions of quinoxaline antibiotic and DNA: the molecular structure of a TRIOSTIN A-D(GCGTACGC) complex | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | DNA/ANTIBIOTIC / BISINTERCALATOR / DESIPEPTIDE / QUINOXALINE / ANTIBIOTIC / ANTITUMOR / DNA-ANTIBIOTIC COMPLEX | ||||||
| 機能・相同性 | TRIOSTIN A / 2-CARBOXYQUINOXALINE / : / DNA 機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  STREPTOMYCINEAE (バクテリア) STREPTOMYCINEAE (バクテリア) | ||||||
| 手法 |  X線回折 / 解像度: 2 Å X線回折 / 解像度: 2 Å | ||||||
 データ登録者 データ登録者 | Wang, A.H.-J. / Ughetto, G. / Quigley, G.J. / Rich, A. | ||||||
 引用 引用 |  ジャーナル: J.Biomol.Struct.Dyn. / 年: 1986 ジャーナル: J.Biomol.Struct.Dyn. / 年: 1986タイトル: Interactions of Quinoxaline Antibiotic and DNA: The Molecular Structure of a Triostin A-D(Gcgtacgc) Complex. 著者: Wang, A.H. / Ughetto, G. / Quigley, G.J. / Rich, A. #1:  ジャーナル: Science / 年: 1986 ジャーナル: Science / 年: 1986タイトル: Non-Watson-Crick G-C and A-T Base Pairs in a DNA-Antibiotic Complex 著者: Quigley, G.J. / Ughetto, G. / Van Der Marel, G.A. / Van Boom, J.H. / Wang, A.H.-J. / Rich, A. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1vs2.cif.gz 1vs2.cif.gz | 18.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1vs2.ent.gz pdb1vs2.ent.gz | 10.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1vs2.json.gz 1vs2.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1vs2_validation.pdf.gz 1vs2_validation.pdf.gz | 366.2 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1vs2_full_validation.pdf.gz 1vs2_full_validation.pdf.gz | 381.6 KB | 表示 | |
| XML形式データ |  1vs2_validation.xml.gz 1vs2_validation.xml.gz | 4.6 KB | 表示 | |
| CIF形式データ |  1vs2_validation.cif.gz 1vs2_validation.cif.gz | 5.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/vs/1vs2 https://data.pdbj.org/pub/pdb/validation_reports/vs/1vs2 ftp://data.pdbj.org/pub/pdb/validation_reports/vs/1vs2 ftp://data.pdbj.org/pub/pdb/validation_reports/vs/1vs2 | HTTPS FTP |
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 2427.605 Da / 分子数: 1 / 由来タイプ: 合成 | ||
|---|---|---|---|
| #2: タンパク質・ペプチド | 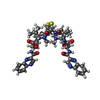  タイプ: Cyclic depsipeptide / クラス: 抗がん剤 / 分子量: 794.982 Da / 分子数: 1 / 由来タイプ: 天然 タイプ: Cyclic depsipeptide / クラス: 抗がん剤 / 分子量: 794.982 Da / 分子数: 1 / 由来タイプ: 天然詳細: TRIOSTIN IS A BICYCLIC OCTADEPSIPEPTIDE. BICYCLIZATION IS ACHIEVED BY LINKING THE N- AND THE C- TERMINI, AND A DISULPHIDE BOND BETWEEN RESIDUES 3 AND 7. THE TWO QUINOXALINE CHROMOPHORES ARE ...詳細: TRIOSTIN IS A BICYCLIC OCTADEPSIPEPTIDE. BICYCLIZATION IS ACHIEVED BY LINKING THE N- AND THE C- TERMINI, AND A DISULPHIDE BOND BETWEEN RESIDUES 3 AND 7. THE TWO QUINOXALINE CHROMOPHORES ARE LINKED TO THE D-SERINE RESIDUES, RESIDUES 1 AND 5. 由来: (天然)  STREPTOMYCINEAE (バクテリア) / 参照: NOR: NOR01129, TRIOSTIN A STREPTOMYCINEAE (バクテリア) / 参照: NOR: NOR01129, TRIOSTIN A | ||
| #3: 化合物 | 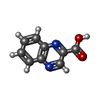  タイプ: Cyclic depsipeptide / クラス: 抗がん剤 / 分子量: 174.156 Da / 分子数: 2 / 由来タイプ: 合成 / 式: C9H6N2O2 タイプ: Cyclic depsipeptide / クラス: 抗がん剤 / 分子量: 174.156 Da / 分子数: 2 / 由来タイプ: 合成 / 式: C9H6N2O2詳細: TRIOSTIN IS A BICYCLIC OCTADEPSIPEPTIDE. BICYCLIZATION IS ACHIEVED BY LINKING THE N- AND THE C- TERMINI, AND A DISULPHIDE BOND BETWEEN RESIDUES 3 AND 7. THE TWO QUINOXALINE CHROMOPHORES ARE ...詳細: TRIOSTIN IS A BICYCLIC OCTADEPSIPEPTIDE. BICYCLIZATION IS ACHIEVED BY LINKING THE N- AND THE C- TERMINI, AND A DISULPHIDE BOND BETWEEN RESIDUES 3 AND 7. THE TWO QUINOXALINE CHROMOPHORES ARE LINKED TO THE D-SERINE RESIDUES, RESIDUES 1 AND 5. 参照: TRIOSTIN A 構成要素の詳細 | TRIOSTIN IS A BICYCLIC OCTADEPSIPEPTIDE, A MEMBER OF THE QUINOXALINE CLASS OF ANTIBIOTICS. HERE, ...TRIOSTIN IS A BICYCLIC OCTADEPSIP | |
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.84 Å3/Da / 溶媒含有率: 67.94 % | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | pH: 4.5 / 詳細: PH 4.50, VAPOR DIFFUSION | ||||||||||||||||||||||||||||||||||||
| 溶液の組成 |
|
-データ収集
| 回折 | 平均測定温度: 285 K |
|---|---|
| 検出器 | タイプ: NICOLET P3 / 検出器: DIFFRACTOMETER |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 相対比: 1 |
| 反射 | 最高解像度: 2 Å / Num. obs: 1130 / Observed criterion σ(I): 1.5 |
- 解析
解析
| ソフトウェア | 名称: NUCLSQ / 分類: 精密化 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 2→10 Å /
| ||||||||||||
| Refine Biso | クラス: ALL ATOMS / 詳細: TR / Treatment: isotropic | ||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2→10 Å
|
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj