+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1tze | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | SIGNAL TRANSDUCTION ADAPTOR GROWTH FACTOR, GRB2 SH2 DOMAIN COMPLEXED WITH PHOSPHOTYROSYL HEPTAPEPTIDE LYS-PRO-PHE-PTYR-VAL-ASN-VAL-NH2 (KFPPYVNC-NH2) | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | COMPLEX (SIGNAL TRANSDUCTION/PEPTIDE) / SIGNAL TRANSDUCTION ADAPTOR GROWTH FACTOR BOUND PROTEIN 2 (GRB2) SRC HOMOLOGY 2 DOMAIN (SH2) / PHOSPHOTYROSYL HEPTAPEPTIC LIGAND / COMPLEX (SIGNAL TRANSDUCTION-PEPTIDE) / COMPLEX (SIGNAL TRANSDUCTION-PEPTIDE) complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / : / Regulation of T cell activation by CD28 family / : / Signaling by FGFR3 fusions in cancer / anatomical structure formation involved in morphogenesis / guanyl-nucleotide exchange factor adaptor activity / Grb2-EGFR complex / branching involved in labyrinthine layer morphogenesis / STAT5 Activation ...: / : / Regulation of T cell activation by CD28 family / : / Signaling by FGFR3 fusions in cancer / anatomical structure formation involved in morphogenesis / guanyl-nucleotide exchange factor adaptor activity / Grb2-EGFR complex / branching involved in labyrinthine layer morphogenesis / STAT5 Activation / Co-inhibition by BTLA / COP9 signalosome / neurotrophin TRKA receptor binding / Activated NTRK2 signals through PI3K / transmembrane receptor protein tyrosine kinase adaptor activity / MET receptor recycling / Signaling by cytosolic FGFR1 fusion mutants / Interleukin-15 signaling / MET activates PTPN11 / negative regulation of natural killer cell mediated cytotoxicity / MET activates RAP1 and RAC1 / vesicle membrane / Signaling by LTK / MET activates PI3K/AKT signaling / CD28 dependent Vav1 pathway / Signal regulatory protein family interactions / epidermal growth factor receptor binding / Regulation of KIT signaling / PI-3K cascade:FGFR3 / natural killer cell mediated cytotoxicity / STAT5 activation downstream of FLT3 ITD mutants / PI-3K cascade:FGFR2 / PI-3K cascade:FGFR4 / PI-3K cascade:FGFR1 / positive regulation of actin filament polymerization / endodermal cell differentiation / GRB2:SOS provides linkage to MAPK signaling for Integrins / RHOU GTPase cycle / regulation of MAPK cascade / RET signaling / PI3K events in ERBB2 signaling / insulin receptor substrate binding / Interleukin-3, Interleukin-5 and GM-CSF signaling / PI3K Cascade / SOS-mediated signalling / Activated NTRK3 signals through RAS / Activated NTRK2 signals through RAS / signal transduction in response to DNA damage / fibroblast growth factor receptor signaling pathway / SHC1 events in ERBB4 signaling / RHO GTPases Activate WASPs and WAVEs / Role of LAT2/NTAL/LAB on calcium mobilization / Interleukin receptor SHC signaling / Signalling to RAS / GAB1 signalosome / Signal attenuation / Activated NTRK2 signals through FRS2 and FRS3 / SHC-related events triggered by IGF1R / Schwann cell development / SHC-mediated cascade:FGFR3 / MET activates RAS signaling / SHC-mediated cascade:FGFR2 / Signaling by PDGFRA transmembrane, juxtamembrane and kinase domain mutants / Signaling by PDGFRA extracellular domain mutants / SHC-mediated cascade:FGFR4 / Erythropoietin activates RAS / Signaling by FGFR4 in disease / SHC-mediated cascade:FGFR1 / FRS-mediated FGFR3 signaling / Signaling by CSF3 (G-CSF) / Signaling by FLT3 ITD and TKD mutants / ephrin receptor binding / FRS-mediated FGFR2 signaling / FRS-mediated FGFR4 signaling / InlB-mediated entry of Listeria monocytogenes into host cell / Signaling by FGFR3 in disease / FRS-mediated FGFR1 signaling / Tie2 Signaling / Signaling by FGFR2 in disease / phosphotyrosine residue binding / GRB2 events in EGFR signaling / SHC1 events in EGFR signaling / myelination / Signaling by FLT3 fusion proteins / FLT3 Signaling / EGFR Transactivation by Gastrin / Signaling by FGFR1 in disease / FCERI mediated Ca+2 mobilization / NCAM signaling for neurite out-growth / GRB2 events in ERBB2 signaling / cellular response to ionizing radiation / Downstream signal transduction / SHC1 events in ERBB2 signaling / Insulin receptor signalling cascade / insulin-like growth factor receptor signaling pathway / Regulation of signaling by CBL / Negative regulation of FGFR3 signaling / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / Constitutive Signaling by Overexpressed ERBB2 / Signaling by phosphorylated juxtamembrane, extracellular and kinase domain KIT mutants 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / 解像度: 2.1 Å X線回折 / 解像度: 2.1 Å | ||||||
 データ登録者 データ登録者 | Rahuel, J. / Grutter, M.G. | ||||||
 引用 引用 |  ジャーナル: Nat.Struct.Biol. / 年: 1996 ジャーナル: Nat.Struct.Biol. / 年: 1996タイトル: Structural basis for specificity of Grb2-SH2 revealed by a novel ligand binding mode. 著者: Rahuel, J. / Gay, B. / Erdmann, D. / Strauss, A. / Garcia-Echeverria, C. / Furet, P. / Caravatti, G. / Fretz, H. / Schoepfer, J. / Grutter, M.G. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1tze.cif.gz 1tze.cif.gz | 36.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1tze.ent.gz pdb1tze.ent.gz | 24.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1tze.json.gz 1tze.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1tze_validation.pdf.gz 1tze_validation.pdf.gz | 422.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1tze_full_validation.pdf.gz 1tze_full_validation.pdf.gz | 423.1 KB | 表示 | |
| XML形式データ |  1tze_validation.xml.gz 1tze_validation.xml.gz | 7.7 KB | 表示 | |
| CIF形式データ |  1tze_validation.cif.gz 1tze_validation.cif.gz | 10.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/tz/1tze https://data.pdbj.org/pub/pdb/validation_reports/tz/1tze ftp://data.pdbj.org/pub/pdb/validation_reports/tz/1tze ftp://data.pdbj.org/pub/pdb/validation_reports/tz/1tze | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 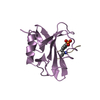
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 5 | 
| ||||||||
| 単位格子 |
| ||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: タンパク質 | 分子量: 11447.027 Da / 分子数: 1 / 断片: SH2 DOMAIN RESIDUES 55 - 152 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 細胞株: BL21 / 遺伝子: GRB2 55-152 / プラスミド: PET-3C / 生物種 (発現宿主): Escherichia coli / 遺伝子 (発現宿主): GRB2 55-152 / 発現宿主: Homo sapiens (ヒト) / 細胞株: BL21 / 遺伝子: GRB2 55-152 / プラスミド: PET-3C / 生物種 (発現宿主): Escherichia coli / 遺伝子 (発現宿主): GRB2 55-152 / 発現宿主:  |
|---|---|
| #2: タンパク質・ペプチド | 分子量: 945.009 Da / 分子数: 1 / 由来タイプ: 組換発現 |
| #3: 水 | ChemComp-HOH / |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.29 Å3/Da / 溶媒含有率: 46.37 % | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | *PLUS pH: 6.5 / 手法: 蒸気拡散法 | ||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 放射光源 | 波長: 1.5418 |
|---|---|
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1995年10月3日 |
| 放射 | 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | Num. obs: 7237 / % possible obs: 100 % / Observed criterion σ(I): 0 / 冗長度: 7.3 % / Rmerge(I) obs: 0.05 |
| 反射 | *PLUS 最高解像度: 2.1 Å / Num. measured all: 45126 / Rmerge(I) obs: 0.05 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 2.1→10 Å / σ(F): 0 /
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 26.8 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.34 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.1→10 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / バージョン: 3.1 / 分類: refinement X-PLOR / バージョン: 3.1 / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj
























