| 登録情報 | データベース: PDB / ID: 1rlw
|
|---|
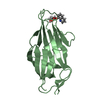
| タイトル | CALCIUM-PHOSPHOLIPID BINDING DOMAIN FROM CYTOSOLIC PHOSPHOLIPASE A2 |
|---|
 要素 要素 | PHOSPHOLIPASE A2 |
|---|
 キーワード キーワード | HYDROLASE / C2 DOMAIN / CALB DOMAIN |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Arachidonate metabolism / phosphatidylglycerol catabolic process / icosanoid metabolic process / platelet activating factor biosynthetic process / Acyl chain remodeling of CL / monoacylglycerol biosynthetic process / Hydrolysis of LPC / glycerophospholipid catabolic process / lysophospholipase / O-acyltransferase activity ...Arachidonate metabolism / phosphatidylglycerol catabolic process / icosanoid metabolic process / platelet activating factor biosynthetic process / Acyl chain remodeling of CL / monoacylglycerol biosynthetic process / Hydrolysis of LPC / glycerophospholipid catabolic process / lysophospholipase / O-acyltransferase activity / phosphatidylcholine acyl-chain remodeling / Acyl chain remodelling of PG / Acyl chain remodelling of PC / calcium-independent phospholipase A2 activity / Acyl chain remodelling of PI / Acyl chain remodelling of PS / Acyl chain remodelling of PE / ceramide 1-phosphate binding / Synthesis of PA / glycerol metabolic process / arachidonate metabolic process / phosphatidylinositol-5-phosphate binding / phospholipase A2 activity / positive regulation of T-helper 1 type immune response / positive regulation of prostaglandin secretion / phospho-PLA2 pathway / calcium-dependent phospholipid binding / leukotriene biosynthetic process / phosphatidylinositol-3-phosphate binding / positive regulation of macrophage activation / positive regulation of platelet activation / phospholipase A2 / phosphatidylinositol-4-phosphate binding / phosphatidylcholine catabolic process / calcium-dependent phospholipase A2 activity / COPI-independent Golgi-to-ER retrograde traffic / prostaglandin biosynthetic process / phosphatidylcholine lysophospholipase activity / arachidonate secretion / cellular response to antibiotic / Platelet sensitization by LDL / establishment of localization in cell / ADP signalling through P2Y purinoceptor 1 / nuclear envelope / regulation of cell population proliferation / mitochondrial inner membrane / Golgi membrane / intracellular membrane-bounded organelle / calcium ion binding / endoplasmic reticulum membrane / endoplasmic reticulum / Golgi apparatus / nucleus / cytosol / cytoplasm類似検索 - 分子機能 Lysophospholipase, catalytic domain / Cytosolic phospholipase A2, C2 domain / Lysophospholipase catalytic domain / PLA2c domain profile. / Cytoplasmic phospholipase A2, catalytic subunit / C2 domain / Protein kinase C conserved region 2 (CalB) / C2 domain / Acyl transferase/acyl hydrolase/lysophospholipase / C2 domain ...Lysophospholipase, catalytic domain / Cytosolic phospholipase A2, C2 domain / Lysophospholipase catalytic domain / PLA2c domain profile. / Cytoplasmic phospholipase A2, catalytic subunit / C2 domain / Protein kinase C conserved region 2 (CalB) / C2 domain / Acyl transferase/acyl hydrolase/lysophospholipase / C2 domain / C2 domain profile. / C2 domain superfamily / Immunoglobulin-like / Sandwich / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多重同系置換 / 解像度: 2.4 Å 多重同系置換 / 解像度: 2.4 Å |
|---|
 データ登録者 データ登録者 | Perisic, O. / Williams, R.L. |
|---|
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 1998 ジャーナル: J.Biol.Chem. / 年: 1998
タイトル: Crystal structure of a calcium-phospholipid binding domain from cytosolic phospholipase A2.
著者: Perisic, O. / Fong, S. / Lynch, D.E. / Bycroft, M. / Williams, R.L. |
|---|
| 履歴 | | 登録 | 1997年10月28日 | 処理サイト: BNL |
|---|
| 改定 1.0 | 1998年2月25日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年3月24日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2021年11月3日 | Group: Data collection / Database references / Derived calculations
カテゴリ: database_2 / diffrn_source ...database_2 / diffrn_source / pdbx_struct_conn_angle / struct_conn / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _diffrn_source.pdbx_synchrotron_site / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr1_symmetry / _pdbx_struct_conn_angle.ptnr2_auth_comp_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr2_label_atom_id / _pdbx_struct_conn_angle.ptnr2_label_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.ptnr3_symmetry / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr1_symmetry / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_conn.ptnr2_symmetry / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
| 改定 1.4 | 2024年2月14日 | Group: Data collection / カテゴリ: chem_comp_atom / chem_comp_bond |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  多重同系置換 / 解像度: 2.4 Å
多重同系置換 / 解像度: 2.4 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Biol.Chem. / 年: 1998
ジャーナル: J.Biol.Chem. / 年: 1998 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1rlw.cif.gz
1rlw.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1rlw.ent.gz
pdb1rlw.ent.gz PDB形式
PDB形式 1rlw.json.gz
1rlw.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1rlw_validation.pdf.gz
1rlw_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1rlw_full_validation.pdf.gz
1rlw_full_validation.pdf.gz 1rlw_validation.xml.gz
1rlw_validation.xml.gz 1rlw_validation.cif.gz
1rlw_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/rl/1rlw
https://data.pdbj.org/pub/pdb/validation_reports/rl/1rlw ftp://data.pdbj.org/pub/pdb/validation_reports/rl/1rlw
ftp://data.pdbj.org/pub/pdb/validation_reports/rl/1rlw リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 細胞内の位置: CYTOSOL / プラスミド: MINI-PRSETA / 発現宿主:
Homo sapiens (ヒト) / 細胞内の位置: CYTOSOL / プラスミド: MINI-PRSETA / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  EMBL/DESY, HAMBURG
EMBL/DESY, HAMBURG  / ビームライン: BW7B / 波長: 0.8838
/ ビームライン: BW7B / 波長: 0.8838  解析
解析 多重同系置換 / 解像度: 2.4→15 Å / 交差検証法: THROUGHOUT / σ(F): 0
多重同系置換 / 解像度: 2.4→15 Å / 交差検証法: THROUGHOUT / σ(F): 0  ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj










