+ データを開く
データを開く
- 基本情報
基本情報
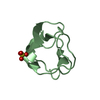
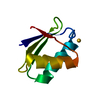
| 登録情報 | データベース: PDB / ID: 1r63 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | STRUCTURAL ROLE OF A BURIED SALT BRIDGE IN THE 434 REPRESSOR DNA-BINDING DOMAIN, NMR, 20 STRUCTURES | ||||||
 要素 要素 | REPRESSOR PROTEIN FROM BACTERIOPHAGE 434 | ||||||
 キーワード キーワード | GENE REGULATING PROTEIN / PHAGE 434 REPRESSOR / HELIX-TURN-HELIX / DNA-BINDING DOMAIN | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  Phage 434 (ファージ) Phage 434 (ファージ) | ||||||
| 手法 | 溶液NMR / DISTANCE GEOMETRY WITH DIANA, ENERGY MINIMIZATION WITH OPAL | ||||||
 データ登録者 データ登録者 | Pervushin, K.V. / Billeter, M. / Siegal, G. / Wuthrich, K. | ||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 1996 ジャーナル: J.Mol.Biol. / 年: 1996タイトル: Structural role of a buried salt bridge in the 434 repressor DNA-binding domain. 著者: Pervushin, K. / Billeter, M. / Siegal, G. / Wuthrich, K. #1:  ジャーナル: J.Mol.Biol. / 年: 1992 ジャーナル: J.Mol.Biol. / 年: 1992タイトル: Determination of the Nuclear Magnetic Resonance Solution Structure of the DNA-Binding Domain (Residues 1 to 69) of the 434 Repressor and Comparison with the X-Ray Crystal Structure 著者: Neri, D. / Billeter, M. / Wuthrich, K. #2:  ジャーナル: J.Mol.Biol. / 年: 1989 ジャーナル: J.Mol.Biol. / 年: 1989タイトル: Structure of the Amino-Terminal Domain of Phage 434 Repressor at 2.0 A Resolution 著者: Mondragon, A. / Subbiah, S. / Almo, S.C. / Drottar, M. / Harrison, S.C. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1r63.cif.gz 1r63.cif.gz | 427.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1r63.ent.gz pdb1r63.ent.gz | 364.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1r63.json.gz 1r63.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1r63_validation.pdf.gz 1r63_validation.pdf.gz | 356.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1r63_full_validation.pdf.gz 1r63_full_validation.pdf.gz | 463.3 KB | 表示 | |
| XML形式データ |  1r63_validation.xml.gz 1r63_validation.xml.gz | 18.1 KB | 表示 | |
| CIF形式データ |  1r63_validation.cif.gz 1r63_validation.cif.gz | 32.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/r6/1r63 https://data.pdbj.org/pub/pdb/validation_reports/r6/1r63 ftp://data.pdbj.org/pub/pdb/validation_reports/r6/1r63 ftp://data.pdbj.org/pub/pdb/validation_reports/r6/1r63 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 6900.871 Da / 分子数: 1 / 断片: DNA-BINDING DOMAIN, RESIDUES 1 - 63 / 由来タイプ: 組換発現 / 由来: (組換発現)  Phage 434 (ファージ) / 属: Lambda-like viruses / 生物種: Enterobacteria phage lambda / 発現宿主: Phage 434 (ファージ) / 属: Lambda-like viruses / 生物種: Enterobacteria phage lambda / 発現宿主:  |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 試料状態 | pH: 4.8 / 温度: 286 K |
|---|---|
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター | タイプ: Bruker AMX600 / 製造業者: Bruker / モデル: AMX600 / 磁場強度: 600 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: DISTANCE GEOMETRY WITH DIANA, ENERGY MINIMIZATION WITH OPAL ソフトェア番号: 1 | ||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: DIANA PENALTY FUNCTION 計算したコンフォーマーの数: 20 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj



