| 登録情報 | データベース: PDB / ID: 1nu2
|
|---|
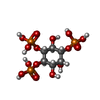
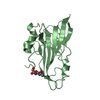
| タイトル | Crystal structure of the murine Disabled-1 (Dab1) PTB domain-ApoER2 peptide-PI-4,5P2 ternary complex |
|---|
 要素 要素 | - Disabled homolog 1
- peptide derived from murine Apolipoprotein E Receptor-2
|
|---|
 キーワード キーワード | SIGNALING PROTEIN / beta-sandwich |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
cell-cell adhesion involved in neuronal-glial interactions involved in cerebral cortex radial glia guided migration / lateral motor column neuron migration / Reelin signalling pathway / radial glia guided migration of Purkinje cell / cerebral cortex radially oriented cell migration / Golgi localization / cerebellum structural organization / radial glia-guided pyramidal neuron migration / ventral spinal cord development / negative regulation of axonogenesis ...cell-cell adhesion involved in neuronal-glial interactions involved in cerebral cortex radial glia guided migration / lateral motor column neuron migration / Reelin signalling pathway / radial glia guided migration of Purkinje cell / cerebral cortex radially oriented cell migration / Golgi localization / cerebellum structural organization / radial glia-guided pyramidal neuron migration / ventral spinal cord development / negative regulation of axonogenesis / motor neuron migration / reelin-mediated signaling pathway / negative regulation of receptor signaling pathway via JAK-STAT / negative regulation of cell adhesion / regulation of synapse maturation / astrocyte differentiation / adult walking behavior / small GTPase-mediated signal transduction / dendrite development / cerebral cortex cell migration / negative regulation of astrocyte differentiation / cell surface receptor signaling pathway via JAK-STAT / signaling adaptor activity / positive regulation of neuron differentiation / SH2 domain binding / axonogenesis / hippocampus development / central nervous system development / phospholipid binding / neuron differentiation / neuron migration / nervous system development / cell adhesion / intracellular signal transduction / intracellular membrane-bounded organelle / synapse / perinuclear region of cytoplasm / glutamatergic synapse / cytoplasm類似検索 - 分子機能 : / : / Disabled homolog 2-like, sulfatide-binding motifs / Phosphotyrosine interaction domain (PTB/PID) / Phosphotyrosine interaction domain (PID) profile. / Phosphotyrosine-binding domain, phosphotyrosine-interaction (PI) domain / PTB/PI domain / Pleckstrin-homology domain (PH domain)/Phosphotyrosine-binding domain (PTB) / PH-domain like / PH-like domain superfamily ...: / : / Disabled homolog 2-like, sulfatide-binding motifs / Phosphotyrosine interaction domain (PTB/PID) / Phosphotyrosine interaction domain (PID) profile. / Phosphotyrosine-binding domain, phosphotyrosine-interaction (PI) domain / PTB/PI domain / Pleckstrin-homology domain (PH domain)/Phosphotyrosine-binding domain (PTB) / PH-domain like / PH-like domain superfamily / Roll / Mainly Beta類似検索 - ドメイン・相同性 D-MYO-INOSITOL-1,4,5-TRIPHOSPHATE / Disabled homolog 1類似検索 - 構成要素 |
|---|
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
|---|
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 1.9 Å 分子置換 / 解像度: 1.9 Å |
|---|
 データ登録者 データ登録者 | Stolt, P.C. / Jeon, H. / Song, H.K. / Herz, J. / Eck, M.J. / Blacklow, S.C. |
|---|
 引用 引用 |  ジャーナル: Structure / 年: 2003 ジャーナル: Structure / 年: 2003
タイトル: Origins of Peptide Selectivity and Phosphoinositide Binding Revealed by Structures of Disabled-1 PTB Domain Complexes
著者: Stolt, P.C. / Jeon, H. / Song, H.K. / Herz, J. / Eck, M.J. / Blacklow, S.C. |
|---|
| 履歴 | | 登録 | 2003年1月30日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2003年4月15日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月29日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2018年4月4日 | Group: Data collection / カテゴリ: diffrn_source / Item: _diffrn_source.type |
|---|
| 改定 1.4 | 2023年8月16日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
| 改定 1.5 | 2024年3月13日 | Group: Source and taxonomy / Structure summary / カテゴリ: entity / pdbx_entity_src_syn / Item: _entity.details |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  分子置換 / 解像度: 1.9 Å
分子置換 / 解像度: 1.9 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Structure / 年: 2003
ジャーナル: Structure / 年: 2003 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1nu2.cif.gz
1nu2.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1nu2.ent.gz
pdb1nu2.ent.gz PDB形式
PDB形式 1nu2.json.gz
1nu2.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/nu/1nu2
https://data.pdbj.org/pub/pdb/validation_reports/nu/1nu2 ftp://data.pdbj.org/pub/pdb/validation_reports/nu/1nu2
ftp://data.pdbj.org/pub/pdb/validation_reports/nu/1nu2 リンク
リンク 集合体
集合体
 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU RU200 / 波長: 1.5418
回転陽極 / タイプ: RIGAKU RU200 / 波長: 1.5418  解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj