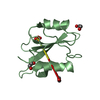
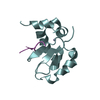
登録情報 データベース : PDB / ID : 1mv0タイトル NMR STRUCTURE OF THE TUMOR SUPPRESSOR BIN1: ALTERNATIVE SPLICING IN MELANOMA AND INTERACTION WITH C-MYC Myc box-dependent-interacting protein 1 Myc proto-oncogene protein キーワード / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / データ登録者 Pineda-Lucena, A. / Arrowsmith, C.H. ジャーナル : J.Mol.Biol. / 年 : 2005タイトル : A structure-based model of the c-Myc/Bin1 protein interaction shows alternative splicing of Bin1 and c-Myc phosphorylation are key binding determinants.著者 : Pineda-Lucena, A. / Ho, C.S. / Mao, D.Y. / Sheng, Y. / Laister, R.C. / Muhandiram, R. / Lu, Y. / Seet, B.T. / Katz, S. / Szyperski, T. / Penn, L.Z. / Arrowsmith, C.H. 履歴 登録 2002年9月24日 登録サイト / 処理サイト 改定 1.0 2003年9月30日 Provider / タイプ 改定 1.1 2008年4月28日 Group 改定 1.2 2011年7月13日 Group 改定 1.3 2022年2月23日 Group / Derived calculationsカテゴリ / pdbx_struct_assembly / pdbx_struct_oper_listItem / _database_2.pdbx_database_accession改定 1.4 2024年5月22日 Group / カテゴリ / chem_comp_bond
すべて表示 表示を減らす Remark 999 SEQUENCE The protein crystallized by the author contains Lys465 which corresponds to Glu576 in the ... SEQUENCE The protein crystallized by the author contains Lys465 which corresponds to Glu576 in the Swiss-Prot entry O00499. This residue conflict is noted in the database reference.
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: J.Mol.Biol. / 年: 2005
ジャーナル: J.Mol.Biol. / 年: 2005 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1mv0.cif.gz
1mv0.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1mv0.ent.gz
pdb1mv0.ent.gz PDB形式
PDB形式 1mv0.json.gz
1mv0.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1mv0_validation.pdf.gz
1mv0_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1mv0_full_validation.pdf.gz
1mv0_full_validation.pdf.gz 1mv0_validation.xml.gz
1mv0_validation.xml.gz 1mv0_validation.cif.gz
1mv0_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/mv/1mv0
https://data.pdbj.org/pub/pdb/validation_reports/mv/1mv0 ftp://data.pdbj.org/pub/pdb/validation_reports/mv/1mv0
ftp://data.pdbj.org/pub/pdb/validation_reports/mv/1mv0 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / プラスミド: pET15b / 発現宿主:
Homo sapiens (ヒト) / プラスミド: pET15b / 発現宿主: 
 Homo sapiens (ヒト) / プラスミド: pET15b / 発現宿主:
Homo sapiens (ヒト) / プラスミド: pET15b / 発現宿主: 
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj










 NMRPipe
NMRPipe