+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1mag | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
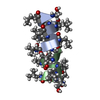
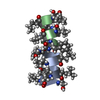
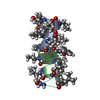
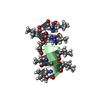
| タイトル | GRAMICIDIN A IN HYDRATED DMPC BILAYERS, SOLID STATE NMR | |||||||||
 要素 要素 | GRAMICIDIN A | |||||||||
 キーワード キーワード | ANTIBIOTIC / GRAMICIDIN / ANTIFUNGAL / ANTIBACTERIAL / MEMBRANE ION CHANNEL / LINEAR GRAMICIDIN / ORIENTED BILAYERS | |||||||||
| 機能・相同性 | GRAMICIDIN A / :  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  BREVIBACILLUS BREVIS (バクテリア) BREVIBACILLUS BREVIS (バクテリア) | |||||||||
| 手法 | 個体NMR / simulated annealing | |||||||||
 データ登録者 データ登録者 | Ketchem, R.R. / Roux, B. / Cross, T.A. | |||||||||
 引用 引用 |  ジャーナル: J.Biomol.NMR / 年: 1996 ジャーナル: J.Biomol.NMR / 年: 1996タイトル: Macromolecular Structural Elucidation with Solid-State NMR-Derived Orientational Constraints. 著者: Ketchem, R.R. / Lee, K.C. / Huo, S. / Cross, T.A. #1:  ジャーナル: Biological Membranes: A Molecular Perspective from Computation and Experiment ジャーナル: Biological Membranes: A Molecular Perspective from Computation and Experiment年: 1996 タイトル: Computational Refinement Through Solid State NMR and Energy Constraints of a Membrane Bound Polypeptide 著者: Ketchem, R.R. / Roux, B. / Cross, T.A. #2: ジャーナル: Biochemistry / 年: 1995 タイトル: Lipid-Peptide Interface: Valine Conformation and Dynamics in the Gramicidin Channel 著者: Lee, K.C. / Huo, S. / Cross, T.A. #3: ジャーナル: Biochemistry / 年: 1995 タイトル: Tryptophan Dynamics and Structural Refinement in a Lipid Bilayer Environment: Solid State NMR of the Gramicidin Channel 著者: Hu, W. / Lazo, N.D. / Cross, T.A. #4: ジャーナル: Science / 年: 1993 タイトル: High-Resolution Conformation of Gramicidin a in a Lipid Bilayer by Solid-State NMR 著者: Ketchem, R.R. / Hu, W. / Cross, T.A. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1mag.cif.gz 1mag.cif.gz | 20 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1mag.ent.gz pdb1mag.ent.gz | 15.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1mag.json.gz 1mag.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1mag_validation.pdf.gz 1mag_validation.pdf.gz | 334.4 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1mag_full_validation.pdf.gz 1mag_full_validation.pdf.gz | 334.2 KB | 表示 | |
| XML形式データ |  1mag_validation.xml.gz 1mag_validation.xml.gz | 2.1 KB | 表示 | |
| CIF形式データ |  1mag_validation.cif.gz 1mag_validation.cif.gz | 2.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ma/1mag https://data.pdbj.org/pub/pdb/validation_reports/ma/1mag ftp://data.pdbj.org/pub/pdb/validation_reports/ma/1mag ftp://data.pdbj.org/pub/pdb/validation_reports/ma/1mag | HTTPS FTP |
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 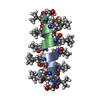
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
-実験情報
-実験
| 実験 | 手法: 個体NMR | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 試料状態 | pH: 7 / 温度: 303 K |
|---|---|
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター | タイプ: CHEMAGNETICS CMX / 製造業者: CHEMAGNETICS / モデル: CMX / 磁場強度: 400 MHz |
|---|
- 解析
解析
| NMR software |
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 詳細: CHARMM23 STRUCTURE REFINED WITH TORC (TOTAL REFINEMENT OF CONSTRAINTS), DEVELOPED IN AUTHORS' LAB. TORC RUNS AS A MODULE WITHIN CHARMM AND UTILIZES A SIMULATED ANNEALING PROTOCOL TO REFINE ...詳細: CHARMM23 STRUCTURE REFINED WITH TORC (TOTAL REFINEMENT OF CONSTRAINTS), DEVELOPED IN AUTHORS' LAB. TORC RUNS AS A MODULE WITHIN CHARMM AND UTILIZES A SIMULATED ANNEALING PROTOCOL TO REFINE THE STRUCTURE AGAINST BOTH THE SOLID STATE NMR DATA (INCLUDING SOLID STATE NMR DERIVED ORIENTATIONAL CONSTRAINTS) AND THE CHARMM ENERGY. | |||||||||
| NMRアンサンブル | 登録したコンフォーマーの数: 1 |
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj

