+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1k42 | ||||||
|---|---|---|---|---|---|---|---|
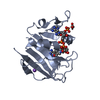
| タイトル | The Solution Structure of the CBM4-2 Carbohydrate Binding Module from a Thermostable Rhodothermus marinus Xylanase. | ||||||
 要素 要素 | Xylanase | ||||||
 キーワード キーワード | HYDROLASE / beta-sandwich formed by 11 strands. Binding-site cleft. Solvent exposed aromatics (Trp69 / Phe110) in binding cleft. Two helical twists. Two calcium binding sites. | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報endo-1,4-beta-xylanase / xylan catabolic process / hydrolase activity, hydrolyzing O-glycosyl compounds 類似検索 - 分子機能 | ||||||
| 生物種 |   Rhodothermus marinus (バクテリア) Rhodothermus marinus (バクテリア) | ||||||
| 手法 | 溶液NMR / hybrid distance geometry, simulated annealing using XPLOR | ||||||
 データ登録者 データ登録者 | Simpson, P.J. / Jamieson, S.J. / Abou-Hachem, M. / Nordberg-Karlsson, E. / Gilbert, H.J. / Holst, O. / Williamson, M.P. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 2002 ジャーナル: Biochemistry / 年: 2002タイトル: The solution structure of the CBM4-2 carbohydrate binding module from a thermostable Rhodothermus marinus xylanase. 著者: Simpson, P.J. / Jamieson, S.J. / Abou-Hachem, M. / Karlsson, E.N. / Gilbert, H.J. / Holst, O. / Williamson, M.P. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1k42.cif.gz 1k42.cif.gz | 582.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1k42.ent.gz pdb1k42.ent.gz | 484.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1k42.json.gz 1k42.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1k42_validation.pdf.gz 1k42_validation.pdf.gz | 346.2 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1k42_full_validation.pdf.gz 1k42_full_validation.pdf.gz | 509.1 KB | 表示 | |
| XML形式データ |  1k42_validation.xml.gz 1k42_validation.xml.gz | 67.4 KB | 表示 | |
| CIF形式データ |  1k42_validation.cif.gz 1k42_validation.cif.gz | 89.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/k4/1k42 https://data.pdbj.org/pub/pdb/validation_reports/k4/1k42 ftp://data.pdbj.org/pub/pdb/validation_reports/k4/1k42 ftp://data.pdbj.org/pub/pdb/validation_reports/k4/1k42 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 18103.803 Da / 分子数: 1 断片: Second family 4 carbohydrate binding module (CBM4-2)(Residues 211-373) 由来タイプ: 組換発現 由来: (組換発現)   Rhodothermus marinus (バクテリア) Rhodothermus marinus (バクテリア)遺伝子: xyn10A / プラスミド: pET-25b(+) / 生物種 (発現宿主): Escherichia coli / 発現宿主:  |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 | 内容: 1.5 mM CBM4-2 15N-labelled, and 13C,15N double labelled protein; 50 mM CaCl2, 50 mM sodium acetate-d3, pH 6.0, 10% D2O, 10 mM sodium azide, 0.1 mM sodium trimethylsilylpropionate (TSP) 溶媒系: 90% H2O/10% D2O |
|---|---|
| 試料状態 | イオン強度: 0.2 M / pH: 6 / 圧: ambient / 温度: 310 K |
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射波長 | 相対比: 1 | |||||||||||||||
| NMRスペクトロメーター |
|
- 解析
解析
| NMR software |
| ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: hybrid distance geometry, simulated annealing using XPLOR ソフトェア番号: 1 詳細: The final set of restraints contained 1654 non-redundant unambiguous NOEs and 17 ambiguous NOEs, 93 dihedral angle restraints, 72 chi1 and 1 chi2 restraint, and 65 pairs of hydrogen bond ...詳細: The final set of restraints contained 1654 non-redundant unambiguous NOEs and 17 ambiguous NOEs, 93 dihedral angle restraints, 72 chi1 and 1 chi2 restraint, and 65 pairs of hydrogen bond restraints, plus 177 backbone dihedral restraints based on 13C shifts from TALOS. | ||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with acceptable covalent geometry,structures with the least restraint violations,structures with the lowest energy 計算したコンフォーマーの数: 50 / 登録したコンフォーマーの数: 12 |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj
 X-PLOR
X-PLOR