+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1i0c | ||||||
|---|---|---|---|---|---|---|---|
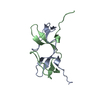
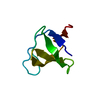
| タイトル | EPS8 SH3 CLOSED MONOMER | ||||||
 要素 要素 | EPIDERMAL GROWTH FACTOR RECEPTOR KINASE SUBSTRATE EPS8 | ||||||
 キーワード キーワード | HORMONE/GROWTH FACTOR / HORMONE-GROWTH FACTOR complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of actin filament length / actin polymerization-dependent cell motility / dendritic cell migration / stereocilium tip / actin crosslink formation / stereocilium bundle / behavioral response to ethanol / barbed-end actin filament capping / exit from mitosis / stereocilium ...regulation of actin filament length / actin polymerization-dependent cell motility / dendritic cell migration / stereocilium tip / actin crosslink formation / stereocilium bundle / behavioral response to ethanol / barbed-end actin filament capping / exit from mitosis / stereocilium / NMDA selective glutamate receptor complex / Rac protein signal transduction / brush border / actin filament bundle assembly / regulation of postsynaptic membrane neurotransmitter receptor levels / cellular response to leukemia inhibitory factor / adult locomotory behavior / small GTPase binding / ruffle membrane / regulation of cell shape / actin binding / growth cone / actin cytoskeleton organization / cell cortex / postsynaptic density / synapse / glutamatergic synapse / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2 Å 分子置換 / 解像度: 2 Å | ||||||
 データ登録者 データ登録者 | Kishan, K.V.R. / Newcomer, M.E. | ||||||
 引用 引用 |  ジャーナル: Protein Sci. / 年: 2001 ジャーナル: Protein Sci. / 年: 2001タイトル: Effect of pH and salt bridges on structural assembly: molecular structures of the monomer and intertwined dimer of the Eps8 SH3 domain. 著者: Kishan, K.V. / Newcomer, M.E. / Rhodes, T.H. / Guilliot, S.D. #1:  ジャーナル: Nat.Struct.Biol. / 年: 1997 ジャーナル: Nat.Struct.Biol. / 年: 1997タイトル: The sh3 domain of eps8 exists as a novel intertwined dimer. 著者: Kishan, K.V. / Scita, G. / Wong, W.T. / Di Fiore, P.P. / Newcomer, M.E. | ||||||
| 履歴 |
| ||||||
| Remark 300 | BIOMOLECULE: 1 THIS ENTRY CONTAINS THE CRYSTALLOGRAPHIC ASYMMETRIC UNIT WHICH CONSISTS OF 2 CHAIN(S) ...BIOMOLECULE: 1 THIS ENTRY CONTAINS THE CRYSTALLOGRAPHIC ASYMMETRIC UNIT WHICH CONSISTS OF 2 CHAIN(S). SEE REMARK 350 FOR INFORMATION ON GENERATING THE BIOLOGICAL MOLECULE(S). THE BIOLOGICAL UNIT IS A MONOMER. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1i0c.cif.gz 1i0c.cif.gz | 37.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1i0c.ent.gz pdb1i0c.ent.gz | 25.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1i0c.json.gz 1i0c.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1i0c_validation.pdf.gz 1i0c_validation.pdf.gz | 429.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1i0c_full_validation.pdf.gz 1i0c_full_validation.pdf.gz | 431.1 KB | 表示 | |
| XML形式データ |  1i0c_validation.xml.gz 1i0c_validation.xml.gz | 7.6 KB | 表示 | |
| CIF形式データ |  1i0c_validation.cif.gz 1i0c_validation.cif.gz | 10 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/i0/1i0c https://data.pdbj.org/pub/pdb/validation_reports/i0/1i0c ftp://data.pdbj.org/pub/pdb/validation_reports/i0/1i0c ftp://data.pdbj.org/pub/pdb/validation_reports/i0/1i0c | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 7044.960 Da / 分子数: 2 / 断片: SH3 DOMAIN / 由来タイプ: 組換発現 / 由来: (組換発現)   #2: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.85 Å3/Da / 溶媒含有率: 33.42 % | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | pH: 4 / 詳細: pH 4.0 | |||||||||||||||||||||||||
| 結晶化 | *PLUS 手法: 蒸気拡散法, ハンギングドロップ法 | |||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
|---|---|
| 放射波長 | 相対比: 1 |
| 反射 | 解像度: 2→21.54 Å / Num. all: 6799 / Num. obs: 6795 / % possible obs: 100 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / 冗長度: 3.6 % / Biso Wilson estimate: 13.1 Å2 / Limit h max: 21 / Limit h min: -20 / Limit k max: 24 / Limit k min: -20 / Limit l max: 18 / Limit l min: 0 / Observed criterion F max: 318300.17 / Observed criterion F min: 0.55 / Rmerge(I) obs: 0.069 |
| 反射 | *PLUS % possible obs: 99.7 % / Num. measured all: 24299 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: pdb entry 1AOJ 解像度: 2→22 Å / Rfactor Rfree error: 0.009 / Occupancy max: 1 / Occupancy min: 1 / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: engh & huber
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: CNS bulk solvent model used / Bsol: 40.1813 Å2 / ksol: 0.342613 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 58.2 Å2 / Biso mean: 25.6 Å2 / Biso min: 12.69 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2→22 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Xplor file |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: CNS / バージョン: 0.5 / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最高解像度: 2 Å / 最低解像度: 22 Å / σ(F): 0 / % reflection Rfree: 10.9 % / Rfactor obs: 0.189 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | *PLUS Rfactor Rfree: 0.232 / % reflection Rfree: 10.4 % / Rfactor Rwork: 0.235 |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj

