+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1hzs | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
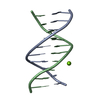
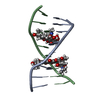
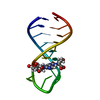
| タイトル | Crystal structure of a peptide nucleic acid duplex (BT-PNA) containing a bicyclic analogue of thymine | |||||||||
 要素 要素 | PEPTIDE NUCLEIC ACID | |||||||||
 キーワード キーワード | PEPTIDE NUCLEIC ACID / double helix / nucleobase analogue / P-form helix / left-handed / right-handed | |||||||||
| 機能・相同性 | DNA 機能・相同性情報 機能・相同性情報 | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.82 Å 分子置換 / 解像度: 1.82 Å | |||||||||
 データ登録者 データ登録者 | Eldrup, A.B. / Nielsen, B.B. / Haaima, G. / Rasmussen, H. / Kastrup, J.S. / Christensen, C. / Nielsen, P.E. | |||||||||
 引用 引用 | ジャーナル: Eur.J.Org.Chem. / 年: 2001 タイトル: 1,8-Naphthyridin-2(1H)-ones. Novel Bicyclic and Tricyclic Analogues of Thymine in Peptide Nucleic Acids (PNAs) 著者: Eldrup, A.B. / Nielsen, B.B. / Haaima, G. / Rasmussen, H. / Kastrup, J.S. / Christensen, C. / Nielsen, P.E. #1:  ジャーナル: Nat.Struct.Biol. / 年: 1997 ジャーナル: Nat.Struct.Biol. / 年: 1997タイトル: Crystal Structure of a Peptide Nucleic Acid (PNA) Duplex at 1.7 A Resolution 著者: Rasmussen, H. / Kastrup, J.S. / Nielsen, J.N. / Nielsen, J.M. / Nielsen, P.E. #2:  ジャーナル: New J.Chem. / 年: 1999 ジャーナル: New J.Chem. / 年: 1999タイトル: Peptide Nucleic Acids (PNA) Derived from N-(N-methylaminoethyl)glycine. Synthesis, Hybridization and Structural Properties 著者: Haima, G. / Rasmussen, H. / Schmidt, G. / Jensen, D.K. / Kastrup, J.S. / Stafshede, P.W. / Norden, B. / Buchardt, O. / Nielsen, P.E. | |||||||||
| 履歴 |
| |||||||||
| Remark 300 | BIOMOLECULE: 1,2 THIS ENTRY CONTAINS THE CRYSTALLOGRAPHIC ASYMMETRIC UNIT WHICH CONSISTS OF 4 ... BIOMOLECULE: 1,2 THIS ENTRY CONTAINS THE CRYSTALLOGRAPHIC ASYMMETRIC UNIT WHICH CONSISTS OF 4 CHAIN(S). SEE REMARK 350 FOR INFORMATION ON GENERATING THE BIOLOGICAL MOLECULE(S). CHAINS A AND B BUILD A RIGHT-HANDED PNA DOUBLE HELIX AND CHAINS C AND D BUILD A LEFT-HANDED DOUBLE HELIX. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1hzs.cif.gz 1hzs.cif.gz | 27.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1hzs.ent.gz pdb1hzs.ent.gz | 20.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1hzs.json.gz 1hzs.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1hzs_validation.pdf.gz 1hzs_validation.pdf.gz | 441 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1hzs_full_validation.pdf.gz 1hzs_full_validation.pdf.gz | 451.8 KB | 表示 | |
| XML形式データ |  1hzs_validation.xml.gz 1hzs_validation.xml.gz | 7.1 KB | 表示 | |
| CIF形式データ |  1hzs_validation.cif.gz 1hzs_validation.cif.gz | 8.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/hz/1hzs https://data.pdbj.org/pub/pdb/validation_reports/hz/1hzs ftp://data.pdbj.org/pub/pdb/validation_reports/hz/1hzs ftp://data.pdbj.org/pub/pdb/validation_reports/hz/1hzs | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1pupS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
| ||||||||
| 詳細 | PNA double helix |
- 要素
要素
| #1: DNA鎖 | 分子量: 1733.858 Da / 分子数: 4 / 由来タイプ: 合成 / 詳細: Synthesized as described in the primary reference #2: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.43 Å3/Da / 溶媒含有率: 49.28 % | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 4 詳細: 2-propanol, ammonium sulphate, pH 4, VAPOR DIFFUSION, HANGING DROP, temperature 293.0K | ||||||||||||||||||||
| 溶液の組成 |
| ||||||||||||||||||||
| 結晶化 | *PLUS 温度: 20 ℃ | ||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 293 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  EMBL/DESY, HAMBURG EMBL/DESY, HAMBURG  / ビームライン: X31 / 波長: 1.05 Å / ビームライン: X31 / 波長: 1.05 Å |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1999年8月18日 / 詳細: segmental toroidal mirror |
| 放射 | モノクロメーター: silicon / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.05 Å / 相対比: 1 |
| 反射 | 解像度: 1.82→30 Å / Num. all: 6245 / Num. obs: 6233 / % possible obs: 97.2 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / 冗長度: 3.8 % / Biso Wilson estimate: 19.1 Å2 / Rmerge(I) obs: 0.055 / Net I/σ(I): 20.1 |
| 反射 シェル | 解像度: 1.82→1.84 Å / 冗長度: 2.9 % / Rmerge(I) obs: 0.281 / Mean I/σ(I) obs: 2.6 / Num. unique all: 169 / % possible all: 81.6 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB entry 1PUP 解像度: 1.82→6 Å / 交差検証法: THROUGHOUT / σ(F): 3 / σ(I): 0 / 立体化学のターゲット値: own target values / 詳細: OWN PARAMETER AND TOPOLOGY FILE CREATED
| |||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 16.2 Å2 | |||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.82→6 Å
| |||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.82→1.9 Å / Total num. of bins used: 8 /
| |||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / バージョン: 3.851 / 分類: refinement X-PLOR / バージョン: 3.851 / 分類: refinement | |||||||||||||||||||||||||
| 精密化 | *PLUS 最低解像度: 6 Å / σ(F): 3 / % reflection Rfree: 10 % / Rfactor obs: 0.212 / Rfactor Rfree: 0.269 | |||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | |||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS Biso mean: 16.2 Å2 | |||||||||||||||||||||||||
| 拘束条件 | *PLUS
| |||||||||||||||||||||||||
| LS精密化 シェル | *PLUS Rfactor Rfree: 0.266 / % reflection Rfree: 10 % / Rfactor Rwork: 0.241 |
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj
