| 登録情報 | データベース: PDB / ID: 1hfg
|
|---|
| タイトル | NMR solution structure of vMIP-II 1-71 from Kaposi's sarcoma-associated herpesvirus (minimized average structure). |
|---|
 要素 要素 | VIRAL MACROPHAGE INFLAMMATORY PROTEIN-II |
|---|
 キーワード キーワード | CHEMOKINE / CXCR4 / ANATAGONIST / VMIP-II |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
CXCR chemokine receptor binding / chemokine activity / immune response / protein-containing complex / extracellular space類似検索 - 分子機能 CC chemokine, conserved site / Small cytokines (intercrine/chemokine) C-C subfamily signature. / Chemokine beta/gamma/delta / Intercrine alpha family (small cytokine C-X-C) (chemokine CXC). / Chemokine interleukin-8-like domain / Chemokine interleukin-8-like superfamily / Small cytokines (intecrine/chemokine), interleukin-8 like / OB fold (Dihydrolipoamide Acetyltransferase, E2P) - #40 / OB fold (Dihydrolipoamide Acetyltransferase, E2P) / Beta Barrel / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   HUMAN HERPESVIRUS 8 (ヘルペスウイルス) HUMAN HERPESVIRUS 8 (ヘルペスウイルス) |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
| Model type details | MINIMIZED AVERAGE |
|---|
 データ登録者 データ登録者 | Crump, M.P. / Elisseeva, E. / Gong, J.-H. / Clark-Lewis, I. / Sykes, B.D. |
|---|
 引用 引用 |  ジャーナル: FEBS Lett. / 年: 2001 ジャーナル: FEBS Lett. / 年: 2001
タイトル: Structure/Function of Human Herpesvirus-8 Mip-II (1-71) and the Antagonist N-Terminal Segment (1-10)
著者: Crump, M.P. / Elisseeva, E. / Gong, J.-H. / Clark-Lewis, I. / Sykes, B.D. |
|---|
| 履歴 | | 登録 | 2000年12月1日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2001年1月7日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年5月8日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2024年10月9日 | Group: Data collection / Database references ...Data collection / Database references / Other / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_database_status / pdbx_entry_details / pdbx_modification_feature
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_mr |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 HUMAN HERPESVIRUS 8 (ヘルペスウイルス)
HUMAN HERPESVIRUS 8 (ヘルペスウイルス) データ登録者
データ登録者 引用
引用 ジャーナル: FEBS Lett. / 年: 2001
ジャーナル: FEBS Lett. / 年: 2001 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1hfg.cif.gz
1hfg.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1hfg.ent.gz
pdb1hfg.ent.gz PDB形式
PDB形式 1hfg.json.gz
1hfg.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1hfg_validation.pdf.gz
1hfg_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1hfg_full_validation.pdf.gz
1hfg_full_validation.pdf.gz 1hfg_validation.xml.gz
1hfg_validation.xml.gz 1hfg_validation.cif.gz
1hfg_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/hf/1hfg
https://data.pdbj.org/pub/pdb/validation_reports/hf/1hfg ftp://data.pdbj.org/pub/pdb/validation_reports/hf/1hfg
ftp://data.pdbj.org/pub/pdb/validation_reports/hf/1hfg リンク
リンク 集合体
集合体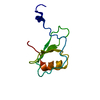
 要素
要素
 HUMAN HERPESVIRUS 8 (ヘルペスウイルス)
HUMAN HERPESVIRUS 8 (ヘルペスウイルス) 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj
 X-PLOR
X-PLOR