+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1gmk | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
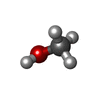
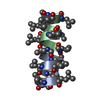
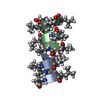
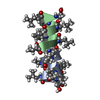
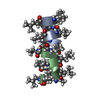
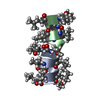
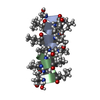
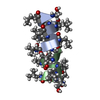
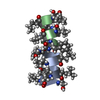
| タイトル | GRAMICIDIN/KSCN COMPLEX | |||||||||
 要素 要素 | GRAMICIDIN A | |||||||||
 キーワード キーワード | ANTIBIOTIC / GRAMICIDIN / ANTIFUNGAL / ANTIBACTERIAL / MEMBRANE ION CHANNEL / LINEAR GRAMICIDIN / DOUBLE HELIX | |||||||||
| 機能・相同性 | GRAMICIDIN A / : / METHANOL / THIOCYANATE ION / :  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  BREVIBACILLUS BREVIS (バクテリア) BREVIBACILLUS BREVIS (バクテリア) | |||||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.5 Å 分子置換 / 解像度: 2.5 Å | |||||||||
 データ登録者 データ登録者 | Doyle, D.A. / Wallace, B.A. | |||||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 1997 ジャーナル: J.Mol.Biol. / 年: 1997タイトル: Crystal Structure of the Gramicidin/Potassium Thiocyanate Complex. 著者: Doyle, D.A. / Wallace, B.A. #1:  ジャーナル: Biophys.J. / 年: 1994 ジャーナル: Biophys.J. / 年: 1994タイトル: The Structure of the Gramicidin/Kscn Complex 著者: Doyle, D.A. / Wallace, B.A. #2:  ジャーナル: Ann.N.Y.Acad.Sci. / 年: 1984 ジャーナル: Ann.N.Y.Acad.Sci. / 年: 1984タイトル: Crystalline Ion Complexes of Gramicidin A 著者: Kimball, M.R. / Wallace, B.A. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1gmk.cif.gz 1gmk.cif.gz | 27.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1gmk.ent.gz pdb1gmk.ent.gz | 21 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1gmk.json.gz 1gmk.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1gmk_validation.pdf.gz 1gmk_validation.pdf.gz | 403.7 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1gmk_full_validation.pdf.gz 1gmk_full_validation.pdf.gz | 409.3 KB | 表示 | |
| XML形式データ |  1gmk_validation.xml.gz 1gmk_validation.xml.gz | 4.5 KB | 表示 | |
| CIF形式データ |  1gmk_validation.cif.gz 1gmk_validation.cif.gz | 5.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/gm/1gmk https://data.pdbj.org/pub/pdb/validation_reports/gm/1gmk ftp://data.pdbj.org/pub/pdb/validation_reports/gm/1gmk ftp://data.pdbj.org/pub/pdb/validation_reports/gm/1gmk | HTTPS FTP |
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質・ペプチド |   タイプ: Polypeptide / クラス: 抗生剤 / 分子量: 1882.294 Da / 分子数: 4 / 由来タイプ: 天然 タイプ: Polypeptide / クラス: 抗生剤 / 分子量: 1882.294 Da / 分子数: 4 / 由来タイプ: 天然詳細: GRAMICIDIN A IS A HEXADECAMERIC HELICAL PEPTIDE WITH ALTERNATING D,L CHARACTERISTICS. THE N-TERM IS FORMYLATED (RESIDUE 0). THE C-TERM IS CAPPED WITH ETHANOLAMINE (RESIDUE 16). 由来: (天然)  BREVIBACILLUS BREVIS (バクテリア) / 参照: NOR: NOR00243, GRAMICIDIN A BREVIBACILLUS BREVIS (バクテリア) / 参照: NOR: NOR00243, GRAMICIDIN A#2: 化合物 | ChemComp-K / #3: 化合物 | ChemComp-SCN / #4: 化合物 | ChemComp-MOH / 構成要素の詳細 | GRAMICIDIN IS A HETEROGENEOUS MIXTURE OF SEVERAL COMPOUNDS INCLUDING GRAMICIDIN A, B AND C WHICH ...GRAMICIDIN | Has protein modification | Y | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | 解説: DATA COLLECTED AT ROOM TEMPERATURE WITH CRYSTAL IN METHANOL. | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 詳細: CRYSTALLIZED IN METHANOL | ||||||||||||||||||||
| 結晶化 | *PLUS 手法: unknown | ||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 293 K |
|---|---|
| 放射光源 | 由来: SEALED TUBE / タイプ: ENRAF-NONIUS / 波長: 1.5418 |
| 検出器 | タイプ: ENRAF-NONIUS FAST / 検出器: DIFFRACTOMETER / 日付: 1991年10月11日 |
| 放射 | モノクロメーター: NI FILTER / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 2.5→10 Å / Num. obs: 5476 / % possible obs: 73.3 % / Observed criterion σ(I): 3 / 冗長度: 2 % / Rmerge(I) obs: 0.0325 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: GRAMICIDIN/CSCL COMPLEX, THEN GRAMICIDIN/CSSCN COMPLEX, BOTH WITHOUT IONS 解像度: 2.5→10 Å / σ(F): 3 詳細: THE LOW TEMPERATURE FACTORS OF ATOMS N OF SCN 1, 2, 3, AND 4 ARE PROBABLY DUE TO POSITIONAL DISORDER OF THE THIOCYANATE MOLECULE WITH THE RESULT THAT OCCASIONALLY THE HEAVY SULFUR ATOM IS ...詳細: THE LOW TEMPERATURE FACTORS OF ATOMS N OF SCN 1, 2, 3, AND 4 ARE PROBABLY DUE TO POSITIONAL DISORDER OF THE THIOCYANATE MOLECULE WITH THE RESULT THAT OCCASIONALLY THE HEAVY SULFUR ATOM IS SITUATED CLOSE TO THE PRESENT LOCATION OF THE NITROGEN. TWO METHANOL MOLECULES, MOH 5 AND MOH 19, WERE PLACED IN DENSITIES THAT ARE BELIEVED TO BELONG TO COMBINED DISORDERED CONFORMATIONS OF TRP A 15 AND TRP C 9 (MOH 19), AND TRP D 15 AND TRP A 9 (MOH 5).
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.5→10 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / バージョン: 3.1 / 分類: refinement X-PLOR / バージョン: 3.1 / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー






















 PDBj
PDBj