+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1dqs | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | CRYSTAL STRUCTURE OF DEHYDROQUINATE SYNTHASE (DHQS) COMPLEXED WITH CARBAPHOSPHONATE, NAD+ AND ZN2+ | ||||||
 要素 要素 | PROTEIN (3-DEHYDROQUINATE SYNTHASE) | ||||||
 キーワード キーワード | LYASE / SHIKIMATE PATHWAY ENZYME / MULTI-STEP ENZYME / OXIDOREDUCTASE / PHOSPHATE ELIMINATION / INTRA MOLECULAR ALDOL CONDENSATION / NAD+ BINDING / ZN2+ BINDING / CYCLASE / AROMATIC AMINO ACID BIOSYNTHESIS | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報3-dehydroquinate synthase / 3-dehydroquinate synthase activity / shikimate kinase / shikimate kinase activity / shikimate dehydrogenase (NADP+) / shikimate 3-dehydrogenase (NADP+) activity / 3-phosphoshikimate 1-carboxyvinyltransferase / 3-phosphoshikimate 1-carboxyvinyltransferase activity / 3-dehydroquinate dehydratase / 3-dehydroquinate dehydratase activity ...3-dehydroquinate synthase / 3-dehydroquinate synthase activity / shikimate kinase / shikimate kinase activity / shikimate dehydrogenase (NADP+) / shikimate 3-dehydrogenase (NADP+) activity / 3-phosphoshikimate 1-carboxyvinyltransferase / 3-phosphoshikimate 1-carboxyvinyltransferase activity / 3-dehydroquinate dehydratase / 3-dehydroquinate dehydratase activity / chorismate biosynthetic process / aromatic amino acid family biosynthetic process / amino acid biosynthetic process / ATP binding / metal ion binding / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単一同系置換・異常分散 / 解像度: 1.8 Å 単一同系置換・異常分散 / 解像度: 1.8 Å | ||||||
 データ登録者 データ登録者 | Carpenter, E.P. / Hawkins, A.R. / Frost, J.W. / Brown, K.A. | ||||||
 引用 引用 |  ジャーナル: Nature / 年: 1998 ジャーナル: Nature / 年: 1998タイトル: Structure of dehydroquinate synthase reveals an active site capable of multistep catalysis. 著者: Carpenter, E.P. / Hawkins, A.R. / Frost, J.W. / Brown, K.A. #1:  ジャーナル: J.Am.Chem.Soc. / 年: 1998 ジャーナル: J.Am.Chem.Soc. / 年: 1998タイトル: Reactivation of 3-Dehydroquinate Synthase by Lanthanide Cations 著者: Moore, J.D. / Skinner, M.A. / Swatman, D.R. / Hawkins, A.R. / Brown, K.A. #2:  ジャーナル: J.Am.Chem.Soc. / 年: 1997 ジャーナル: J.Am.Chem.Soc. / 年: 1997タイトル: Cyclohexenyl and Cyclohexylidene Inhibitors of 3-Dehydroquinate Synthase: Active Site Interactions Relevant to Enzyme Mechanism and Inhibitor Design 著者: Montchamp, J.L. / Frost, J.W. #3:  ジャーナル: Biochem.J. / 年: 1994 ジャーナル: Biochem.J. / 年: 1994タイトル: Efficient Independent Activity of a Monomeric, Monofunctional Dehydroquinate Synthase Derived from the N-Terminus of the Pentafunctional Arom Protein of Aspergillus Nidulans 著者: Moore, J.D. / Coggins, J.R. / Virden, R. / Hawkins, A.R. #4:  ジャーナル: J.Gen.Microbiol. / 年: 1993 ジャーナル: J.Gen.Microbiol. / 年: 1993タイトル: The Pre-Chorismate (Shikimate) and Quinate Pathways in Filamentous Fungi: Theoretical and Practical Aspects 著者: Hawkins, A.R. / Lamb, H.K. / Moore, J.D. / Charles, I.G. / Roberts, C.F. #5:  ジャーナル: Biochem.J. / 年: 1992 ジャーナル: Biochem.J. / 年: 1992タイトル: Overproduction in Escherichia Coli of the Dehydroquinate Synthase Domain of the Aspergillus Nidulans Pentafunctional Arom Protein 著者: Van Den Hombergh, J.P. / Moore, J.D. / Charles, I.G. / Hawkins, A.R. #6:  ジャーナル: Biochemistry / 年: 1989 ジャーナル: Biochemistry / 年: 1989タイトル: Dehydroquinate Synthase: The Use of Substrate Analogues to Probe the Early Steps of the Catalyzed Reaction 著者: Bender, S.L. / Widlanski, T. / Knowles, J.R. #7:  ジャーナル: J.Biol.Chem. / 年: 1963 ジャーナル: J.Biol.Chem. / 年: 1963タイトル: The Enzymatic Conversion of 3-Deoxy-D-Arabino-Heptulosinic Acid 7-Phosphate to 5-Dehydroquinate 著者: Srinivasen, P.R. / Rothchild, J. / Sprinson, D.B. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1dqs.cif.gz 1dqs.cif.gz | 172.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1dqs.ent.gz pdb1dqs.ent.gz | 135.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1dqs.json.gz 1dqs.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/dq/1dqs https://data.pdbj.org/pub/pdb/validation_reports/dq/1dqs ftp://data.pdbj.org/pub/pdb/validation_reports/dq/1dqs ftp://data.pdbj.org/pub/pdb/validation_reports/dq/1dqs | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 2分子 AB
| #1: タンパク質 | 分子量: 42966.535 Da / 分子数: 2 断片: N-TERMINAL DOMAIN OF THE PENTAFUNCTIONAL AROM PROTEIN 由来タイプ: 組換発現 詳細: DHQS IS A DIMER, WITH ONE NAD+, ONE ZN2+ AND ONE SUBSTRATE ANALOGUE INHIBITOR (CARBAPHOSPHONATE, CRB) BOUND PER MONOMER 由来: (組換発現)   株 (発現宿主): GLW38 (AROB-) FROM THE GLASGOW CULTURE COLLECTION 参照: UniProt: P07547, EC: 4.6.1.3 |
|---|
-非ポリマー , 5種, 725分子 


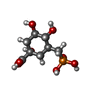





| #2: 化合物 | | #3: 化合物 | ChemComp-CL / | #4: 化合物 | #5: 化合物 | #6: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.9 Å3/Da / 溶媒含有率: 32 % |
|---|---|
| 結晶化 | pH: 6.8 / 詳細: pH 6.8 |
| 結晶 | *PLUS |
| 結晶化 | *PLUS 手法: unknown |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SRS SRS  / ビームライン: PX7.2 / 波長: 1.488 / ビームライン: PX7.2 / 波長: 1.488 |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1996年2月1日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.488 Å / 相対比: 1 |
| 反射 | 解像度: 1.7→25 Å / Num. obs: 79632 / % possible obs: 91.2 % / Observed criterion σ(I): -3 / 冗長度: 3.7 % / Biso Wilson estimate: 19.22 Å2 / Rmerge(I) obs: 0.068 / Net I/σ(I): 15.1 |
| 反射 シェル | 解像度: 1.7→1.76 Å / Rmerge(I) obs: 0.341 / Mean I/σ(I) obs: 3.3 / % possible all: 84.8 |
| 反射 | *PLUS Num. measured all: 297189 |
| 反射 シェル | *PLUS % possible obs: 84.8 % |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  単一同系置換・異常分散 / 解像度: 1.8→25 Å / 交差検証法: THROUGHOUT 単一同系置換・異常分散 / 解像度: 1.8→25 Å / 交差検証法: THROUGHOUT
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 20.58 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.249 Å / Luzzati d res low obs: 5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.8→25 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: REFMAC / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS Rfactor obs: 0.173 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj





