[English] 日本語
 Yorodumi
Yorodumi- PDB-1bw8: MU2 ADAPTIN SUBUNIT (AP50) OF AP2 ADAPTOR (SECOND DOMAIN), COMPLE... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 1bw8 | ||||||
|---|---|---|---|---|---|---|---|
| Title | MU2 ADAPTIN SUBUNIT (AP50) OF AP2 ADAPTOR (SECOND DOMAIN), COMPLEXED WITH EGFR INTERNALIZATION PEPTIDE FYRALM | ||||||
 Components Components |
| ||||||
 Keywords Keywords | PEPTIDE BINDING PROTEIN / ENDOCYTOSIS / ADAPTOR / PEPTIDE COMPLEX | ||||||
| Function / homology |  Function and homology information Function and homology informationditerpenoid metabolic process / positive regulation of prolactin secretion / response to hydroxyisoflavone / ovulation cycle / positive regulation of mucus secretion / Gap junction degradation / Formation of annular gap junctions / WNT5A-dependent internalization of FZD2, FZD5 and ROR2 / LDL clearance / tongue development ...diterpenoid metabolic process / positive regulation of prolactin secretion / response to hydroxyisoflavone / ovulation cycle / positive regulation of mucus secretion / Gap junction degradation / Formation of annular gap junctions / WNT5A-dependent internalization of FZD2, FZD5 and ROR2 / LDL clearance / tongue development / Retrograde neurotrophin signalling / VLDLR internalisation and degradation / WNT5A-dependent internalization of FZD4 / ubiquitin-dependent endocytosis / midgut development / extrinsic component of presynaptic endocytic zone membrane / MHC class II antigen presentation / hydrogen peroxide metabolic process / regulation of vesicle size / AP-2 adaptor complex / postsynaptic neurotransmitter receptor internalization / Recycling pathway of L1 / response to cobalamin / positive regulation of synaptic vesicle endocytosis / Cargo recognition for clathrin-mediated endocytosis / clathrin-cargo adaptor activity / Clathrin-mediated endocytosis / vesicle budding from membrane / clathrin-dependent endocytosis / signal sequence binding / positive regulation of bone resorption / negative regulation of mitotic cell cycle / multivesicular body, internal vesicle lumen / negative regulation of cardiocyte differentiation / positive regulation of epidermal growth factor receptor signaling pathway / positive regulation of protein kinase C signaling / epidermal growth factor receptor activity / epidermal growth factor binding / response to UV-A / low-density lipoprotein particle receptor binding / ERBB2-EGFR signaling pathway / morphogenesis of an epithelial fold / response to lipid / digestive tract morphogenesis / intracellular vesicle / positive regulation of vasoconstriction / eyelid development in camera-type eye / negative regulation of epidermal growth factor receptor signaling pathway / cerebral cortex cell migration / Trafficking of GluR2-containing AMPA receptors / protein tyrosine kinase activator activity / positive regulation of receptor internalization / positive regulation of phosphorylation / positive regulation of glial cell proliferation / endocytic vesicle / epidermis development / synaptic vesicle endocytosis / hair follicle development / positive regulation of G1/S transition of mitotic cell cycle / cellular response to dexamethasone stimulus / negative regulation of protein localization to plasma membrane / embryonic placenta development / positive regulation of synaptic transmission, glutamatergic / salivary gland morphogenesis / positive regulation of superoxide anion generation / neuron projection morphogenesis / clathrin-coated pit / transmembrane receptor protein tyrosine kinase activity / positive regulation of smooth muscle cell proliferation / astrocyte activation / receptor-mediated endocytosis / liver regeneration / lung development / synaptic membrane / positive regulation of DNA repair / positive regulation of DNA replication / epithelial cell proliferation / positive regulation of epithelial cell proliferation / positive regulation of protein localization to plasma membrane / intracellular protein transport / cellular response to amino acid stimulus / phosphatidylinositol 3-kinase/protein kinase B signal transduction / cellular response to estradiol stimulus / liver development / cellular response to mechanical stimulus / cell-cell adhesion / receptor protein-tyrosine kinase / response to calcium ion / circadian rhythm / negative regulation of protein catabolic process / receptor internalization / positive regulation of miRNA transcription / cellular response to growth factor stimulus / integrin binding / vasodilation / cellular response to xenobiotic stimulus / epidermal growth factor receptor signaling pathway / kinase binding / ruffle membrane / positive regulation of fibroblast proliferation Similarity search - Function | ||||||
| Biological species |  | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MIR / Resolution: 2.65 Å MIR / Resolution: 2.65 Å | ||||||
 Authors Authors | Owen, D.J. / Evans, P.R. | ||||||
 Citation Citation |  Journal: Science / Year: 1998 Journal: Science / Year: 1998Title: A structural explanation for the recognition of tyrosine-based endocytotic signals. Authors: Owen, D.J. / Evans, P.R. | ||||||
| History |
|
- Structure visualization
Structure visualization
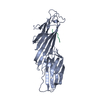
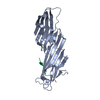
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  1bw8.cif.gz 1bw8.cif.gz | 66.7 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb1bw8.ent.gz pdb1bw8.ent.gz | 48.7 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  1bw8.json.gz 1bw8.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Summary document |  1bw8_validation.pdf.gz 1bw8_validation.pdf.gz | 430 KB | Display |  wwPDB validaton report wwPDB validaton report |
|---|---|---|---|---|
| Full document |  1bw8_full_validation.pdf.gz 1bw8_full_validation.pdf.gz | 437 KB | Display | |
| Data in XML |  1bw8_validation.xml.gz 1bw8_validation.xml.gz | 12.9 KB | Display | |
| Data in CIF |  1bw8_validation.cif.gz 1bw8_validation.cif.gz | 16.8 KB | Display | |
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/bw/1bw8 https://data.pdbj.org/pub/pdb/validation_reports/bw/1bw8 ftp://data.pdbj.org/pub/pdb/validation_reports/bw/1bw8 ftp://data.pdbj.org/pub/pdb/validation_reports/bw/1bw8 | HTTPS FTP |
-Related structure data
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
| #1: Protein | Mass: 36706.617 Da / Num. of mol.: 1 / Fragment: INTERNALIZATION SIGNAL BINDING DOMAIN Source method: isolated from a genetically manipulated source Source: (gene. exp.)   |
|---|---|
| #2: Protein/peptide | Mass: 800.988 Da / Num. of mol.: 1 / Source method: obtained synthetically / Details: THE PROTEIN WAS CHEMICALLY SYNTHESIZED / References: UniProt: A8IP97*PLUS |
| #3: Water | ChemComp-HOH / |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 4.45 Å3/Da / Density % sol: 72.33 % | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Crystal grow | Method: vapor diffusion, hanging drop / pH: 7.1 Details: HANGING DROP, 2.2M NACL, 0.4M NA/K PHOSPHATE, 10MM DTT 0.1M MES PH 7.1, 15% GLYCEROL, 16DEGREES, MOLAR RATIO OF PEPTIDE TO PROTEIN 3:1, vapor diffusion - hanging drop | ||||||||||||||||||||||||||||||||||||
| Crystal grow | *PLUS Temperature: 16 ℃ / Method: vapor diffusion, hanging drop / PH range low: 7.1 / PH range high: 6.5 | ||||||||||||||||||||||||||||||||||||
| Components of the solutions | *PLUS
|
-Data collection
| Diffraction | Mean temperature: 105 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SRS SRS  / Beamline: PX7.2 / Wavelength: 1.488 / Beamline: PX7.2 / Wavelength: 1.488 |
| Detector | Type: MAR scanner 300 mm plate / Detector: IMAGE PLATE / Date: May 15, 1998 / Details: MIRROR |
| Radiation | Monochromator: YES / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.488 Å / Relative weight: 1 |
| Reflection | Resolution: 2.65→35 Å / Num. obs: 119506 / % possible obs: 99 % / Observed criterion σ(I): 4 / Redundancy: 9.2 % / Biso Wilson estimate: 82 Å2 / Rmerge(I) obs: 0.094 / Rsym value: 0.094 / Net I/σ(I): 21.3 |
| Reflection shell | Resolution: 2.65→2.79 Å / Redundancy: 8.1 % / Rmerge(I) obs: 0.882 / Mean I/σ(I) obs: 2.1 / Rsym value: 0.882 / % possible all: 97 |
| Reflection | *PLUS Rmerge(I) obs: 0.089 |
| Reflection shell | *PLUS % possible obs: 96.7 % |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MIR / Resolution: 2.65→35 Å / SU B: 9.2 / SU ML: 0.2 / Cross valid method: THROUGHOUT / σ(F): 0 / ESU R: 0.35 / ESU R Free: 0.29 MIR / Resolution: 2.65→35 Å / SU B: 9.2 / SU ML: 0.2 / Cross valid method: THROUGHOUT / σ(F): 0 / ESU R: 0.35 / ESU R Free: 0.29
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 60 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.65→35 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Software | *PLUS Name: REFMAC / Classification: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement | *PLUS Lowest resolution: 35 Å / σ(F): 0 / % reflection Rfree: 4.4 % / Rfactor Rwork: 0.25 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | *PLUS Biso mean: 60 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints | *PLUS
|
 Movie
Movie Controller
Controller














 PDBj
PDBj





