| 登録情報 | データベース: PDB / ID: 1ao4
|
|---|
| タイトル | COBALT(III)-PEPLOMYCIN COMPLEX DETERMINED BY NMR STUDIES |
|---|
 要素 要素 | - (0) x 3-O-carbamoyl-alpha-D-mannopyranose-(1-2)-alpha-L-gulopyranose
- (1) x AGLYCON OF PEPLOMYCIN
- (2) x COBALT (III) ION
- (3) x HYDROGEN PEROXIDE
|
|---|
 キーワード キーワード | INHIBITOR / ANTICANCER DRUGS / PEPLOMYCIN / PEPLEOMYCIN / BLEOMYCIN / DNA / TWO-DIMENSIONAL NMR / SOLUTION STRUCTURES |
|---|
| 機能・相同性 | COBALT (III) ION / HYDROGEN PEROXIDE / AGLYCON OF PEPLOMYCIN 機能・相同性情報 機能・相同性情報 |
|---|
| 手法 | 溶液NMR |
|---|
 データ登録者 データ登録者 | Caceres-Cortes, J. / Sugiyama, H. / Ikudome, K. / Saito, I. / Wang, A.H.-J. |
|---|
 引用 引用 |  ジャーナル: Eur.J.Biochem. / 年: 1997 ジャーナル: Eur.J.Biochem. / 年: 1997
タイトル: Structures of cobalt(III)-pepleomycin and cobalt(III)-deglycopepleomycin (green forms) determined by NMR studies.
著者: Caceres-Cortes, J. / Sugiyama, H. / Ikudome, K. / Saito, I. / Wang, A.H. |
|---|
| 履歴 | | 登録 | 1997年7月16日 | 登録サイト: BNL / 処理サイト: RCSB |
|---|
| 改定 1.0 | 1999年7月30日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月26日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 2.0 | 2020年7月29日 | Group: Atomic model / Data collection ...Atomic model / Data collection / Derived calculations / Structure summary
カテゴリ: atom_site / chem_comp ...atom_site / chem_comp / entity / pdbx_branch_scheme / pdbx_chem_comp_identifier / pdbx_entity_branch / pdbx_entity_branch_descriptor / pdbx_entity_branch_link / pdbx_entity_branch_list / pdbx_entity_nonpoly / pdbx_nonpoly_scheme / pdbx_struct_assembly_gen / pdbx_struct_conn_angle / struct_asym / struct_conn / struct_site / struct_site_gen
Item: _atom_site.Cartn_x / _atom_site.Cartn_y ..._atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_atom_id / _atom_site.auth_comp_id / _atom_site.auth_seq_id / _atom_site.label_asym_id / _atom_site.label_atom_id / _atom_site.label_comp_id / _atom_site.label_entity_id / _atom_site.type_symbol / _chem_comp.mon_nstd_flag / _chem_comp.name / _chem_comp.type / _pdbx_struct_assembly_gen.asym_id_list / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.pdbx_value_order / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 2.1 | 2023年12月27日 | Group: Data collection / Database references / Structure summary
カテゴリ: chem_comp / chem_comp_atom ...chem_comp / chem_comp_atom / chem_comp_bond / database_2
Item: _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 データ登録者
データ登録者 引用
引用 ジャーナル: Eur.J.Biochem. / 年: 1997
ジャーナル: Eur.J.Biochem. / 年: 1997 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1ao4.cif.gz
1ao4.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1ao4.ent.gz
pdb1ao4.ent.gz PDB形式
PDB形式 1ao4.json.gz
1ao4.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1ao4_validation.pdf.gz
1ao4_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1ao4_full_validation.pdf.gz
1ao4_full_validation.pdf.gz 1ao4_validation.xml.gz
1ao4_validation.xml.gz 1ao4_validation.cif.gz
1ao4_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/ao/1ao4
https://data.pdbj.org/pub/pdb/validation_reports/ao/1ao4 ftp://data.pdbj.org/pub/pdb/validation_reports/ao/1ao4
ftp://data.pdbj.org/pub/pdb/validation_reports/ao/1ao4 リンク
リンク 集合体
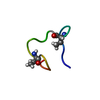
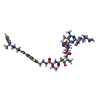
集合体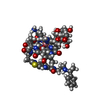
 要素
要素 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj





 X-PLOR
X-PLOR