+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 6y13 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
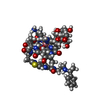
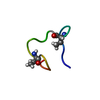
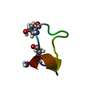
| Title | Bicyclic stapled peptide bp70 at 1.1 Angstrom resolution | |||||||||
 Components Components | bp70 | |||||||||
 Keywords Keywords | ANTIBIOTIC / Antimicrobial / Bicyclic / Lectin | |||||||||
| Biological species | synthetic construct (others) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / MOLECULAR REPLACEMENT /  molecular replacement / Resolution: 1.112 Å molecular replacement / Resolution: 1.112 Å | |||||||||
 Authors Authors | Baeriswyl, S. / Stocker, A. / Reymond, J.-L. | |||||||||
| Funding support |  Switzerland, 1items Switzerland, 1items
| |||||||||
 Citation Citation |  Journal: Rsc Chem Biol / Year: 2021 Journal: Rsc Chem Biol / Year: 2021Title: A mixed chirality alpha-helix in a stapled bicyclic and a linear antimicrobial peptide revealed by X-ray crystallography. Authors: Baeriswyl, S. / Personne, H. / Di Bonaventura, I. / Kohler, T. / van Delden, C. / Stocker, A. / Javor, S. / Reymond, J.L. #1:  Journal: Rsc Chem Biol / Year: 2021 Journal: Rsc Chem Biol / Year: 2021Title: A mixed chirality alpha-helix in a stapled bicyclic and a linear antimicrobial peptide revealed by X-ray crystallography. Authors: Baeriswyl, S. / Personne, H. / Di Bonaventura, I. / Kohler, T. / van Delden, C. / Stocker, A. / Javor, S. / Reymond, J.L. #2:  Journal: Rsc Chem Biol / Year: 2021 Journal: Rsc Chem Biol / Year: 2021Title: A mixed chirality alpha-helix in a stapled bicyclic and a linear antimicrobial peptide revealed by X-ray crystallography. Authors: Baeriswyl, S. / Personne, H. / Di Bonaventura, I. / Kohler, T. / van Delden, C. / Stocker, A. / Javor, S. / Reymond, J.L. #3:  Journal: Chemrxiv / Year: 2021 Journal: Chemrxiv / Year: 2021Title: Mixed chirality alpha-helix in a stapled bicyclic and a linear antimicrobial peptide revealed by X-ray crystallography Authors: Personne, H. / Baeriswyl, S. / Di Bonaventura, I. / Kohler, T. / van Delden, C. / Stocker, A. / Javor, S. / Reymond, J.-L. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  6y13.cif.gz 6y13.cif.gz | 18.5 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb6y13.ent.gz pdb6y13.ent.gz | 11.4 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  6y13.json.gz 6y13.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/y1/6y13 https://data.pdbj.org/pub/pdb/validation_reports/y1/6y13 ftp://data.pdbj.org/pub/pdb/validation_reports/y1/6y13 ftp://data.pdbj.org/pub/pdb/validation_reports/y1/6y13 | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  6y0uC  6y0vC 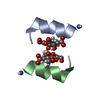 6y14C  6y1sC  7nefC  7newC C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
| #1: Protein/peptide | Mass: 1533.839 Da / Num. of mol.: 1 / Source method: obtained synthetically / Details: Bicyclic stapled peptide bp70 / Source: (synth.) synthetic construct (others) |
|---|---|
| Has ligand of interest | Y |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 1.44 Å3/Da / Density % sol: 38.76 % |
|---|---|
| Crystal grow | Temperature: 291 K / Method: vapor diffusion, sitting drop / pH: 6.5 Details: 1.6 M AMMONIUM SULFATE, 0.1 M MES MONOHYDRATE PH 6.5, 10% V/V 1,4-DIOXANE, 2% V/V GLYCEROL |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SLS SLS  / Beamline: X06DA / Wavelength: 0.8 Å / Beamline: X06DA / Wavelength: 0.8 Å |
| Detector | Type: DECTRIS PILATUS 2M-F / Detector: PIXEL / Date: Dec 20, 2017 Details: COLLIMATING MIRROR (M1), BARTELS MONOCHROMATOR (DCCM), TOROIDAL MIRROR (M2) |
| Radiation | Monochromator: BARTELS MONOCHROMATOR / Protocol: MAD / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.8 Å / Relative weight: 1 |
| Reflection | Resolution: 1.112→26.16 Å / Num. obs: 7887 / % possible obs: 94.7 % / Redundancy: 6.2 % / Biso Wilson estimate: 13.083 Å2 / CC1/2: 0.977 / CC star: 0.994 / R split: 0.136 / Rmerge(I) obs: 0.354 / Rpim(I) all: 0.096 / Rrim(I) all: 0.367 / Χ2: 0.83 / Net I/av σ(I): 1.24 / Net I/σ(I): 2.67 |
| Reflection shell | Resolution: 1.112→1.117 Å / Redundancy: 3.4 % / Mean I/σ(I) obs: 0.79 / Num. unique obs: 897 / CC1/2: 0.163 / Rrim(I) all: 1.24 / % possible all: 67.2 |
-Phasing
| Phasing | Method:  molecular replacement molecular replacement | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Phasing MR |
|
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: XXXX Resolution: 1.112→26.16 Å / Num. parameters: 946 / Num. restraintsaints: 1190 / Cross valid method: FREE R-VALUE / σ(F): 0 / Stereochemistry target values: ENGH AND HUBER
| |||||||||||||||||||||||||||||||||
| Solvent computation | Solvent model: MOEWS & KRETSINGER, J.MOL.BIOL.91(1973)201-228 | |||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 77.47 Å2 / Biso mean: 23.1498 Å2 / Biso min: 6.86 Å2 | |||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 1.112→26.16 Å
| |||||||||||||||||||||||||||||||||
| Refine LS restraints |
|
 Movie
Movie Controller
Controller








 PDBj
PDBj