+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8512 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the human HCN1 hyperpolarization-activated cyclic nucleotide-gated ion channel in complex with cAMP | |||||||||
 マップデータ マップデータ | Human HCN1 hyperpolarization-activated cyclic nucleotide-gated ion channel in complex with cAMP, low-pass filtered to 3.51 A, B-factor sharpened at -120 A2 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | pacemaker ion channel / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報intracellular cAMP-activated cation channel activity involved in regulation of presynaptic membrane potential / positive regulation of membrane hyperpolarization / HCN channels / general adaptation syndrome, behavioral process / HCN channel complex / retinal cone cell development / intracellularly cAMP-activated cation channel activity / negative regulation of action potential / regulation of SA node cell action potential / regulation of membrane depolarization ...intracellular cAMP-activated cation channel activity involved in regulation of presynaptic membrane potential / positive regulation of membrane hyperpolarization / HCN channels / general adaptation syndrome, behavioral process / HCN channel complex / retinal cone cell development / intracellularly cAMP-activated cation channel activity / negative regulation of action potential / regulation of SA node cell action potential / regulation of membrane depolarization / apical dendrite / maternal behavior / sodium ion import across plasma membrane / negative regulation of synaptic transmission, glutamatergic / response to L-glutamate / apical protein localization / voltage-gated monoatomic cation channel activity / voltage-gated sodium channel activity / potassium ion import across plasma membrane / phosphatidylinositol-3,4,5-trisphosphate binding / regulation of heart rate by cardiac conduction / voltage-gated potassium channel activity / potassium channel activity / neuronal action potential / cellular response to interferon-beta / cAMP binding / presynaptic active zone membrane / phosphatidylinositol-4,5-bisphosphate binding / axon terminus / potassium ion transmembrane transport / dendrite membrane / sodium ion transmembrane transport / cellular response to cAMP / dendritic shaft / regulation of membrane potential / response to calcium ion / protein homotetramerization / basolateral plasma membrane / postsynaptic membrane / axon / neuronal cell body / dendrite / glutamatergic synapse / cell surface / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.51 Å | |||||||||
 データ登録者 データ登録者 | Lee C-H / MacKinnon R | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2017 ジャーナル: Cell / 年: 2017タイトル: Structures of the Human HCN1 Hyperpolarization-Activated Channel. 著者: Chia-Hsueh Lee / Roderick MacKinnon /  要旨: Hyperpolarization-activated cyclic nucleotide-gated (HCN) channels underlie the control of rhythmic activity in cardiac and neuronal pacemaker cells. In HCN, the polarity of voltage dependence is ...Hyperpolarization-activated cyclic nucleotide-gated (HCN) channels underlie the control of rhythmic activity in cardiac and neuronal pacemaker cells. In HCN, the polarity of voltage dependence is uniquely reversed. Intracellular cyclic adenosine monophosphate (cAMP) levels tune the voltage response, enabling sympathetic nerve stimulation to increase the heart rate. We present cryo-electron microscopy structures of the human HCN channel in the absence and presence of cAMP at 3.5 Å resolution. HCN channels contain a K channel selectivity filter-forming sequence from which the amino acids create a unique structure that explains Na and K permeability. The voltage sensor adopts a depolarized conformation, and the pore is closed. An S4 helix of unprecedented length extends into the cytoplasm, contacts the C-linker, and twists the inner helical gate shut. cAMP binding rotates cytoplasmic domains to favor opening of the inner helical gate. These structures advance understanding of ion selectivity, reversed polarity gating, and cAMP regulation in HCN channels. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8512.map.gz emd_8512.map.gz | 49.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8512-v30.xml emd-8512-v30.xml emd-8512.xml emd-8512.xml | 14 KB 14 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8512.png emd_8512.png | 147.3 KB | ||
| Filedesc metadata |  emd-8512.cif.gz emd-8512.cif.gz | 5.6 KB | ||
| その他 |  emd_8512_additional.map.gz emd_8512_additional.map.gz | 37.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8512 http://ftp.pdbj.org/pub/emdb/structures/EMD-8512 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8512 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8512 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8512_validation.pdf.gz emd_8512_validation.pdf.gz | 589.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8512_full_validation.pdf.gz emd_8512_full_validation.pdf.gz | 589.5 KB | 表示 | |
| XML形式データ |  emd_8512_validation.xml.gz emd_8512_validation.xml.gz | 5.8 KB | 表示 | |
| CIF形式データ |  emd_8512_validation.cif.gz emd_8512_validation.cif.gz | 6.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8512 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8512 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8512 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8512 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8512.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8512.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human HCN1 hyperpolarization-activated cyclic nucleotide-gated ion channel in complex with cAMP, low-pass filtered to 3.51 A, B-factor sharpened at -120 A2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.3 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Human HCN1 hyperpolarization-activated cyclic nucleotide-gated ion channel in...
| ファイル | emd_8512_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human HCN1 hyperpolarization-activated cyclic nucleotide-gated ion channel in complex with cAMP: unfiltered, unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human HCN1 hyperpolarization-activated cyclic nucleotide-gated io...
| 全体 | 名称: Human HCN1 hyperpolarization-activated cyclic nucleotide-gated ion channel in complex with cAMP |
|---|---|
| 要素 |
|
-超分子 #1: Human HCN1 hyperpolarization-activated cyclic nucleotide-gated io...
| 超分子 | 名称: Human HCN1 hyperpolarization-activated cyclic nucleotide-gated ion channel in complex with cAMP タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Potassium/sodium hyperpolarization-activated cyclic nucleotide-ga...
| 分子 | 名称: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 11 タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 74.643734 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEGGGKPNSS SNSRDDGNSV FPAKASATGA GPAAAEKRLG TPPGGGGAGA KEHGNSVCFK VDGGGGGGGG GGGGEEPAGG FEDAEGPRR QYGFMQRQFT SMLQPGVNKF SLRMFGSQKA VEKEQERVKT AGFWIIHPYS DFRFYWDLIM LIMMVGNLVI I PVGITFFT ...文字列: MEGGGKPNSS SNSRDDGNSV FPAKASATGA GPAAAEKRLG TPPGGGGAGA KEHGNSVCFK VDGGGGGGGG GGGGEEPAGG FEDAEGPRR QYGFMQRQFT SMLQPGVNKF SLRMFGSQKA VEKEQERVKT AGFWIIHPYS DFRFYWDLIM LIMMVGNLVI I PVGITFFT EQTTTPWIIF NVASDTVFLL DLIMNFRTGT VNEDSSEIIL DPKVIKMNYL KSWFVVDFIS SIPVDYIFLI VE KGMDSEV YKTARALRIV RFTKILSLLR LLRLSRLIRY IHQWEEIFHM TYDLASAVVR IFNLIGMMLL LCHWDGCLQF LVP LLQDFP PDCWVSLNEM VNDSWGKQYS YALFKAMSHM LCIGYGAQAP VSMSDLWITM LSMIVGATCY AMFVGHATAL IQSL DSSRR QYQEKYKQVE QYMSFHKLPA DMRQKIHDYY EHRYQGKIFD EENILNELND PLREEIVNFN CRKLVATMPL FANAD PNFV TAMLSKLRFE VFQPGDYIIR EGAVGKKMYF IQHGVAGVIT KSSKEMKLTD GSYFGEICLL TKGRRTASVR ADTYCR LYS LSVDNFNEVL EEYPMMRRAF ETVAIDRLDR IGKKNSILLQ KFQKDLNTGV FNNQENEILK QIVKHDREMV QAALPRE SS SVLNTDPDAE KPRFASNL UniProtKB: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 1, Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 1 |
-分子 #2: Potassium/sodium hyperpolarization-activated cyclic nucleotide-ga...
| 分子 | 名称: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 11 タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 1.635006 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) |
-分子 #3: ADENOSINE-3',5'-CYCLIC-MONOPHOSPHATE
| 分子 | 名称: ADENOSINE-3',5'-CYCLIC-MONOPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 4 / 式: CMP |
|---|---|
| 分子量 | 理論値: 329.206 Da |
| Chemical component information | 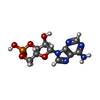 ChemComp-CMP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 1.78 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 3.3000000000000003 µm 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C4 (4回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.51 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 125339 |
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)