+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8451 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of bL17-depleted large ribosomal subunit assembly intermediate - Class E1 | ||||||||||||||||||||||||
 マップデータ マップデータ | Class E1 bL17-Depleted 50S Ribosomal Biogenesis Intermediate | ||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
| 生物種 |  | ||||||||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.2 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Tan Y / Davis JH / Lyumkis D | ||||||||||||||||||||||||
| 資金援助 |  米国, 7件 米国, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2016 ジャーナル: Cell / 年: 2016タイトル: Modular Assembly of the Bacterial Large Ribosomal Subunit. 著者: Joseph H Davis / Yong Zi Tan / Bridget Carragher / Clinton S Potter / Dmitry Lyumkis / James R Williamson /  要旨: The ribosome is a complex macromolecular machine and serves as an ideal system for understanding biological macromolecular assembly. Direct observation of ribosome assembly in vivo is difficult, as ...The ribosome is a complex macromolecular machine and serves as an ideal system for understanding biological macromolecular assembly. Direct observation of ribosome assembly in vivo is difficult, as few intermediates have been isolated and thoroughly characterized. Herein, we deploy a genetic system to starve cells of an essential ribosomal protein, which results in the accumulation of assembly intermediates that are competent for maturation. Quantitative mass spectrometry and single-particle cryo-electron microscopy reveal 13 distinct intermediates, which were each resolved to ∼4-5 Å resolution and could be placed in an assembly pathway. We find that ribosome biogenesis is a parallel process, that blocks of structured rRNA and proteins assemble cooperatively, and that the entire process is dynamic and can be "re-routed" through different pathways as needed. This work reveals the complex landscape of ribosome assembly in vivo and provides the requisite tools to characterize additional assembly pathways for ribosomes and other macromolecular machines. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8451.map.gz emd_8451.map.gz | 100.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8451-v30.xml emd-8451-v30.xml emd-8451.xml emd-8451.xml | 24.9 KB 24.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8451_fsc.xml emd_8451_fsc.xml | 11.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8451.png emd_8451.png | 160.9 KB | ||
| マスクデータ |  emd_8451_msk_1.map emd_8451_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| その他 |  emd_8451_additional.map.gz emd_8451_additional.map.gz emd_8451_half_map_1.map.gz emd_8451_half_map_1.map.gz emd_8451_half_map_2.map.gz emd_8451_half_map_2.map.gz | 20 MB 20.1 MB 20.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8451 http://ftp.pdbj.org/pub/emdb/structures/EMD-8451 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8451 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8451 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8451_validation.pdf.gz emd_8451_validation.pdf.gz | 78.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8451_full_validation.pdf.gz emd_8451_full_validation.pdf.gz | 77.8 KB | 表示 | |
| XML形式データ |  emd_8451_validation.xml.gz emd_8451_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8451 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8451 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8451 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8451 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8434C  8440C  8441C  8442C  8443C  8444C  8445C  8446C  8447C  8448C  8449C  8450C  8452C  8453C  8455C  8456C  8457C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10076 (タイトル: CryoEM Dataset of L17-Depleted 50S Ribosomal Intermediates EMPIAR-10076 (タイトル: CryoEM Dataset of L17-Depleted 50S Ribosomal IntermediatesData size: 50.3 Data #1: Particle stack of L17-depleted 50S Ribosomal Assembly Intermediates [picked particles - multiframe - unprocessed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8451.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8451.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Class E1 bL17-Depleted 50S Ribosomal Biogenesis Intermediate | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
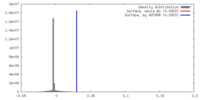
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_8451_msk_1.map emd_8451_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
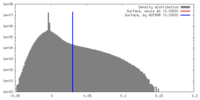
| 密度ヒストグラム |
-追加マップ: Class E1 bL17-Depleted 50S Ribosomal Biogenesis Intermediate -...
| ファイル | emd_8451_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Class E1 bL17-Depleted 50S Ribosomal Biogenesis Intermediate - Unsharpened | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Class E1 bL17-Depleted 50S Ribosomal Biogenesis Intermediate -...
| ファイル | emd_8451_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Class E1 bL17-Depleted 50S Ribosomal Biogenesis Intermediate - Halfmap 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Class E1 bL17-Depleted 50S Ribosomal Biogenesis Intermediate -...
| ファイル | emd_8451_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Class E1 bL17-Depleted 50S Ribosomal Biogenesis Intermediate - Halfmap 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : bL17-depleted large ribosomal subunit assembly intermediate - Class E1
| 全体 | 名称: bL17-depleted large ribosomal subunit assembly intermediate - Class E1 |
|---|---|
| 要素 |
|
-超分子 #1: bL17-depleted large ribosomal subunit assembly intermediate - Class E1
| 超分子 | 名称: bL17-depleted large ribosomal subunit assembly intermediate - Class E1 タイプ: complex / ID: 1 / 親要素: 0 詳細: E. coli large ribosome subunit purified from bL17-depleted cells. |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
詳細: Most of the sucrose was removed by spin concentration | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: C-Flat CF-1.2/1.3-4Au / 材質: GOLD / メッシュ: 400 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 298 K / 装置: GATAN CRYOPLUNGE 3 / 詳細: 3 microliter of the sample was added.. | ||||||||||||||||||
| 詳細 | Fraction was obtained directly from a sucrose gradient, and was spin-concentrated in a 100 kDa MW-cutoff filter. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 詳細 | In order to account for highly preferred orientation of the specimen, data was acquired using tilts ranging from 10-60 degrees, in addition to untilted images at 0 degrees. The CTF was estimated on a per-particle basis to account for the gradient of CTF values across individual micrographs |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-50 / 撮影したグリッド数: 2 / 実像数: 2479 / 平均露光時間: 10.0 sec. / 平均電子線量: 34.27 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 倍率(補正後): 38167 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)