+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7786 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | human PKD2 F604P mutant | |||||||||
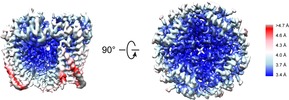
 マップデータ マップデータ | Human PKD2 F604P mutant | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Ion Channel / TRP channel / PKD2 / PC2 / TRPP2 / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報detection of nodal flow / metanephric smooth muscle tissue development / metanephric cortex development / metanephric cortical collecting duct development / metanephric distal tubule development / polycystin complex / mesonephric tubule development / mesonephric duct development / metanephric part of ureteric bud development / renal tubule morphogenesis ...detection of nodal flow / metanephric smooth muscle tissue development / metanephric cortex development / metanephric cortical collecting duct development / metanephric distal tubule development / polycystin complex / mesonephric tubule development / mesonephric duct development / metanephric part of ureteric bud development / renal tubule morphogenesis / determination of liver left/right asymmetry / HLH domain binding / metanephric ascending thin limb development / metanephric mesenchyme development / metanephric S-shaped body morphogenesis / basal cortex / renal artery morphogenesis / positive regulation of inositol 1,4,5-trisphosphate-sensitive calcium-release channel activity / calcium-induced calcium release activity / migrasome / cilium organization / VxPx cargo-targeting to cilium / detection of mechanical stimulus / muscle alpha-actinin binding / regulation of calcium ion import / voltage-gated monoatomic ion channel activity / placenta blood vessel development / cellular response to hydrostatic pressure / cation channel complex / cellular response to fluid shear stress / outward rectifier potassium channel activity / actinin binding / cellular response to osmotic stress / non-motile cilium / determination of left/right symmetry / voltage-gated monoatomic cation channel activity / inorganic cation transmembrane transport / aorta development / neural tube development / motile cilium / voltage-gated sodium channel activity / ciliary membrane / branching involved in ureteric bud morphogenesis / protein heterotetramerization / negative regulation of G1/S transition of mitotic cell cycle / spinal cord development / cytoplasmic side of endoplasmic reticulum membrane / heart looping / centrosome duplication / voltage-gated potassium channel activity / cell surface receptor signaling pathway via JAK-STAT / potassium channel activity / embryonic placenta development / voltage-gated calcium channel activity / transcription regulator inhibitor activity / monoatomic cation channel activity / cytoskeletal protein binding / cellular response to cAMP / release of sequestered calcium ion into cytosol / potassium ion transmembrane transport / sodium ion transmembrane transport / cytoplasmic vesicle membrane / cellular response to calcium ion / liver development / basal plasma membrane / lumenal side of endoplasmic reticulum membrane / cellular response to reactive oxygen species / establishment of localization in cell / phosphoprotein binding / protein tetramerization / calcium ion transmembrane transport / Wnt signaling pathway / intracellular calcium ion homeostasis / calcium ion transport / mitotic spindle / positive regulation of nitric oxide biosynthetic process / cell-cell junction / lamellipodium / regulation of cell population proliferation / heart development / ATPase binding / positive regulation of cytosolic calcium ion concentration / basolateral plasma membrane / protein homotetramerization / transmembrane transporter binding / cell surface receptor signaling pathway / regulation of cell cycle / ciliary basal body / cilium / signaling receptor binding / negative regulation of cell population proliferation / calcium ion binding / positive regulation of gene expression / endoplasmic reticulum membrane / endoplasmic reticulum / Golgi apparatus / protein homodimerization activity / positive regulation of transcription by RNA polymerase II / extracellular exosome / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.54 Å | |||||||||
 データ登録者 データ登録者 | Zheng W / Yang X | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2018 ジャーナル: Nat Commun / 年: 2018タイトル: Hydrophobic pore gates regulate ion permeation in polycystic kidney disease 2 and 2L1 channels. 著者: Wang Zheng / Xiaoyong Yang / Ruikun Hu / Ruiqi Cai / Laura Hofmann / Zhifei Wang / Qiaolin Hu / Xiong Liu / David Bulkley / Yong Yu / Jingfeng Tang / Veit Flockerzi / Ying Cao / Erhu Cao / Xing-Zhen Chen /     要旨: PKD2 and PKD1 genes are mutated in human autosomal dominant polycystic kidney disease. PKD2 can form either a homomeric cation channel or a heteromeric complex with the PKD1 receptor, presumed to ...PKD2 and PKD1 genes are mutated in human autosomal dominant polycystic kidney disease. PKD2 can form either a homomeric cation channel or a heteromeric complex with the PKD1 receptor, presumed to respond to ligand(s) and/or mechanical stimuli. Here, we identify a two-residue hydrophobic gate in PKD2L1, and a single-residue hydrophobic gate in PKD2. We find that a PKD2 gain-of-function gate mutant effectively rescues PKD2 knockdown-induced phenotypes in embryonic zebrafish. The structure of a PKD2 activating mutant F604P by cryo-electron microscopy reveals a π- to α-helix transition within the pore-lining helix S6 that leads to repositioning of the gate residue and channel activation. Overall the results identify hydrophobic gates and a gating mechanism of PKD2 and PKD2L1. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7786.map.gz emd_7786.map.gz | 3.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7786-v30.xml emd-7786-v30.xml emd-7786.xml emd-7786.xml | 19.5 KB 19.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7786.png emd_7786.png | 477.7 KB | ||
| Filedesc metadata |  emd-7786.cif.gz emd-7786.cif.gz | 6.5 KB | ||
| その他 |  emd_7786_half_map_1.map.gz emd_7786_half_map_1.map.gz emd_7786_half_map_2.map.gz emd_7786_half_map_2.map.gz | 17.7 MB 17.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7786 http://ftp.pdbj.org/pub/emdb/structures/EMD-7786 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7786 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7786 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7786.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7786.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human PKD2 F604P mutant | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.2156 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Human PKD2 F604P mutant
| ファイル | emd_7786_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human PKD2 F604P mutant | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Human PKD2 F604P mutant
| ファイル | emd_7786_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human PKD2 F604P mutant | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : PKD2
| 全体 | 名称: PKD2 |
|---|---|
| 要素 |
|
-超分子 #1: PKD2
| 超分子 | 名称: PKD2 / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 300 kDa/nm |
-分子 #1: Polycystin-2
| 分子 | 名称: Polycystin-2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 84.996336 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EIEMQRIRQA AARDPPAGAA ASPSPPLSSC SRQAWSRDNP GFEAEEEEEE VEGEEGGMVV EMDVEWRPGS RRSAASSAVS SVGARSRGL GGYHGAGHPS GRRRRREDQG PPCPSPVGGG DPLHRHLPLE GQPPRVAWAE RLVRGLRGLW GTRLMEESST N REKYLKSV ...文字列: EIEMQRIRQA AARDPPAGAA ASPSPPLSSC SRQAWSRDNP GFEAEEEEEE VEGEEGGMVV EMDVEWRPGS RRSAASSAVS SVGARSRGL GGYHGAGHPS GRRRRREDQG PPCPSPVGGG DPLHRHLPLE GQPPRVAWAE RLVRGLRGLW GTRLMEESST N REKYLKSV LRELVTYLLF LIVLCILTYG MMSSNVYYYT RMMSQLFLDT PVSKTEKTNF KTLSSMEDFW KFTEGSLLDG LY WKMQPSN QTEADNRSFI FYENLLLGVP RIRQLRVRNG SCSIPQDLRD EIKECYDVYS VSSEDRAPFG PRNGTAWIYT SEK DLNGSS HWGIIATYSG AGYYLDLSRT REETAAQVAS LKKNVWLDRG TRATFIDFSV YNANINLFCV VRLLVEFPAT GGVI PSWQF QPLKLIRYVT TFDFFLAACE IIFCFFIFYY VVEEILEIRI HKLHYFRSFW NCLDVVIVVL SVVAIGINIY RTSNV EVLL QFLEDQNTFP NFEHLAYWQI QFNNIAAVTV FFVWIKLFKF INFNRTMSQL STTMSRCAKD LFGFAIMPFI IFLAYA QLA YLVFGTQVDD FSTFQECIFT QFRIILGDIN FAEIEEANRV LGPIYFTTFV FFMFFILLNM FLAIINDTYS EVKSDLA QQ KAEMELSDLI RKGYHKALVK LKLKKNTVDD ISESLRQGGG KLNFDELRQD LKGKGHTDAE IEAIFTKYDQ DGDQELTE H EHQQMRDDLE KEREDLDLD UniProtKB: Polycystin-2 |
-分子 #2: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 2 / コピー数: 12 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)