+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7007 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of Mical Oxidized Actin (Class 1) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | F-Actin / Mical / cytoskeleton / helix / STRUCTURAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytoskeletal motor activator activity / myosin heavy chain binding / tropomyosin binding / actin filament bundle / troponin I binding / filamentous actin / mesenchyme migration / actin filament bundle assembly / skeletal muscle myofibril / striated muscle thin filament ...cytoskeletal motor activator activity / myosin heavy chain binding / tropomyosin binding / actin filament bundle / troponin I binding / filamentous actin / mesenchyme migration / actin filament bundle assembly / skeletal muscle myofibril / striated muscle thin filament / skeletal muscle thin filament assembly / actin monomer binding / skeletal muscle fiber development / stress fiber / titin binding / actin filament polymerization / actin filament / filopodium / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / calcium-dependent protein binding / lamellipodium / cell body / hydrolase activity / protein domain specific binding / calcium ion binding / positive regulation of gene expression / magnesium ion binding / ATP binding / identical protein binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
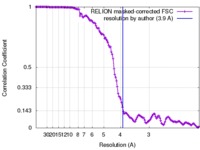
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Grintsevich EE / Ge P | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2017 ジャーナル: Nat Commun / 年: 2017タイトル: Catastrophic disassembly of actin filaments via Mical-mediated oxidation. 著者: Elena E Grintsevich / Peng Ge / Michael R Sawaya / Hunkar Gizem Yesilyurt / Jonathan R Terman / Z Hong Zhou / Emil Reisler /  要旨: Actin filament assembly and disassembly are vital for cell functions. MICAL Redox enzymes are important post-translational effectors of actin that stereo-specifically oxidize actin's M44 and M47 ...Actin filament assembly and disassembly are vital for cell functions. MICAL Redox enzymes are important post-translational effectors of actin that stereo-specifically oxidize actin's M44 and M47 residues to induce cellular F-actin disassembly. Here we show that Mical-oxidized (Mox) actin can undergo extremely fast (84 subunits/s) disassembly, which depends on F-actin's nucleotide-bound state. Using near-atomic resolution cryoEM reconstruction and single filament TIRF microscopy we identify two dynamic and structural states of Mox-actin. Modeling actin's D-loop region based on our 3.9 Å cryoEM reconstruction suggests that oxidation by Mical reorients the side chain of M44 and induces a new intermolecular interaction of actin residue M47 (M47-O-T351). Site-directed mutagenesis reveals that this interaction promotes Mox-actin instability. Moreover, we find that Mical oxidation of actin allows for cofilin-mediated severing even in the presence of inorganic phosphate. Thus, in conjunction with cofilin, Mical oxidation of actin promotes F-actin disassembly independent of the nucleotide-bound state. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7007.map.gz emd_7007.map.gz | 14.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7007-v30.xml emd-7007-v30.xml emd-7007.xml emd-7007.xml | 16.8 KB 16.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7007_fsc.xml emd_7007_fsc.xml | 13.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7007.png emd_7007.png | 52 KB | ||
| Filedesc metadata |  emd-7007.cif.gz emd-7007.cif.gz | 6.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7007 http://ftp.pdbj.org/pub/emdb/structures/EMD-7007 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7007 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7007 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7007_validation.pdf.gz emd_7007_validation.pdf.gz | 636.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7007_full_validation.pdf.gz emd_7007_full_validation.pdf.gz | 635.9 KB | 表示 | |
| XML形式データ |  emd_7007_validation.xml.gz emd_7007_validation.xml.gz | 11.5 KB | 表示 | |
| CIF形式データ |  emd_7007_validation.cif.gz emd_7007_validation.cif.gz | 15.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7007 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7007 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7007 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7007 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7007.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7007.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.041 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
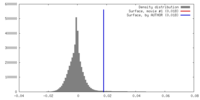
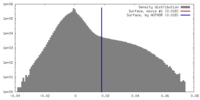
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Filamentous Actin Oxidized by Mical at M44 and M47
| 全体 | 名称: Filamentous Actin Oxidized by Mical at M44 and M47 |
|---|---|
| 要素 |
|
-超分子 #1: Filamentous Actin Oxidized by Mical at M44 and M47
| 超分子 | 名称: Filamentous Actin Oxidized by Mical at M44 and M47 / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Actin, alpha skeletal muscle
| 分子 | 名称: Actin, alpha skeletal muscle / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 42.141973 KDa |
| 配列 | 文字列: MCDEDETTAL VCDNGSGLVK AGFAGDDAPR AVFPSIVGRP RHQGV(SME)VG(SME)G QKDSYVGDEA QSKRGILTLK YP IE(HIC)GIIT NWDDMEKIWH HTFYNELRVA PEEHPTLLTE APLNPKANRE KMTQIMFETF NVPAMYVAIQ AVLSLYASG RTTGIVLDSG ...文字列: MCDEDETTAL VCDNGSGLVK AGFAGDDAPR AVFPSIVGRP RHQGV(SME)VG(SME)G QKDSYVGDEA QSKRGILTLK YP IE(HIC)GIIT NWDDMEKIWH HTFYNELRVA PEEHPTLLTE APLNPKANRE KMTQIMFETF NVPAMYVAIQ AVLSLYASG RTTGIVLDSG DGVTHNVPIY EGYALPHAIM RLDLAGRDLT DYLMKILTER GYSFVTTAER EIVRDIKEKL CYVALDFENE MATAASSSS LEKSYELPDG QVITIGNERF RCPETLFQPS FIGMESAGIH ETTYNSIMKC DIDIRKDLYA NNVMSGGTTM Y PGIADRMQ KEITALAPST MKIKIIAPPE RKYSVWIGGS ILASLSTFQQ MWITKQEYDE AGPSIVHRKC F UniProtKB: Actin, alpha skeletal muscle |
-分子 #2: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 3 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 240 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV / 詳細: Blot Force 1, Blot time 4s. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: Gatan Bio Quantum |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 4-20 / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-6av9: |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)