+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6827 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of the Exocyst Complex | |||||||||||||||
 マップデータ マップデータ | Cryo-EM map of the yeast Exocyst complex overall structure at 5.5A resolution | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | exocyst / coiled-coil / EXOCYTOSIS | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報vesicle tethering involved in exocytosis / exocyst assembly / exocyst localization / negative regulation of SNARE complex assembly / endoplasmic reticulum inheritance / exocyst / prospore membrane / incipient cellular bud site / cellular bud tip / Golgi inheritance ...vesicle tethering involved in exocytosis / exocyst assembly / exocyst localization / negative regulation of SNARE complex assembly / endoplasmic reticulum inheritance / exocyst / prospore membrane / incipient cellular bud site / cellular bud tip / Golgi inheritance / cellular bud neck / Golgi to plasma membrane transport / mating projection tip / vesicle docking involved in exocytosis / spliceosomal complex assembly / exocytosis / transport vesicle / Rho protein signal transduction / phosphatidylinositol-4,5-bisphosphate binding / SNARE binding / cell periphery / intracellular protein transport / small GTPase binding / intracellular protein localization / protein transport / plasma membrane / cytoplasm 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   | |||||||||||||||
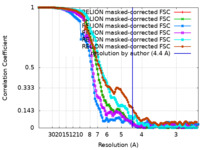
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.4 Å | |||||||||||||||
 データ登録者 データ登録者 | Mei K / Li Y | |||||||||||||||
| 資金援助 |  中国, 中国,  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2018 ジャーナル: Nat Struct Mol Biol / 年: 2018タイトル: Cryo-EM structure of the exocyst complex. 著者: Kunrong Mei / Yan Li / Shaoxiao Wang / Guangcan Shao / Jia Wang / Yuehe Ding / Guangzuo Luo / Peng Yue / Jun-Jie Liu / Xinquan Wang / Meng-Qiu Dong / Hong-Wei Wang / Wei Guo /   要旨: The exocyst is an evolutionarily conserved octameric protein complex that mediates the tethering of post-Golgi secretory vesicles to the plasma membrane during exocytosis and is implicated in many ...The exocyst is an evolutionarily conserved octameric protein complex that mediates the tethering of post-Golgi secretory vesicles to the plasma membrane during exocytosis and is implicated in many cellular processes such as cell polarization, cytokinesis, ciliogenesis and tumor invasion. Using cryo-EM and chemical cross-linking MS (CXMS), we solved the structure of the Saccharomyces cerevisiae exocyst complex at an average resolution of 4.4 Å. Our model revealed the architecture of the exocyst and led to the identification of the helical bundles that mediate the assembly of the complex at its core. Sequence analysis suggests that these regions are evolutionarily conserved across eukaryotic systems. Additional cell biological data suggest a mechanism for exocyst assembly that leads to vesicle tethering at the plasma membrane. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6827.map.gz emd_6827.map.gz | 12.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6827-v30.xml emd-6827-v30.xml emd-6827.xml emd-6827.xml | 44.1 KB 44.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_6827_fsc.xml emd_6827_fsc.xml emd_6827_fsc_1.xml emd_6827_fsc_1.xml emd_6827_fsc_2.xml emd_6827_fsc_2.xml emd_6827_fsc_3.xml emd_6827_fsc_3.xml emd_6827_fsc_4.xml emd_6827_fsc_4.xml emd_6827_fsc_5.xml emd_6827_fsc_5.xml | 12.7 KB 12.7 KB 12.8 KB 12.8 KB 12.7 KB 12.7 KB | 表示 表示 表示 表示 表示 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_6827.png emd_6827.png emd_6827_1.png emd_6827_1.png emd_6827_2.png emd_6827_2.png emd_6827_3.png emd_6827_3.png emd_6827_4.png emd_6827_4.png emd_6827_5.png emd_6827_5.png | 41.6 KB 41.6 KB 32.4 KB 27.3 KB 30.4 KB 47 KB | ||
| マスクデータ |  emd_6827_msk_1.map emd_6827_msk_1.map emd_6827_msk_2.map emd_6827_msk_2.map emd_6827_msk_3.map emd_6827_msk_3.map emd_6827_msk_4.map emd_6827_msk_4.map emd_6827_msk_5.map emd_6827_msk_5.map | 178 MB 178 MB 178 MB 178 MB 178 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-6827.cif.gz emd-6827.cif.gz | 12.5 KB | ||
| その他 |  emd_6827_additional_1.map.gz emd_6827_additional_1.map.gz emd_6827_additional_2.map.gz emd_6827_additional_2.map.gz emd_6827_additional_3.map.gz emd_6827_additional_3.map.gz emd_6827_additional_4.map.gz emd_6827_additional_4.map.gz | 9.8 MB 9.7 MB 14.4 MB 13.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6827 http://ftp.pdbj.org/pub/emdb/structures/EMD-6827 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6827 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6827 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6827_validation.pdf.gz emd_6827_validation.pdf.gz | 471.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6827_full_validation.pdf.gz emd_6827_full_validation.pdf.gz | 471 KB | 表示 | |
| XML形式データ |  emd_6827_validation.xml.gz emd_6827_validation.xml.gz | 13.1 KB | 表示 | |
| CIF形式データ |  emd_6827_validation.cif.gz emd_6827_validation.cif.gz | 17.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6827 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6827 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6827 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6827 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5yfpMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10206 (タイトル: Cryo electron microscopy micrographs of yeast Exocyst complex EMPIAR-10206 (タイトル: Cryo electron microscopy micrographs of yeast Exocyst complexData size: 10.0 TB Data #1: Unaligned multi-frame micrographs of Yeast Exocyst complex [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6827.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6827.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of the yeast Exocyst complex overall structure at 5.5A resolution | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.30654 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_6827_msk_1.map emd_6827_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
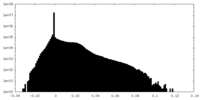
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_6827_msk_2.map emd_6827_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
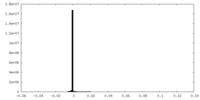
| 密度ヒストグラム |
-マスク #3
| ファイル |  emd_6827_msk_3.map emd_6827_msk_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
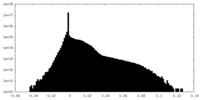
| 密度ヒストグラム |
-マスク #4
| ファイル |  emd_6827_msk_4.map emd_6827_msk_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
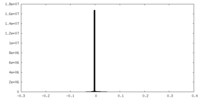
| 密度ヒストグラム |
-マスク #5
| ファイル |  emd_6827_msk_5.map emd_6827_msk_5.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Cryo-EM map of the yeast Exocyst complex head-part...
| ファイル | emd_6827_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of the yeast Exocyst complex head-part structure at 6.7A resolution | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Cryo-EM map of the yeast Exocyst complex top...
| ファイル | emd_6827_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of the yeast Exocyst complex top head-part structure at 6.2A resolution | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Cryo-EM map of the yeast Exocyst complex bottom...
| ファイル | emd_6827_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of the yeast Exocyst complex bottom leg-part structure at 4.6A resolution | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Cryo-EM map of the yeast Exocyst complex body-part...
| ファイル | emd_6827_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of the yeast Exocyst complex body-part structure at 4.4A resolution | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM map of the Yeast Exocyst complex overall structure at 5.5...
| 全体 | 名称: Cryo-EM map of the Yeast Exocyst complex overall structure at 5.5A resolution |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM map of the Yeast Exocyst complex overall structure at 5.5...
| 超分子 | 名称: Cryo-EM map of the Yeast Exocyst complex overall structure at 5.5A resolution タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: overall map of Yeast Exocyst complex |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 844 KDa |
-分子 #1: Exocyst complex component SEC3
| 分子 | 名称: Exocyst complex component SEC3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 154.889547 KDa |
| 配列 | 文字列: MRSSKSPFKR KSHSRETSHD ENTSFFHKRT ISGSSAHHSR NVSQGAVPSS APPVSGGNYS HKRNVSRASN SSQTSNFLAE QYERDRKAI INCCFSRPDH KTGEPPNNYI THVRIIEDSK FPSSRPPPDS KLENKKKRLL ILSAKPNNAK LIQIHKAREN S DGSFQIGR ...文字列: MRSSKSPFKR KSHSRETSHD ENTSFFHKRT ISGSSAHHSR NVSQGAVPSS APPVSGGNYS HKRNVSRASN SSQTSNFLAE QYERDRKAI INCCFSRPDH KTGEPPNNYI THVRIIEDSK FPSSRPPPDS KLENKKKRLL ILSAKPNNAK LIQIHKAREN S DGSFQIGR TWQLTELVRV EKDLEISEGF ILTMSKKYYW ETNSAKERTV FIKSLITLYI QTFEGHVPEL VNWDLSLFYL DE RSYQRAV ITNRPGSVSP IKSPTSNFTT NTTQSVGSVP FSAPTERTRR SETESVNPVS TPASVEYHAG MKSLNKAPYS SNS TLNEVN KRYELEQQQQ QEEAELRRLE EQKRLQLQKE NEMKRLEEER RIKQEERKRQ MELEHQRQLE EEERKRQMEL EAKK QMELK RQRQFEEEQR LKKERELLEI QRKQREQETA ERLKKEEQEA LAKKEEEEKS KRNKVDNESY TQEINGKVDN LLEDL NAVL AEETETTPTM QNGTYVPERS TARAHDQLKK PLNIAKVESL GGSDLNDSIS LSDEIAGLNT SNLSGEDQDE KNDLSF EKG DEVRYSNNFE GEAPHVYHEV SIIQEEAPAV SQKLILPEEN NESEALIESK EEIKTMENID DEVLLEILTD INWSIED DA DSMIERIDLR LAETEYLFNQ NLLSLQKIGP NIRPYEDKVN DECHRIIPTL SLFLMEMSNF SNDIENVESQ DNGLQVES A NKKLLWNTLD ELLKTVSLDE ISLNQLLECP IREKNLPWME NQLNLLLKAF QAIGSDGNEV EYNLREISGL KQRLQFYEK VTKIFLNRIV EEMQKKFSNI RGQDISHDQM IRILTTLLIF SPLILFCKEI SQKSYQAIVE NWNVSIQPVY MELWTKKISQ LQGIDTNDE KMNELSLSQL LNEWDTFRKE RKTNDINPVF KNSFSLLTEC LQTMRQECIV YQNFVEVFFH ISSKHNFEEY I KHFNDPDA PPILLDTVKV MQSDREAAVI ETQLVSRIFQ PIVTRLSSYF VELVKAEPTV APALTFYLEN EIKSLESSNH EF LLSAVTR MYTQIKQVWS DNVEEQVLHF ERISNATTNG EILPGILDLP VGLKNSEDLF QFAKRSMDIK DTDEGYESIE LMN SSFRKL SIAATRSITH KEVNSSINPN LSDTAALNND YMETISLLVN SNWLTEMLSM LNFNKDGIFD TSLQNVKKVF DVEK ESYAS FLLRDTMPKL TAFVYGVSNI IENTNNVNMT NPSRWAAYSR QNLENILLAY TSHEIETLVK RLHTHMVNDF GYHQE NAIN NVLCDKLWSC IQGQTVSLYL KLYTVIDKHY RGTNIRFTKN DIISAFEEYK NA UniProtKB: Exocyst complex component SEC3 |
-分子 #2: Exocyst complex component SEC5
| 分子 | 名称: Exocyst complex component SEC5 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 112.236875 KDa |
| 配列 | 文字列: MDRFQIGDEQ LLRFYQLKTI NPTHSWAQDS SKLNNEEATS NELGVETSFD ILKDFKYGNQ ISIDKESRAY LNDESLSYIR DPLNGQEMS KELQHLPNDS MRLNYLVNSK QFNVKAFLRD MHKQDSFNDL NNSLDRLDSD IQDQSIHLKQ LVGKNFTKYV K IKNKLDQI ...文字列: MDRFQIGDEQ LLRFYQLKTI NPTHSWAQDS SKLNNEEATS NELGVETSFD ILKDFKYGNQ ISIDKESRAY LNDESLSYIR DPLNGQEMS KELQHLPNDS MRLNYLVNSK QFNVKAFLRD MHKQDSFNDL NNSLDRLDSD IQDQSIHLKQ LVGKNFTKYV K IKNKLDQI YKEFDEKTNE KNQCDSPKEN QINVESLNKK VDEVIRTTTF KLKPLMDNYQ KILNYQATKK FIELNKFYFN LP KSLKRCL TNNDFNEFII EYSKGLTLRR RFNQSSDASQ SLVIKRIWTQ IENLLVTYKD LIWNSLINSN FNIDQPQETI LSL FSKLLN LENFINNNQR ESESGNKNTT SSSNENPILR WMSIKMNGFQ NELNELSGHM ISKIIHSQRL ILQNNTNQDK SQGC VELSY YLKINQLFQI ISDTGKDSEG LKSTVEPNKV NTISGTSYLN LNCQPSSQGL TDSPTIIEMW LLILKYINDL WKICD QFIE FWEHIEKFLD GTYQNSIINE KRKENILIGD SNIIESYQKS LILKEEQINE VRLKGEEFIT SVSQNLISFF TSSQSS LPS SLKDSTGDIT RSNKDSGSPL DYGFIPPNCN GLSCLRYLPK IVEPILKFST ELAQLNITTN GITICRNTLS TIINRCV GA ISSTKLRDIS NFYQLENWQV YETVTFSSKS QDSSKNLTFE YGVTQFPEIV TSFQEVSIKT TRDLLFAYEK LPIINGIS V VSYPSKQLLT GIEIQQIISM EAVLEAILKN AAKDKDNPRN SHTILTLTNL QYFRECAFPN ILQYFDDAFE WNLASKNLE LFSLLSKMES SIFGNYLSDL KINLRDTLEE KFHEINWPMY TSNSFRVGDY IIEALMILIV VHSECFRIGP QLIHKILIET QIFIARYLF EAFKPYVGNL SNDGSLQIIV DLEFFQKVMG PLLEKDTEAT LRACLQNCFQ NDTNRLQKCI NEINPIVSAN L KRTAIQFA AFS UniProtKB: Exocyst complex component SEC5 |
-分子 #3: Exocyst complex component SEC6
| 分子 | 名称: Exocyst complex component SEC6 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 93.539703 KDa |
| 配列 | 文字列: MSSDPLQQVC DLIKGDLSLE RVRDIKEQLL KEKSVVEYQL NKESDKYYGE VEESLKLLNL SKNSVTSIKQ QINEVNKLGN DNRFAINRY DILFRATKLY ETVNTTSSIY DRIYNFVALM EHIERLLVAE LAEDALETGC PHLLEIHFLL TSARDFQEQV V VMAKEATE ...文字列: MSSDPLQQVC DLIKGDLSLE RVRDIKEQLL KEKSVVEYQL NKESDKYYGE VEESLKLLNL SKNSVTSIKQ QINEVNKLGN DNRFAINRY DILFRATKLY ETVNTTSSIY DRIYNFVALM EHIERLLVAE LAEDALETGC PHLLEIHFLL TSARDFQEQV V VMAKEATE DAQRTVMKLF SRLSGIISKF DKLLDGLTYD IVEMARAEQI SLAIRLFKIY DLEEREDLRI EAIRNIIKKK EI EIEKSSI KKLPNSKNTA RLQDETPKVI EYPTNKGLYQ EIMSGTISTR TAPRGYKHFL INGINNSISE MFGEMREKYV GDQ KFDVLD NMDWIFNELI IVKEHIANCC PPHWNIFEVY FDQYYKELHS LITDLVESEP ETIIILDILA FDKTFQDTLK QDFG FTKSE VKSVIGDKEK ETLFKDYLNL IVVKMTEWIG NLEKAEFDVF LERSTPPHSD SDGLLFLDGT KTCFQMFTQQ VEVAA GTNQ AKILVGVVER FSDLLTKRQK NWISKISEEI KKQINYNHKY DIDPESITPE DECPGGLVEY LIAVSNDQMK AADYAV AIS SKYGKLVSKV YEKQITNHLE GTLDGFAEVA QCSSLGLITL MFDDLRKPYQ EIFSKTWYMG SQAQQIADTL DEYLLDI KP QMNSVLFVNF IDNVIGETII KFLTALSFEH SFKNKNNKFL EAMKRDFEIF YQLFVKVLDG NESKDTLITQ NFTVMEFF M DLSCEPIDSI LDIWQKYLEV YWDSRIDLLV GILKCRKDVS SSERKKIVQQ ATEMLHEYRR NMEANGVDRE PTLMRRFVL EFEKQ UniProtKB: Exocyst complex component SEC6 |
-分子 #4: Exocyst complex component SEC8
| 分子 | 名称: Exocyst complex component SEC8 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 122.367109 KDa |
| 配列 | 文字列: MDYLKPAQKG RRRGLSINSL SETQQSAMNS SLDHLQNDLN RINLQWNRIL SDNTNPLELA LAFLDDTSVG LGHRYEEFNQ LKSQIGSHL QDVVNEHSQV FNTNVASYGK AVSSIMQAQE QTLNLKNCLK EANEKITTDK GSLQELNDNN LKYTKMIDVL V NIEELLQI ...文字列: MDYLKPAQKG RRRGLSINSL SETQQSAMNS SLDHLQNDLN RINLQWNRIL SDNTNPLELA LAFLDDTSVG LGHRYEEFNQ LKSQIGSHL QDVVNEHSQV FNTNVASYGK AVSSIMQAQE QTLNLKNCLK EANEKITTDK GSLQELNDNN LKYTKMIDVL V NIEELLQI PEKIEENIRK ENFHQVQILL ERGFILMNNK SLKTVEILKP INQQLELQEH LLFNNLIEEI HDIMYSKSNK TN FTRVTNN DIFKIISISH NGFTSLENYL YNIVNIDIME HSKTINKNLE QFIHDQSLNK GNIMLQENAA TQAPLAPSRN QEN EGFNRI GFLLKTINNI NKLPVAFNII TERAKEEIHN IIVKSTESIR SKHPSLLKMA TSLKNDNHFG LPVQDILSII LREC FWEIF LKLLYAIQCH RAIFEMSNIL QPTSSAKPAF KFNKIWGKLL DEIELLLVRY INDPELISSN NGSIKPINGA TNNAP TLPK RKNPKIFSLE YNIEDNSSVK DQAFELKALL KDIFPGFSVS SNMDLDSIYV KDESFEQDEP LVPPSVFNMK VILDPF LLF TQSTSTIVPS VLTQNTISSL TFFDDYMNKS FLPKIQMTMD YLFTVEVESN NPYALELSDE NHNIFKTALD FQRLFYN LL NVFNTANTFR EKISYCILDL LNHFYNYYLG LFNSLIGTSD RHLTRKIITA WLQNGILMDQ EQKILNGDET LFHEESIE L FKEIPHFYQA GKGLSKSDLF NNLTLDTILQ FSASVLWILN WLPGLKKAIN IDEVSQEPML DADRLRSSWT FSESMDLNY SNPSSSPNSL GNLKILLDDK ASKKFDETID GFKTLKFKLI TILRFNIRAL CIYDIGSFFQ NTKIWNMDVG SIELDQNIAS LISELRRTE SKLKQQLPEK EKNSIFIGLD IVNNYALIKG AKSIKVLNHN GIKKMLRNVN VLQHAYRNLS SEPSKINMNV T MNFYSLCG SSEAELFEYI KDNELPHCSV EDLKTILRLQ FSEEMHRQLK RQSTSSTKGS IKPSNKRYTE ALEKLSNLEK EQ SKEGART KIGKLKSKLN AVHTANEK UniProtKB: Exocyst complex component SEC8 |
-分子 #5: Exocyst complex component SEC10
| 分子 | 名称: Exocyst complex component SEC10 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 100.459578 KDa |
| 配列 | 文字列: MNSLYELDPK WKKLLKTDNF LGGLTVNEFV QELSKDHRND VLIDANTKNL PTNEKDQDAI REAIWKQLDP KPYIRTFEST LKELKNLNE ETLNKRQYFS EQVATQEVIH SENVIKLSKD LHTTLLTFDK LDDRLTNVTQ VVSPLGDKLE TAIKKKQNYI Q SVELIRRY ...文字列: MNSLYELDPK WKKLLKTDNF LGGLTVNEFV QELSKDHRND VLIDANTKNL PTNEKDQDAI REAIWKQLDP KPYIRTFEST LKELKNLNE ETLNKRQYFS EQVATQEVIH SENVIKLSKD LHTTLLTFDK LDDRLTNVTQ VVSPLGDKLE TAIKKKQNYI Q SVELIRRY NDFYSMGKSD IVEQLRLSKN WKLNLKSVKL MKNLLILSSK LETSSIPKTI NTKLVIEKYS EMMENELLEN FN SAYRENN FTKLNEIAII LNNFNGGVNV IQSFINQHDY FIDTKQIDLE NEFENVFIKN VKFKEQLIDF ENHSVIIETS MQN LINDVE TVIKNESKIV KRVFEEKATH VIQLFIQRVF AQKIEPRFEV LLRNSLSISN LAYVRILHGL FTLFGKFTKS LIDY FQLLE IDDSNQILST TLEQCFADLF SHYLYDRSKY FGIEKRSLEA ILVDMTSKFT VNYDKEINKR VLLDKYKEKL STNVD AFMH SPRGNTHSRQ DSTSRSKLSQ FNSFLKTHLD KDHLSLNRTN TLSDSFNNSS SSTQYDVANN SSSLVNSSFT ASDIDN SPN SPANYSLNDV DSMLKCVVES TARVMELIPN KAHLYILEIL KIMFLGIVDS YMEIALEVAY WKICKVDINK TAGVVNL NF LKFISMSTEI LDLLSISIKS IFLPLLNNSP EIKAQIIEMT NSQIQKMEIL INIILQETIT VISTKFSAIL CKQKKKDF V PKSQELLDQD TLPAIEIVNI LNLIFEQSSK FLKGKNLQTF LTLIGEELYG LLLSHYSHFQ VNSIGGVVVT KDIIGYQTA IEDWGVASLI DKFATLRELA NLFTVQPELL ESLTKEGHLA DIGRDIIQSY ISNREDFNHD NFINSVKLNF R UniProtKB: Exocyst complex component SEC10 |
-分子 #6: Exocyst complex component SEC15
| 分子 | 名称: Exocyst complex component SEC15 / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 105.166641 KDa |
| 配列 | 文字列: MDQEGQPLLS KDFQQVLLAT ASGNNSSWTE RAVLNNESTD AVKHEPALGQ NDVFDLDPLS FDKWVPFLRR ALDKNQLDPV IDELENSIE DNFQGLELQL LQDSQMNDKL ETSIDEIANI QGMVQDTLSS EISKFQIRLS ESANELIVKK QMYVNNKKIS L KISEATIL ...文字列: MDQEGQPLLS KDFQQVLLAT ASGNNSSWTE RAVLNNESTD AVKHEPALGQ NDVFDLDPLS FDKWVPFLRR ALDKNQLDPV IDELENSIE DNFQGLELQL LQDSQMNDKL ETSIDEIANI QGMVQDTLSS EISKFQIRLS ESANELIVKK QMYVNNKKIS L KISEATIL ITKVVRILEL SSKCQELITE RKFFKVLQNL DSLEKLYLQE FKNYNFQFLI EIYNSIPFLQ KVTKDECINL IR NSLNLNL GKNLIKVGQE FVAIYENELL PQWLETRSKM KLTNFKFNSP IEISMRDESF LAKLNLGEFF QLDDFHDSIM IFQ NLNELS VLSGEFNKEY ELRKTKLMYP LIWKKNKTAA YQMDSLLRGT GTTPGSTAHD VSTDDPFTQS LSLHFLQDYF LKIL GFLLY DINLNKATEF ILVDNNYNST NEFWDGLMDR LSPYLSYFID EKLKTEEDMI KLKDFLCIYV AILENFKLNI EPLYK ILVS IFEKFCSVSL RAFDDEFQIL LNDDDFMPLS INDKTLYEKV LKICWMKEGE HLSLPDPTNG EPFAVTLPFS PLYPMT CTL AKKTYSKITA FLSIFYRHEL HTLNNILVKT MDDIFNDIVN KKIRSKLEST SREEIAQILV NLDYFIIAAK EFSNFMT RE NILQNPDMEI RLSSIKYLAE SRKLAETKLI ELIDSKISDI LETIEIDWQI TEVRQDPDIS IIDLAQFLEM MFASTLQN L PYSVQTLLIF REFDSLTRQF MGLLLHDTPS TITHESIMNF EVDVNYLESI IPRIFPSTPG TIDSNGYQSP MTPSTPTFP NANGVDAPTL FENNIKSLEA TFMELKQCIE LLKTQGKDYN EPEIRLRKYS RIRQEDAALL LSKIQHFVSS VEGANGDDTS VMDSSSIFN SESASVIDSN TSRIAKFFNR R UniProtKB: Exocyst complex component SEC15 |
-分子 #7: Exocyst complex component EXO70
| 分子 | 名称: Exocyst complex component EXO70 / タイプ: protein_or_peptide / ID: 7 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 71.382328 KDa |
| 配列 | 文字列: MPAEIDIDEA DVLVLSQELQ KTSKLTFEIN KSLKKIAATS NQSSQLFTPI LARNNVLTTL QRNIESTLNS VASVKDLANE ASKYEIILQ KGINQVGLKQ YTQVVHKLDD MLEDIQSGQA NREENSEFHG ILTHLEQLIK RSEAQLRVYF ISILNSIKPF D PQINITKK ...文字列: MPAEIDIDEA DVLVLSQELQ KTSKLTFEIN KSLKKIAATS NQSSQLFTPI LARNNVLTTL QRNIESTLNS VASVKDLANE ASKYEIILQ KGINQVGLKQ YTQVVHKLDD MLEDIQSGQA NREENSEFHG ILTHLEQLIK RSEAQLRVYF ISILNSIKPF D PQINITKK MPFPYYEDQQ LGALSWILDY FHGNSEGSII QDILVGERSK LILKCMAFLE PFAKEISTAK NAPYEKGSSG MN SYTEALL GFIANEKSLV DDLYSQYTES KPHVLSQILS PLISAYAKLF GANLKIVRSN LENFGFFSFE LVESINDVKK SLR GKELQN YNLLQDCTQE VRQVTQSLFR DAIDRIIKKA NSISTIPSNN GVTEATVDTM SRLRKFSEYK NGCLGAMDNI TREN WLPSN YKEKEYTLQN EALNWEDHNV LLSCFISDCI DTLAVNLERK AQIALMPNQE PDVANPNSSK NKHKQRIGFF ILMNL TLVE QIVEKSELNL MLAGEGHSRL ERLKKRYISY MVSDWRDLTA NLMDSVFIDS SGKKSKDKEQ IKEKFRKFNE GFEDLV SKT KQYKLSDPSL KVTLKSEIIS LVMPMYERFY SRYKDSFKNP RKHIKYTPDE LTTVLNQLVR UniProtKB: Exocyst complex component EXO70 |
-分子 #8: Exocyst complex component EXO84
| 分子 | 名称: Exocyst complex component EXO84 / タイプ: protein_or_peptide / ID: 8 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 85.649672 KDa |
| 配列 | 文字列: MVEFSLKKAR NNWKHVKKSA SSPAKQKTPP SPAKPKQKTK KNPYSDLKDP ATSYTLPTIN ARERSRVATS MQRRLSIHNT NYAPPTLDY SMPLPDMPNM IVPNDNVDSS HNNSSFTTEN ESVSSKGPSN SLNLSTADLS LNDSSYNKVP ARSAMRNTVN P SGSNDPFN ...文字列: MVEFSLKKAR NNWKHVKKSA SSPAKQKTPP SPAKPKQKTK KNPYSDLKDP ATSYTLPTIN ARERSRVATS MQRRLSIHNT NYAPPTLDY SMPLPDMPNM IVPNDNVDSS HNNSSFTTEN ESVSSKGPSN SLNLSTADLS LNDSSYNKVP ARSAMRNTVN P SGSNDPFN NSTSLRKMLA NPHFNAKDFV HDKLGNASAI TIDKFTSNLT DLSIQVQEEV KLNINKSYNE IMTVNNDLNV AM LELKRVR ANINDLNEVL DQCTKIAEKR LQLQDQIDQE RQGNFNNVES HSNSPALLPP LKAGQNGNLM RRDRSSVLIL EKF WDTELD QLFKNVEGAQ KFINSTKGRH ILMNSANWME LNTTTGKPLQ MVQIFILNDL VLIADKSRDK QNDFIVSQCY PLKD VTVTQ EEFSTKRLLF KFSNSNSSLY ECRDADECSR LLDVIRKAKD DLCDIFHVEE ENSKRIRESF RYLQSTQQTP GRENN RSPN KNKRRSMGGS ITPGRNVTGA MDQYLLQNLT LSMHSRPRSR DMSSTAQRLK FLDEGVEEID IELARLRFES AVETLL DIE SQLEDLSERI SDEELMLLNL ISLKIEQRRE AISSKLSQSI LSSNEIVHLK SGTENMIKLG LPEQALDLFL QNRSNFI QD LILQIGSVDN PTNYLTQLAV IRFQTIKKTV EDFQDIFKEL GAKISSILVD WCSDEVDNHF KLIDKQLLND EMLSPGSI K SSRKQIDGLK AVGLDFVYKL DEFIKKNSDK IR UniProtKB: Exocyst complex component EXO84 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)

















































































 解析
解析 電子顕微鏡法
電子顕微鏡法