+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6374 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | A putative ATPase mediates RNA transcription and capping in a dsRNA virus | |||||||||
 マップデータ マップデータ | Reconstruction of G-CPV | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Viral ATPase / histidine-mediated guanylyl transfer / conformational changes / regulation of transcription | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Bombyx mori cypovirus 1 (ウイルス) Bombyx mori cypovirus 1 (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Yu XK / Jiang JS / Sun JC / Zhou ZH | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2015 ジャーナル: Elife / 年: 2015タイトル: A putative ATPase mediates RNA transcription and capping in a dsRNA virus. 著者: Xuekui Yu / Jiansen Jiang / Jingchen Sun / Z Hong Zhou /  要旨: mRNA transcription in dsRNA viruses is a highly regulated process but the mechanism of this regulation is not known. Here, by nucleoside triphosphatase (NTPase) assay and comparisons of six high- ...mRNA transcription in dsRNA viruses is a highly regulated process but the mechanism of this regulation is not known. Here, by nucleoside triphosphatase (NTPase) assay and comparisons of six high-resolution (2.9-3.1 Å) cryo-electron microscopy structures of cytoplasmic polyhedrosis virus with bound ligands, we show that the large sub-domain of the guanylyltransferase (GTase) domain of the turret protein (TP) also has an ATP-binding site and is likely an ATPase. S-adenosyl-L-methionine (SAM) acts as a signal and binds the methylase-2 domain of TP to induce conformational change of the viral capsid, which in turn activates the putative ATPase. ATP binding/hydrolysis leads to an enlarged capsid for efficient mRNA synthesis, an open GTase domain for His217-mediated guanylyl transfer, and an open methylase-1 domain for SAM binding and methyl transfer. Taken together, our data support a role of the putative ATPase in mediating the activation of mRNA transcription and capping within the confines of the virus. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6374.map.gz emd_6374.map.gz | 422.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6374-v30.xml emd-6374-v30.xml emd-6374.xml emd-6374.xml | 9.6 KB 9.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  400_6374.gif 400_6374.gif 80_6374.gif 80_6374.gif | 74.3 KB 3.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6374 http://ftp.pdbj.org/pub/emdb/structures/EMD-6374 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6374 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6374 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  3jb0MC  6371C  6375C  6376C  6377C  6378C  3jayC  3jazC  3jb1C  3jb2C  3jb3C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6374.map.gz / 形式: CCP4 / 大きさ: 1.2 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6374.map.gz / 形式: CCP4 / 大きさ: 1.2 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of G-CPV | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
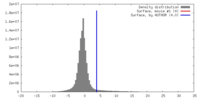
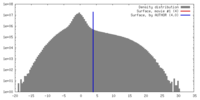
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.104 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cytoplasmic polyhedrosis virus with GTP
| 全体 | 名称: Cytoplasmic polyhedrosis virus with GTP |
|---|---|
| 要素 |
|
-超分子 #1000: Cytoplasmic polyhedrosis virus with GTP
| 超分子 | 名称: Cytoplasmic polyhedrosis virus with GTP / タイプ: sample / ID: 1000 / 集合状態: icosahedral / Number unique components: 1 |
|---|
-超分子 #1: Bombyx mori cypovirus 1
| 超分子 | 名称: Bombyx mori cypovirus 1 / タイプ: virus / ID: 1 / NCBI-ID: 110829 / 生物種: Bombyx mori cypovirus 1 / データベース: NCBI / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
| ウイルス殻 | Shell ID: 1 / 直径: 720 Å / T番号(三角分割数): 1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK II |
|---|
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 135,000 times magnification. |
| 日付 | 2012年4月14日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM デジタル化 - スキャナー: NIKON SUPER COOLSCAN 9000 平均電子線量: 25 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 60535 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.75 mm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Each particle |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 2.9 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: IMIRS / 使用した粒子像数: 71946 |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)