+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6059 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative stain EM structure of the yeast dynein motor domain in the presence of ATP | |||||||||
 マップデータ マップデータ | reconstruction of yeast dynein motor domain in the presence of 5 mM ATP | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | motor proteins / power stroke / dynein / AAA protein / ATPase | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報karyogamy / nuclear migration along microtubule / astral microtubule / establishment of mitotic spindle localization / spindle pole body / minus-end-directed microtubule motor activity / cytoplasmic dynein complex / dynein light intermediate chain binding / nuclear migration / dynein intermediate chain binding ...karyogamy / nuclear migration along microtubule / astral microtubule / establishment of mitotic spindle localization / spindle pole body / minus-end-directed microtubule motor activity / cytoplasmic dynein complex / dynein light intermediate chain binding / nuclear migration / dynein intermediate chain binding / establishment of mitotic spindle orientation / mitotic sister chromatid segregation / cytoplasmic microtubule / cytoplasmic microtubule organization / Neutrophil degranulation / mitotic spindle organization / cell cortex / ATP hydrolysis activity / ATP binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 16.0 Å | |||||||||
 データ登録者 データ登録者 | Bhabha G / Moeller A / Liao M / Speir JA / Vale RD / Cheng Y | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2014 ジャーナル: Cell / 年: 2014タイトル: Allosteric communication in the dynein motor domain. 著者: Gira Bhabha / Hui-Chun Cheng / Nan Zhang / Arne Moeller / Maofu Liao / Jeffrey A Speir / Yifan Cheng / Ronald D Vale /  要旨: Dyneins power microtubule motility using ring-shaped, AAA-containing motor domains. Here, we report X-ray and electron microscopy (EM) structures of yeast dynein bound to different ATP analogs, which ...Dyneins power microtubule motility using ring-shaped, AAA-containing motor domains. Here, we report X-ray and electron microscopy (EM) structures of yeast dynein bound to different ATP analogs, which collectively provide insight into the roles of dynein's two major ATPase sites, AAA1 and AAA3, in the conformational change mechanism. ATP binding to AAA1 triggers a cascade of conformational changes that propagate to all six AAA domains and cause a large movement of the "linker," dynein's mechanical element. In contrast to the role of AAA1 in driving motility, nucleotide transitions in AAA3 gate the transmission of conformational changes between AAA1 and the linker, suggesting that AAA3 acts as a regulatory switch. Further structural and mutational studies also uncover a role for the linker in regulating the catalytic cycle of AAA1. Together, these results reveal how dynein's two major ATP-binding sites initiate and modulate conformational changes in the motor domain during motility. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6059.map.gz emd_6059.map.gz | 740.3 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6059-v30.xml emd-6059-v30.xml emd-6059.xml emd-6059.xml | 9.3 KB 9.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_6059.png emd_6059.png | 155.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6059 http://ftp.pdbj.org/pub/emdb/structures/EMD-6059 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6059 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6059 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6047C  6048C  6049C  6050C  6051C  6052C  6053C  6054C  6055C  6056C  6058C  6060C 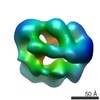 6061C  6062C  6063C  6064C 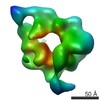 6065C  6066C 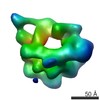 6067C 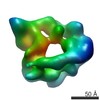 6068C  6069C  6070C  6071C  6072C  6073C  6074C  4w8fC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6059.map.gz / 形式: CCP4 / 大きさ: 1001 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6059.map.gz / 形式: CCP4 / 大きさ: 1001 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | reconstruction of yeast dynein motor domain in the presence of 5 mM ATP | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.26 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : yeast dynein motor domain in the presence of ATP
| 全体 | 名称: yeast dynein motor domain in the presence of ATP |
|---|---|
| 要素 |
|
-超分子 #1000: yeast dynein motor domain in the presence of ATP
| 超分子 | 名称: yeast dynein motor domain in the presence of ATP / タイプ: sample / ID: 1000 / 集合状態: monomer / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 303 KDa |
-分子 #1: dynein
| 分子 | 名称: dynein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 集合状態: monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 303 KDa |
| 組換発現 | 生物種:  |
| 配列 | UniProtKB: Dynein heavy chain, cytoplasmic |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.02 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 50 mM Tris, pH 8.0, 200 mM sodium chloride, 1 mM EGTA, 2 mM magnesium acetate, 1 mM DTT |
| 染色 | タイプ: NEGATIVE 詳細: Samples were applied to freshly glow discharged carbon coated grids and blotted off. Immediately after blotting, a 2% uranyl formate solution was applied for staining and blotted off. The ...詳細: Samples were applied to freshly glow discharged carbon coated grids and blotted off. Immediately after blotting, a 2% uranyl formate solution was applied for staining and blotted off. The stain was applied five times per sample. Samples were allowed to air dry before imaging. |
| グリッド | 詳細: Cu 400 mesh |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 日付 | 2013年9月17日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) 実像数: 611 / 平均電子線量: 54 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 80000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 16.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Relion / 使用した粒子像数: 3319 |
|---|
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















