+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5995 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of beta-galactosidase at 3.2-A resolution obtained by cryo-electron microscopy | |||||||||
 マップデータ マップデータ | Reconstruction of beta-galactosidase | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | atomic resolution cryo-electron microscopy / single-particle EM / direct electron detectors / 3D reconstruction / frame alignment / CTF determination / structure refinement / radiation damage / protein complexes / enzyme active site structure | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報alkali metal ion binding / lactose catabolic process / beta-galactosidase complex / beta-galactosidase / beta-galactosidase activity / carbohydrate binding / magnesium ion binding / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Bartesaghi A / Matthies D / Banerjee S / Merk A / Subramaniam S | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2014 ジャーナル: Proc Natl Acad Sci U S A / 年: 2014タイトル: Structure of β-galactosidase at 3.2-Å resolution obtained by cryo-electron microscopy. 著者: Alberto Bartesaghi / Doreen Matthies / Soojay Banerjee / Alan Merk / Sriram Subramaniam /  要旨: We report the solution structure of Escherichia coli β-galactosidase (∼465 kDa), solved at ∼3.2-Å resolution by using single-particle cryo-electron microscopy (cryo-EM). Densities for most side ...We report the solution structure of Escherichia coli β-galactosidase (∼465 kDa), solved at ∼3.2-Å resolution by using single-particle cryo-electron microscopy (cryo-EM). Densities for most side chains, including those of residues in the active site, and a catalytic Mg(2+) ion can be discerned in the map obtained by cryo-EM. The atomic model derived from our cryo-EM analysis closely matches the 1.7-Å crystal structure with a global rmsd of ∼0.66 Å. There are significant local differences throughout the protein, with clear evidence for conformational changes resulting from contact zones in the crystal lattice. Inspection of the map reveals that although densities for residues with positively charged and neutral side chains are well resolved, systematically weaker densities are observed for residues with negatively charged side chains. We show that the weaker densities for negatively charged residues arise from their greater sensitivity to radiation damage from electron irradiation as determined by comparison of density maps obtained by using electron doses ranging from 10 to 30 e(-)/Å(2). In summary, we establish that it is feasible to use cryo-EM to determine near-atomic resolution structures of protein complexes (<500 kDa) with low symmetry, and that the residue-specific radiation damage that occurs with increasing electron dose can be monitored by using dose fractionation tools available with direct electron detector technology. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5995.map.gz emd_5995.map.gz | 136.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5995-v30.xml emd-5995-v30.xml emd-5995.xml emd-5995.xml | 13.4 KB 13.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_5995_fsc.xml emd_5995_fsc.xml | 13.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  400_5995.gif 400_5995.gif 80_5995.gif 80_5995.gif | 82.5 KB 4.5 KB | ||
| その他 |  emd_5995_additional_1.map.gz emd_5995_additional_1.map.gz emd_5995_half_map_1.map.gz emd_5995_half_map_1.map.gz emd_5995_half_map_2.map.gz emd_5995_half_map_2.map.gz | 59.7 MB 61.2 MB 61.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5995 http://ftp.pdbj.org/pub/emdb/structures/EMD-5995 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5995 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5995 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5995_validation.pdf.gz emd_5995_validation.pdf.gz | 416.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5995_full_validation.pdf.gz emd_5995_full_validation.pdf.gz | 416.2 KB | 表示 | |
| XML形式データ |  emd_5995_validation.xml.gz emd_5995_validation.xml.gz | 12.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5995 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5995 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5995 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5995 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  3j7hMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10012 (タイトル: Structure of β-galactosidase at 3.2-Å resolution obtained by cryo-electron microscopy (frame-averaged micrographs) EMPIAR-10012 (タイトル: Structure of β-galactosidase at 3.2-Å resolution obtained by cryo-electron microscopy (frame-averaged micrographs)Data size: 108.0 Data #1: Beta Galactosidase micrographs [micrographs - single frame])  EMPIAR-10013 (タイトル: Structure of β-galactosidase at 3.2-Å resolution obtained by cryo-electron microscopy EMPIAR-10013 (タイトル: Structure of β-galactosidase at 3.2-Å resolution obtained by cryo-electron microscopyData size: 442.5 Data #1: Full set of multi-frame micrographs [micrographs - multiframe] Data #2: Sample set of multi-frame micrographs [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5995.map.gz / 形式: CCP4 / 大きさ: 146.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5995.map.gz / 形式: CCP4 / 大きさ: 146.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of beta-galactosidase | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.6375 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: emd 5995 additional 1.map
| ファイル | emd_5995_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
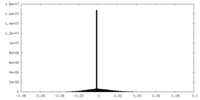
| 密度ヒストグラム |
-添付マップデータ: emd 5995 half map 1.map
| ファイル | emd_5995_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
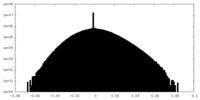
| 密度ヒストグラム |
-添付マップデータ: emd 5995 half map 2.map
| ファイル | emd_5995_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Escherichia coli beta-galactosidase
| 全体 | 名称: Escherichia coli beta-galactosidase |
|---|---|
| 要素 |
|
-超分子 #1000: Escherichia coli beta-galactosidase
| 超分子 | 名称: Escherichia coli beta-galactosidase / タイプ: sample / ID: 1000 / 詳細: The sample was monodisperse. / 集合状態: tetramer / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 465 KDa 手法: ProtParam tool: Gasteiger E., Hoogland C., Gattiker A., Duvaud S., Wilkins M.R., Appel R.D., Bairoch A., Protein Identification and Analysis Tools on the ExPASy Server, (in) John M. Walker ...手法: ProtParam tool: Gasteiger E., Hoogland C., Gattiker A., Duvaud S., Wilkins M.R., Appel R.D., Bairoch A., Protein Identification and Analysis Tools on the ExPASy Server, (in) John M. Walker (ed): The Proteomics Protocols Handbook, Humana Press (2005), pp. 571-607 |
-分子 #1: beta-galactosidase
| 分子 | 名称: beta-galactosidase / タイプ: protein_or_peptide / ID: 1 / Name.synonym: beta-gal, b-gal / コピー数: 1 / 集合状態: tetramer / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 465 KDa |
| 配列 | UniProtKB: Beta-galactosidase / GO: beta-galactosidase activity |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.3 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 25 mM Tris, pH 8.0, 50 mM NaCl, 2 mM MgCl2, 0.5 mM TCEP |
| グリッド | 詳細: 200 mesh Quantifoil R2/2 grids, plasma cleaned |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 90.15 K / 装置: LEICA EM GP / 手法: Blot for 2 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 79.6 K / 最高: 79.8 K / 平均: 79.7 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 105,000 times magnification. Legacy - Electron beam tilt params: 5 |
| 特殊光学系 | エネルギーフィルター - 名称: Gatan, Inc. エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 詳細 | Parallel beam illumination |
| 日付 | 2013年10月31日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GATAN K2 (4k x 4k) / 実像数: 509 / 平均電子線量: 45 e/Å2 詳細: Every image is the average of 38 frames recorded by the direct electron detector. The complete set of electron micrographs used to obtain the density map presented here is available through ...詳細: Every image is the average of 38 frames recorded by the direct electron detector. The complete set of electron micrographs used to obtain the density map presented here is available through the Electron Microscopy Pilot Image ARchive (EMPIAR). |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 105000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダー: Liquid nitrogen cooled 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)