+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5556 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative stain electron microscopy structure of Nup192 | |||||||||
 マップデータ マップデータ | Reconstruction of full-length Nup192 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Nuclear Pore Complex / nuclear envelope / nuclear transport / nucleoporin | |||||||||
| 機能・相同性 | nuclear pore inner ring / nuclear pore organization / structural constituent of nuclear pore / nucleocytoplasmic transport / nuclear pore 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 26.0 Å | |||||||||
 データ登録者 データ登録者 | Sampathkumar P / Kim SJ / Upla P / Rice W / Phillips J / Pieper U / Bonanno JB / Fernandez-Martinez J / Ketaren NE / Matsui T ...Sampathkumar P / Kim SJ / Upla P / Rice W / Phillips J / Pieper U / Bonanno JB / Fernandez-Martinez J / Ketaren NE / Matsui T / Stokes DL / Sauder JM / Burley SK / Sali A / Rout MP / Almo SC | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2013 ジャーナル: Structure / 年: 2013タイトル: Structure, dynamics, evolution, and function of a major scaffold component in the nuclear pore complex. 著者: Parthasarathy Sampathkumar / Seung Joong Kim / Paula Upla / William J Rice / Jeremy Phillips / Benjamin L Timney / Ursula Pieper / Jeffrey B Bonanno / Javier Fernandez-Martinez / Zhanna ...著者: Parthasarathy Sampathkumar / Seung Joong Kim / Paula Upla / William J Rice / Jeremy Phillips / Benjamin L Timney / Ursula Pieper / Jeffrey B Bonanno / Javier Fernandez-Martinez / Zhanna Hakhverdyan / Natalia E Ketaren / Tsutomu Matsui / Thomas M Weiss / David L Stokes / J Michael Sauder / Stephen K Burley / Andrej Sali / Michael P Rout / Steven C Almo /  要旨: The nuclear pore complex, composed of proteins termed nucleoporins (Nups), is responsible for nucleocytoplasmic transport in eukaryotes. Nuclear pore complexes (NPCs) form an annular structure ...The nuclear pore complex, composed of proteins termed nucleoporins (Nups), is responsible for nucleocytoplasmic transport in eukaryotes. Nuclear pore complexes (NPCs) form an annular structure composed of the nuclear ring, cytoplasmic ring, a membrane ring, and two inner rings. Nup192 is a major component of the NPC's inner ring. We report the crystal structure of Saccharomyces cerevisiae Nup192 residues 2-960 [ScNup192(2-960)], which adopts an α-helical fold with three domains (i.e., D1, D2, and D3). Small angle X-ray scattering and electron microscopy (EM) studies reveal that ScNup192(2-960) could undergo long-range transition between "open" and "closed" conformations. We obtained a structural model of full-length ScNup192 based on EM, the structure of ScNup192(2-960), and homology modeling. Evolutionary analyses using the ScNup192(2-960) structure suggest that NPCs and vesicle-coating complexes are descended from a common membrane-coating ancestral complex. We show that suppression of Nup192 expression leads to compromised nuclear transport and hypothesize a role for Nup192 in modulating the permeability of the NPC central channel. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5556.map.gz emd_5556.map.gz | 1.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5556-v30.xml emd-5556-v30.xml emd-5556.xml emd-5556.xml | 15.7 KB 15.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5556_1.png emd_5556_1.png | 30.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5556 http://ftp.pdbj.org/pub/emdb/structures/EMD-5556 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5556 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5556 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5556_validation.pdf.gz emd_5556_validation.pdf.gz | 78.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5556_full_validation.pdf.gz emd_5556_full_validation.pdf.gz | 78 KB | 表示 | |
| XML形式データ |  emd_5556_validation.xml.gz emd_5556_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5556 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5556 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5556 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5556 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5556.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5556.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of full-length Nup192 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.93 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
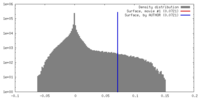
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Full-length Nup192
| 全体 | 名称: Full-length Nup192 |
|---|---|
| 要素 |
|
-超分子 #1000: Full-length Nup192
| 超分子 | 名称: Full-length Nup192 / タイプ: sample / ID: 1000 詳細: The sample was purified using affinity tags then centrifuged on a sucrose gradient. Fractions identified by SDS-PAGE and mass spectrometry. 集合状態: monomer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 192 KDa / 理論値: 192 KDa / 手法: SDS-PAGE |
-分子 #1: Subunit of the Yeast Nuclear Pore Complex inner ring with weight ...
| 分子 | 名称: Subunit of the Yeast Nuclear Pore Complex inner ring with weight 192 kDa タイプ: protein_or_peptide / ID: 1 / Name.synonym: ScNup192 / コピー数: 1 / 集合状態: monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 192 KDa / 理論値: 192 KDa |
| 組換発現 | 生物種:  |
| 配列 | GO: structural constituent of nuclear pore, nuclear pore organization, nucleocytoplasmic transport, nuclear pore, nuclear pore inner ring |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.0035 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 詳細: 20 mM HEPES, 300 mM NaCl, 2 mM MgCl2, 0.01% Tween 20, 0.01 mM DTT |
| 染色 | タイプ: NEGATIVE 詳細: 3 ul protein was added to grids. The sample was blotted and 3 uL 1% uranyl formate added. Uranyl formate was blotted and fresh stain added (three times). The sample was allowed to sit 30 ...詳細: 3 ul protein was added to grids. The sample was blotted and 3 uL 1% uranyl formate added. Uranyl formate was blotted and fresh stain added (three times). The sample was allowed to sit 30 seconds before final blot, then air-dried. |
| グリッド | 詳細: 200 mesh copper grid with thin carbon support, glow discharged 20s with Gatan Solarus 20s in H2/O2. |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法 #1
電子顕微鏡法 #1
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | JEOL 2100F |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 250,000 times magnification |
| 詳細 | Collected on 2028x2048 CCD, 24 um/pixel |
| 日付 | 2012年5月30日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F224 (2k x 2k) デジタル化 - サンプリング間隔: 24 µm / 実像数: 400 / 平均電子線量: 20 e/Å2 / ビット/ピクセル: 16 |
| Tilt angle min | 0 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 81911 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: High tilt / 試料ホルダーモデル: JEOL / Tilt angle max: 50 |
- 電子顕微鏡法 #2
電子顕微鏡法 #2
| Microscopy ID | 2 |
|---|---|
| 顕微鏡 | JEOL 2100F |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 250,000 times magnification |
| 詳細 | Collected on 2028x2048 CCD, 24 um/pixel. 2 sessions are listed; data collected on multiple days between these dates |
| 日付 | 2012年3月1日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F224 (2k x 2k) デジタル化 - サンプリング間隔: 24 µm / 実像数: 400 / 平均電子線量: 20 e/Å2 / ビット/ピクセル: 16 |
| Tilt angle min | 0 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 81911 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: High tilt / 試料ホルダーモデル: JEOL / Tilt angle max: 50 |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Protocol: Rigid Body. The domains were separately fitted by manual docking followed by "Fit in Map"using Chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Cross-correlation 0.754 |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)