+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5538 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM Structure of human defensin 5 bound to a neutralization sensitive adenovirus chimera | |||||||||
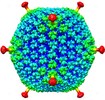
 マップデータ マップデータ | 3D reconstruction of defensin complexed with a neutralization sensitive adenovirus chimera | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | adenovirus / human defensin 5 / neutralization / cryoEM / MDFF / innate immunity | |||||||||
| 生物種 |   Human adenovirus 5 (ヒトアデノウイルス) Human adenovirus 5 (ヒトアデノウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 9.6 Å | |||||||||
 データ登録者 データ登録者 | Flatt JW / Smith JG / Nemerow GR / Stewart PL | |||||||||
 引用 引用 |  ジャーナル: PLoS One / 年: 2013 ジャーナル: PLoS One / 年: 2013タイトル: An intrinsically disordered region of the adenovirus capsid is implicated in neutralization by human alpha defensin 5. 著者: Justin W Flatt / Robert Kim / Jason G Smith / Glen R Nemerow / Phoebe L Stewart /  要旨: Human α-defensins are proteins of the innate immune system that suppress viral and bacterial infections by multiple mechanisms including membrane disruption. For viruses that lack envelopes, such as ...Human α-defensins are proteins of the innate immune system that suppress viral and bacterial infections by multiple mechanisms including membrane disruption. For viruses that lack envelopes, such as human adenovirus (HAdV), other, less well defined, mechanisms must be involved. A previous structural study on the interaction of an α-defensin, human α-defensin 5 (HD5), with HAdV led to a proposed mechanism in which HD5 stabilizes the vertex region of the capsid and blocks uncoating steps required for infectivity. Studies with virus chimeras comprised of capsid proteins from sensitive and resistant serotypes supported this model. To further characterize the critical binding site, we determined subnanometer resolution cryo-electron microscopy (cryoEM) structures of HD5 complexed with both neutralization-sensitive and -resistant HAdV chimeras. Models were built for the vertex regions of these chimeras with monomeric and dimeric forms of HD5 in various initial orientations. CryoEM guided molecular dynamics flexible fitting (MDFF) was used to restrain the majority of the vertex model in well-defined cryoEM density. The RGD-containing penton base loops of both the sensitive and resistant virus chimeras are predicted to be intrinsically disordered, and little cryoEM density is observed for them. In simulations these loops from the sensitive virus chimera, interact with HD5, bridge the penton base and fiber proteins, and provides significant stabilization with a three-fold increase in the intermolecular nonbonded interactions of the vertex complex. In the case of the resistant virus chimera, simulations revealed fewer bridging interactions and reduced stabilization by HD5. This study implicates a key dynamic region in mediating a stabilizing interaction between a viral capsid and a protein of the innate immune system with potent anti-viral activity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5538.map.gz emd_5538.map.gz | 1.3 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5538-v30.xml emd-5538-v30.xml emd-5538.xml emd-5538.xml | 10.5 KB 10.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5538.jpg emd_5538.jpg | 563.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5538 http://ftp.pdbj.org/pub/emdb/structures/EMD-5538 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5538 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5538 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5538_validation.pdf.gz emd_5538_validation.pdf.gz | 78.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5538_full_validation.pdf.gz emd_5538_full_validation.pdf.gz | 77.6 KB | 表示 | |
| XML形式データ |  emd_5538_validation.xml.gz emd_5538_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5538 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5538 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5538 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5538 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5538.map.gz / 形式: CCP4 / 大きさ: 3.2 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5538.map.gz / 形式: CCP4 / 大きさ: 3.2 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D reconstruction of defensin complexed with a neutralization sensitive adenovirus chimera | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
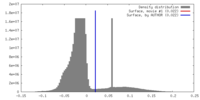
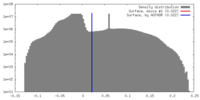
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Complex of an adenovirus chimera Ad5.F35 with human alpha defensin 5
| 全体 | 名称: Complex of an adenovirus chimera Ad5.F35 with human alpha defensin 5 |
|---|---|
| 要素 |
|
-超分子 #1000: Complex of an adenovirus chimera Ad5.F35 with human alpha defensin 5
| 超分子 | 名称: Complex of an adenovirus chimera Ad5.F35 with human alpha defensin 5 タイプ: sample / ID: 1000 / 集合状態: ~3000 molecules bind each adenovirus particle / Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 150 MDa |
-超分子 #1: Human adenovirus 5
| 超分子 | 名称: Human adenovirus 5 / タイプ: virus / ID: 1 / Name.synonym: Human adenovirus type 5 with short fiber / NCBI-ID: 28285 / 生物種: Human adenovirus 5 / データベース: NCBI / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No / Syn species name: Human adenovirus type 5 with short fiber |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) / 別称: VERTEBRATES Homo sapiens (ヒト) / 別称: VERTEBRATES |
| 分子量 | 理論値: 150 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: Capsid / 直径: 1170 Å / T番号(三角分割数): 25 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.16 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 / 詳細: 50mM Tris, 150mM NaCl, 2mM CaCl2, 2mM MgCl2 |
| グリッド | 詳細: Quantifoil R2/4 holey carbon grids, glow discharged |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 30 % / チャンバー内温度: 90 K / 装置: HOMEMADE PLUNGER / 手法: Blot for 4 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 300,000 times magnification Legacy - Electron beam tilt params: 0 |
| 詳細 | Low dose |
| 日付 | 2010年7月12日 |
| 撮影 | カテゴリ: FILM フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) デジタル化 - スキャナー: OTHER / デジタル化 - サンプリング間隔: 15 µm / 実像数: 3515 / 平均電子線量: 20 e/Å2 / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 400000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.26 mm / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 310000 |
| 試料ステージ | 試料ホルダーモデル: OTHER |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | The particles were selected with an in-house script and processed using Frealign |
|---|---|
| CTF補正 | 詳細: Each particle |
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 9.6 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Frealign / 使用した粒子像数: 1014 |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)