+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5024 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Pyrococcus furiosus in apo-state | |||||||||
 マップデータ マップデータ | Archaea RNAP in its (transcription) elongation state. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | transcription / initiation / elongation / RNAP / Archaea / cryo-electron microscopy / negative staining / molecular fitting | |||||||||
| 生物種 | unidentified (未定義) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 18.0 Å | |||||||||
 データ登録者 データ登録者 | Carlo SD / Lin S-C / Taatjes D / Hoenger A | |||||||||
 引用 引用 |  ジャーナル: Transcription / 年: 2010 ジャーナル: Transcription / 年: 2010タイトル: Molecular basis of transcription initiation in Archaea. 著者: Sacha De Carlo / Shih-Chieh Lin / Dylan J Taatjes / Andreas Hoenger /  要旨: Compared with eukaryotes, the archaeal transcription initiation machinery-commonly known as the Pre-Initiation Complex-is relatively simple. The archaeal PIC consists of the TFIIB ortholog TFB, TBP, ...Compared with eukaryotes, the archaeal transcription initiation machinery-commonly known as the Pre-Initiation Complex-is relatively simple. The archaeal PIC consists of the TFIIB ortholog TFB, TBP, and an 11-subunit RNA polymerase (RNAP). The relatively small size of the entire archaeal PIC makes it amenable to structural analysis. Using purified RNAP, TFB, and TBP from the thermophile Pyrococcus furiosus, we assembled the biochemically active PIC at 65ºC. The intact archaeal PIC was isolated by implementing a cross-linking technique followed by size-exclusion chromatography, and the structure of this 440 kDa assembly was determined using electron microscopy and single-particle reconstruction techniques. Combining difference maps with crystal structure docking of various sub-domains, TBP and TFB were localized within the macromolecular PIC. TBP/TFB assemble near the large RpoB subunit and the RpoD/L "foot" domain behind the RNAP central cleft. This location mimics that of yeast TBP and TFIIB in complex with yeast RNAP II. Collectively, these results define the structural organization of the archaeal transcription machinery and suggest a conserved core PIC architecture. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5024.map.gz emd_5024.map.gz | 28.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5024-v30.xml emd-5024-v30.xml emd-5024.xml emd-5024.xml | 9.2 KB 9.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5024_1.jpg emd_5024_1.jpg | 64.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5024 http://ftp.pdbj.org/pub/emdb/structures/EMD-5024 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5024 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5024 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5024.map.gz / 形式: CCP4 / 大きさ: 29.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5024.map.gz / 形式: CCP4 / 大きさ: 29.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Archaea RNAP in its (transcription) elongation state. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.25 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
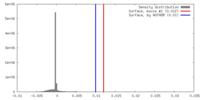
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Pyrococcus furiosus RNAP
| 全体 | 名称: Pyrococcus furiosus RNAP |
|---|---|
| 要素 |
|
-超分子 #1000: Pyrococcus furiosus RNAP
| 超分子 | 名称: Pyrococcus furiosus RNAP / タイプ: sample / ID: 1000 / 集合状態: RNAP and DNA / Number unique components: 2 |
|---|---|
| 分子量 | 実験値: 380 KDa / 手法: Mass spectrometry |
-分子 #1: RNAP apo-state
| 分子 | 名称: RNAP apo-state / タイプ: protein_or_peptide / ID: 1 / Name.synonym: RNAP elongation complex / コピー数: 1 / 集合状態: multi-subunit complex / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) / 細胞: Pyrococcus |
| 分子量 | 実験値: 380 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | 詳細: Hepes 0.1 M, pH 8.0, NaCl 50 mM, EDTA 0.1mM, DTT 2 mM |
| グリッド | 詳細: C-Flat |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 65 % / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM デジタル化 - スキャナー: NIKON SUPER COOLSCAN 9000 デジタル化 - サンプリング間隔: 6.7 µm / 実像数: 32 / 平均電子線量: 15 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 49785 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: 626 / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: phase-flipped |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 18.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: SPIDER, EMAN / 使用した粒子像数: 4611 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称: NORMA |
| 詳細 | Protocol: Rigid Body. Manual fitting with CHIMERA first. |
| 精密化 | 空間: RECIPROCAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)