登録情報 データベース : EMDB / ID : EMD-45729タイトル Cryo-EM model derived from localized reconstruction of human adenovirus 6 (Ad6)-hexon-FII complex 複合体 : Human adenovirus 6 (Ad6) in complex with Coagulation factor FII複合体 : Hexon protein細胞器官・細胞要素 : Human Coagulation Factor IIタンパク質・ペプチド : Hexon proteinリガンド : CALCIUM ION / / / / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 / homo sapiens (ヒト) / Homo sapiens (ヒト)手法 / / 解像度 : 3.7 Å Reddy VS / Ma OX 資金援助 Organization Grant number 国 National Institutes of Health/National Institute Of Allergy and Infectious Diseases (NIH/NIAID) AI161367
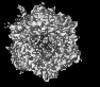
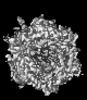
ジャーナル : Nat Commun / 年 : 2024タイトル : Structure-derived insights from blood factors binding to the surfaces of different adenoviruses.著者 : Haley E Mudrick / Shao-Chia Lu / Janarjan Bhandari / Mary E Barry / Jack R Hemsath / Felix G M Andres / Olivia X Ma / Michael A Barry / Vijay S Reddy / 要旨 : The tropism of adenoviruses (Ads) is significantly influenced by the binding of various blood factors. To investigate differences in their binding, we conducted cryo-EM analysis on complexes of ... The tropism of adenoviruses (Ads) is significantly influenced by the binding of various blood factors. To investigate differences in their binding, we conducted cryo-EM analysis on complexes of several human adenoviruses with human platelet factor-4 (PF4), coagulation factors FII (Prothrombin), and FX. While we observed EM densities for FII and FX bound to all the species-C adenoviruses examined, no densities were seen for PF4, even though PF4 can co-pellet with various Ads. Similar to FX, the γ-carboxyglutamic acid (Gla) domain of FII binds within the surface cavity of hexon trimers. While FII binds equally to species-C Ads: Ad5, Ad6, and Ad657, FX exhibits significantly better binding to Ad5 and Ad657 compared to Ad6. Although only the FX-Gla domain is observed at high-resolution (3.7 Å), the entire FX is visible at low-resolution bound to Ad5 in three equivalent binding modes consistent with the 3-fold symmetric hexon. Only the Gla and kringle-1 domains of FII are visible on all the species-C adenoviruses, where the rigid FII binds in an upright fashion, in contrast to the flexible and bent FX. These data suggest that differential binding of FII and FX may shield certain species-C adenoviruses differently against immune molecules, thereby modulating their tropism. 履歴 登録 2024年7月12日 - ヘッダ(付随情報) 公開 2024年11月27日 - マップ公開 2024年11月27日 - 更新 2024年11月27日 - 現状 2024年11月27日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報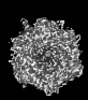
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Human adenovirus 6 (ヒトアデノウイルス) /
Human adenovirus 6 (ヒトアデノウイルス) /  homo sapiens (ヒト) /
homo sapiens (ヒト) /  Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Nat Commun / 年: 2024
ジャーナル: Nat Commun / 年: 2024
 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_45729.map.gz
emd_45729.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-45729-v30.xml
emd-45729-v30.xml emd-45729.xml
emd-45729.xml EMDBヘッダ
EMDBヘッダ emd_45729_fsc.xml
emd_45729_fsc.xml FSCデータファイル
FSCデータファイル emd_45729.png
emd_45729.png emd-45729.cif.gz
emd-45729.cif.gz emd_45729_half_map_1.map.gz
emd_45729_half_map_1.map.gz emd_45729_half_map_2.map.gz
emd_45729_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-45729
http://ftp.pdbj.org/pub/emdb/structures/EMD-45729 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45729
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45729 emd_45729_validation.pdf.gz
emd_45729_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_45729_full_validation.pdf.gz
emd_45729_full_validation.pdf.gz emd_45729_validation.xml.gz
emd_45729_validation.xml.gz emd_45729_validation.cif.gz
emd_45729_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45729
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45729 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45729
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45729 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_45729.map.gz / 形式: CCP4 / 大きさ: 4.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_45729.map.gz / 形式: CCP4 / 大きさ: 4.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 Human adenovirus 6 (ヒトアデノウイルス)
Human adenovirus 6 (ヒトアデノウイルス) Human adenovirus 6 (ヒトアデノウイルス)
Human adenovirus 6 (ヒトアデノウイルス) homo sapiens (ヒト)
homo sapiens (ヒト) Human adenovirus 6 (ヒトアデノウイルス)
Human adenovirus 6 (ヒトアデノウイルス) Homo sapiens (ヒト)
Homo sapiens (ヒト) 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー





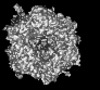
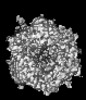
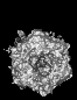























 Y (Sec.)
Y (Sec.) X (Row.)
X (Row.) Z (Col.)
Z (Col.)






































