+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | RuvBL core from SWR1-DNA complex (focused refinement) | ||||||||||||
 マップデータ マップデータ | Focused refinement of RUVBL core from SWR1-DNA structure | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Chromatin Remodeler / Snf2 family ATPase / histone exchange / H2A.Z / GENE REGULATION | ||||||||||||
| 生物種 |  | ||||||||||||
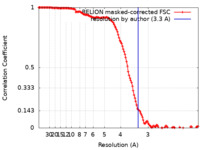
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | ||||||||||||
 データ登録者 データ登録者 | Louder RK / Park G / Wu C | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Cell(Cambridge,Mass.) / 年: 2024 ジャーナル: Cell(Cambridge,Mass.) / 年: 2024タイトル: Molecular basis of global promoter sensing and nucleosome capture by the SWR1 chromatin remodeler 著者: Louder RK / Park G / Ye Z / Cha JS / Gardner AM / Lei Q / Ranjan A / Hollmuller E / Stengel F / Pugh BF / Wu C | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_44107.map.gz emd_44107.map.gz | 15 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-44107-v30.xml emd-44107-v30.xml emd-44107.xml emd-44107.xml | 20.9 KB 20.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_44107_fsc.xml emd_44107_fsc.xml | 13.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_44107.png emd_44107.png | 119.3 KB | ||
| マスクデータ |  emd_44107_msk_1.map emd_44107_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-44107.cif.gz emd-44107.cif.gz | 4.9 KB | ||
| その他 |  emd_44107_additional_1.map.gz emd_44107_additional_1.map.gz emd_44107_half_map_1.map.gz emd_44107_half_map_1.map.gz emd_44107_half_map_2.map.gz emd_44107_half_map_2.map.gz | 123.5 MB 171.4 MB 171 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-44107 http://ftp.pdbj.org/pub/emdb/structures/EMD-44107 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44107 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44107 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_44107_validation.pdf.gz emd_44107_validation.pdf.gz | 849.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_44107_full_validation.pdf.gz emd_44107_full_validation.pdf.gz | 848.8 KB | 表示 | |
| XML形式データ |  emd_44107_validation.xml.gz emd_44107_validation.xml.gz | 21 KB | 表示 | |
| CIF形式データ |  emd_44107_validation.cif.gz emd_44107_validation.cif.gz | 27.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44107 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44107 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44107 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44107 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_44107.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_44107.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused refinement of RUVBL core from SWR1-DNA structure | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.03 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
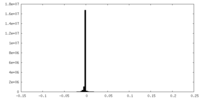
| ファイル |  emd_44107_msk_1.map emd_44107_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Refined map filtered by local resolution (unmasked)
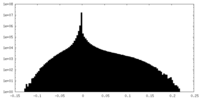
| ファイル | emd_44107_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Refined map filtered by local resolution (unmasked) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
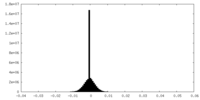
| ファイル | emd_44107_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
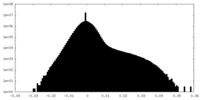
| ファイル | emd_44107_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Native SWR1 bound to DNA.
| 全体 | 名称: Native SWR1 bound to DNA. |
|---|---|
| 要素 |
|
-超分子 #1: Native SWR1 bound to DNA.
| 超分子 | 名称: Native SWR1 bound to DNA. / タイプ: complex / ID: 1 / 親要素: 0 詳細: Endogenously purified yeast SWR1 complex bound to 147-bp dsDNA fragment in the presence of ATPgS. |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 1.19 MDa |
-超分子 #2: Native SWR1 complex
| 超分子 | 名称: Native SWR1 complex / タイプ: complex / ID: 2 / 親要素: 1 / 詳細: Endogenously purified yeast SWR1 complex |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.125 mg/mL | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.6 構成要素:
詳細: 20 mM HEPES pH 7.6, 0.2 mM EDTA, 2 mM MgCl2, 100 mM NaCl, 0.01% IGEPAL CA-630, 3.5% glycerol, and 0.25 mM TCEP, 1 mM ATP-gamma-s, 0.05% glutaraldehyde. | |||||||||||||||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 90 sec. / 前処理 - 雰囲気: AIR | |||||||||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: 6 second blot time and blot force of 12.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 5379 / 平均露光時間: 4.0 sec. / 平均電子線量: 54.0 e/Å2 詳細: Each micrograph was fractionated into 64 frames within a 4 second exposure. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 48543 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.6 µm / 最小 デフォーカス(公称値): 2.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)