+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4341 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of the MDA5-dsRNA filament with 91-degree helical twist | |||||||||
 マップデータ マップデータ | Helical reconstruction of the MDA5-dRNA filament with 91 degree twist at 3.9 A overall resolution | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Protein-RNA complex / helical filament / ATPase / innate immune receptor / IMMUNE SYSTEM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報MDA-5 signaling pathway / Ub-specific processing proteases / positive regulation of response to cytokine stimulus / type I interferon-mediated signaling pathway / negative regulation of viral genome replication / pattern recognition receptor activity / cellular response to exogenous dsRNA / protein complex oligomerization / positive regulation of interferon-alpha production / protein sumoylation ...MDA-5 signaling pathway / Ub-specific processing proteases / positive regulation of response to cytokine stimulus / type I interferon-mediated signaling pathway / negative regulation of viral genome replication / pattern recognition receptor activity / cellular response to exogenous dsRNA / protein complex oligomerization / positive regulation of interferon-alpha production / protein sumoylation / ribonucleoprotein complex binding / antiviral innate immune response / positive regulation of interferon-beta production / response to virus / cellular response to virus / positive regulation of interleukin-6 production / positive regulation of tumor necrosis factor production / double-stranded RNA binding / defense response to virus / single-stranded RNA binding / RNA helicase activity / RNA helicase / protein domain specific binding / innate immune response / ATP hydrolysis activity / mitochondrion / DNA binding / zinc ion binding / ATP binding / identical protein binding / nucleus / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   Pseudomonas phage phi6 (ファージ) Pseudomonas phage phi6 (ファージ) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.93 Å | |||||||||
 データ登録者 データ登録者 | Yu Q / Qu K | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2018 ジャーナル: Mol Cell / 年: 2018タイトル: Cryo-EM Structures of MDA5-dsRNA Filaments at Different Stages of ATP Hydrolysis. 著者: Qin Yu / Kun Qu / Yorgo Modis /  要旨: Double-stranded RNA (dsRNA) is a potent proinflammatory signature of viral infection. Long cytosolic dsRNA is recognized by MDA5. The cooperative assembly of MDA5 into helical filaments on dsRNA ...Double-stranded RNA (dsRNA) is a potent proinflammatory signature of viral infection. Long cytosolic dsRNA is recognized by MDA5. The cooperative assembly of MDA5 into helical filaments on dsRNA nucleates the assembly of a multiprotein type I interferon signaling platform. Here, we determined cryoelectron microscopy (cryo-EM) structures of MDA5-dsRNA filaments with different helical twists and bound nucleotide analogs at resolutions sufficient to build and refine atomic models. The structures identify the filament-forming interfaces, which encode the dsRNA binding cooperativity and length specificity of MDA5. The predominantly hydrophobic interface contacts confer flexibility, reflected in the variable helical twist within filaments. Mutation of filament-forming residues can result in loss or gain of signaling activity. Each MDA5 molecule spans 14 or 15 RNA base pairs, depending on the twist. Variations in twist also correlate with variations in the occupancy and type of nucleotide in the active site, providing insights on how ATP hydrolysis contributes to MDA5-dsRNA recognition. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4341.map.gz emd_4341.map.gz | 4.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4341-v30.xml emd-4341-v30.xml emd-4341.xml emd-4341.xml | 22.6 KB 22.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4341_fsc.xml emd_4341_fsc.xml | 9.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4341.png emd_4341.png | 217.1 KB | ||
| マスクデータ |  emd_4341_msk_1.map emd_4341_msk_1.map | 42.9 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-4341.cif.gz emd-4341.cif.gz | 7.2 KB | ||
| その他 |  emd_4341_half_map_1.map.gz emd_4341_half_map_1.map.gz emd_4341_half_map_2.map.gz emd_4341_half_map_2.map.gz | 12.8 MB 12.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4341 http://ftp.pdbj.org/pub/emdb/structures/EMD-4341 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4341 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4341 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6g1xMC  0012C  0023C  0024C  0143C  0145C  4338C  4340C  6g19C  6g1sC  6gjzC  6gkhC  6gkmC  6h61C  6h66C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10213 (タイトル: mouse MDA5-dsRNA Filaments in presence of 1mM AMPPNP EMPIAR-10213 (タイトル: mouse MDA5-dsRNA Filaments in presence of 1mM AMPPNPData size: 3.5 TB Data #1: mouse MDA5 and bacteriophage dsRNA filaments in complex with 1mM AMPPNP_data1 [micrographs - multiframe] Data #2: Unaligned multi-frame movies of mouse MDA5-dsRNA filaments in complex with 1mM AMPPNP_data2 [micrographs - multiframe] Data #3: Unaligned multi-frame movies of mouse MDA5-dsRNA filaments in complex with 1mM AMPPNP_data3 [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4341.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4341.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Helical reconstruction of the MDA5-dRNA filament with 91 degree twist at 3.9 A overall resolution | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
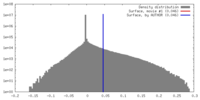
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_4341_msk_1.map emd_4341_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1 used in helical reconstruction in RELION 2.1.0
| ファイル | emd_4341_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 used in helical reconstruction in RELION 2.1.0 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2 used in helical reconstruction in RELION 2.1.0
| ファイル | emd_4341_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 used in helical reconstruction in RELION 2.1.0 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : MDA5-dsRNA helical filament
| 全体 | 名称: MDA5-dsRNA helical filament |
|---|---|
| 要素 |
|
-超分子 #1: MDA5-dsRNA helical filament
| 超分子 | 名称: MDA5-dsRNA helical filament / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 分子量 | 理論値: 2.22 kDa/nm |
-超分子 #2: MDA5
| 超分子 | 名称: MDA5 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 詳細: DExD/H-box helicase consisting of Hel1, Hel2, Hel2i, and pincer domains, followed by a C-terminal domain |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: Double-stranded RNA from bacteriophage Phi6
| 超分子 | 名称: Double-stranded RNA from bacteriophage Phi6 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage phi6 (ファージ) Pseudomonas phage phi6 (ファージ) |
-分子 #1: Interferon-induced helicase C domain-containing protein 1
| 分子 | 名称: Interferon-induced helicase C domain-containing protein 1 タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: RNA helicase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 80.057695 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: LQLRPYQMEV AQPALDGKNI IICLPTGSGK TRVAVYITKD HLDKKKQASE SGKVIVLVNK VMLAEQLFRK EFNPYLKKWY RIIGLSGDT QLKISFPEVV KSYDVIISTA QILENSLLNL ESGDDDGVQL SDFSLIIIDE CHHTNKEAVY NNIMRRYLKQ K LRNNDLKK ...文字列: LQLRPYQMEV AQPALDGKNI IICLPTGSGK TRVAVYITKD HLDKKKQASE SGKVIVLVNK VMLAEQLFRK EFNPYLKKWY RIIGLSGDT QLKISFPEVV KSYDVIISTA QILENSLLNL ESGDDDGVQL SDFSLIIIDE CHHTNKEAVY NNIMRRYLKQ K LRNNDLKK QNKPAIPLPQ ILGLTASPGV GAAKKQSEAE KHILNICANL DAFTIKTVKE NLGQLKHQIK EPCKKFVIAD DT RENPFKE KLLEIMASIQ TYCQKSPMSD FGTQHYEQWA IQMEKKAAKD GNRKDRVCAE HLRKYNEALQ INDTIRMIDA YSH LETFYT DEKEKKFAVL NDSKKSLKLD ETDEFLMNLF FDNKKMLKKL AENPKYENEK LIKLRNTILE QFTRSEESSR GIIF TKTRQ STYALSQWIM ENAKFAEVGV KAHHLIGAGH SSEVKPMTQT EQKEVISKFR TGEINLLIAT TVAEEGLDIK ECNIV IRYG LVTNEIAMVQ ARGRARADES TYVLVTSSGS GVTEREIVND FREKMMYKAI NRVQNMKPEE YAHKILELQV QSILEK KMK VKRSIAKQYN DNPSLITLLC KNCSMLVCSG ENIHVIEKMH HVNMTPEFKG LYIVRENKAL QKKFADYQTN GEIICKC GQ AWGTMMVHKG LDLPCLKIRN FVVNFKNNSP KKQYKKWVEL PIRFPDLDYS EYCL UniProtKB: Interferon-induced helicase C domain-containing protein 1 |
-分子 #2: RNA (5'-R(P*UP*CP*CP*AP*UP*GP*CP*GP*CP*AP*UP*GP*AP*CP*G)-3')
| 分子 | 名称: RNA (5'-R(P*UP*CP*CP*AP*UP*GP*CP*GP*CP*AP*UP*GP*AP*CP*G)-3') タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage phi6 (ファージ) Pseudomonas phage phi6 (ファージ) |
| 分子量 | 理論値: 4.767889 KDa |
| 配列 | 文字列: UCCAUGCGCA UGACG |
-分子 #3: RNA (5'-R(P*CP*GP*UP*CP*AP*UP*GP*CP*GP*CP*AP*UP*GP*GP*A)-3')
| 分子 | 名称: RNA (5'-R(P*CP*GP*UP*CP*AP*UP*GP*CP*GP*CP*AP*UP*GP*GP*A)-3') タイプ: rna / ID: 3 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage phi6 (ファージ) Pseudomonas phage phi6 (ファージ) |
| 分子量 | 理論値: 4.807914 KDa |
| 配列 | 文字列: CGUCAUGCGC AUGGA |
-分子 #4: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 4 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.7 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 詳細: 25 mA | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV | |||||||||||||||
| 詳細 | Filament samples were diluted twofold from 0.8 mg/ml to 0.4 mg/ml immediately prior to plunge freezing |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 3 / 実像数: 1563 / 平均露光時間: 0.8 sec. / 平均電子線量: 29.85 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 75000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 1.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 165 当てはまり具合の基準: Cross-correlation coefficient |
|---|---|
| 得られたモデル |  PDB-6g1x: |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)