+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 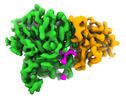 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
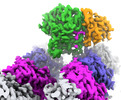
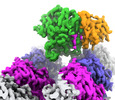
| タイトル | Structure of CCP5 class2 | |||||||||
 マップデータ マップデータ | CCP5 class#2 focused map, b-factor sharpened | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | carboxypeptidase deglutamylation branch glutamate removal microtubule / HYDROLASE / HYDROLASE-SUBSTRATE complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報tubulin-glutamate carboxypeptidase / protein deglutamylation / protein side chain deglutamylation / protein branching point deglutamylation / C-terminal protein deglutamylation / Carboxyterminal post-translational modifications of tubulin / intercellular bridge / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; 金属プロテアーゼ / metallocarboxypeptidase activity / tubulin binding ...tubulin-glutamate carboxypeptidase / protein deglutamylation / protein side chain deglutamylation / protein branching point deglutamylation / C-terminal protein deglutamylation / Carboxyterminal post-translational modifications of tubulin / intercellular bridge / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; 金属プロテアーゼ / metallocarboxypeptidase activity / tubulin binding / mitotic spindle / microtubule cytoskeleton / midbody / defense response to virus / proteolysis / zinc ion binding / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
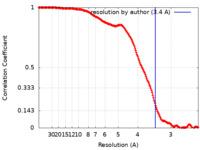
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Chen J / Zehr EA / Gruschus JM / Szyk A / Liu Y / Tanner ME / Tjandra N / Roll-Mecak A | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Tubulin code eraser CCP5 binds branch glutamates by substrate deformation. 著者: Jiayi Chen / Elena A Zehr / James M Gruschus / Agnieszka Szyk / Yanjie Liu / Martin E Tanner / Nico Tjandra / Antonina Roll-Mecak /   要旨: Microtubule function is modulated by the tubulin code, diverse posttranslational modifications that are altered dynamically by writer and eraser enzymes. Glutamylation-the addition of branched ...Microtubule function is modulated by the tubulin code, diverse posttranslational modifications that are altered dynamically by writer and eraser enzymes. Glutamylation-the addition of branched (isopeptide-linked) glutamate chains-is the most evolutionarily widespread tubulin modification. It is introduced by tubulin tyrosine ligase-like enzymes and erased by carboxypeptidases of the cytosolic carboxypeptidase (CCP) family. Glutamylation homeostasis, achieved through the balance of writers and erasers, is critical for normal cell function, and mutations in CCPs lead to human disease. Here we report cryo-electron microscopy structures of the glutamylation eraser CCP5 in complex with the microtubule, and X-ray structures in complex with transition-state analogues. Combined with NMR analysis, these analyses show that CCP5 deforms the tubulin main chain into a unique turn that enables lock-and-key recognition of the branch glutamate in a cationic pocket that is unique to CCP family proteins. CCP5 binding of the sequences flanking the branch point primarily through peptide backbone atoms enables processing of diverse tubulin isotypes and non-tubulin substrates. Unexpectedly, CCP5 exhibits inefficient processing of an abundant β-tubulin isotype in the brain. This work provides an atomistic view into glutamate branch recognition and resolution, and sheds light on homeostasis of the tubulin glutamylation syntax. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
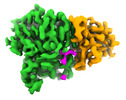
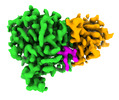
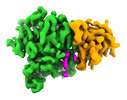
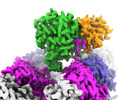
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42951.map.gz emd_42951.map.gz | 8.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42951-v30.xml emd-42951-v30.xml emd-42951.xml emd-42951.xml | 23.5 KB 23.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42951_fsc.xml emd_42951_fsc.xml | 21 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42951.png emd_42951.png | 98.4 KB | ||
| マスクデータ |  emd_42951_msk_1.map emd_42951_msk_1.map | 381.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-42951.cif.gz emd-42951.cif.gz | 6.8 KB | ||
| その他 |  emd_42951_additional_1.map.gz emd_42951_additional_1.map.gz emd_42951_additional_2.map.gz emd_42951_additional_2.map.gz emd_42951_half_map_1.map.gz emd_42951_half_map_1.map.gz emd_42951_half_map_2.map.gz emd_42951_half_map_2.map.gz | 337.3 MB 299.3 MB 300.9 MB 300.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42951 http://ftp.pdbj.org/pub/emdb/structures/EMD-42951 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42951 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42951 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42951_validation.pdf.gz emd_42951_validation.pdf.gz | 635.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42951_full_validation.pdf.gz emd_42951_full_validation.pdf.gz | 635.3 KB | 表示 | |
| XML形式データ |  emd_42951_validation.xml.gz emd_42951_validation.xml.gz | 24.1 KB | 表示 | |
| CIF形式データ |  emd_42951_validation.cif.gz emd_42951_validation.cif.gz | 32.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42951 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42951 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42951 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42951 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8v3rMC  8v3mC  8v3nC  8v3oC  8v3pC  8v3qC  8v3sC  8v4kC  8v4lC  8v4mC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42951.map.gz / 形式: CCP4 / 大きさ: 381.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42951.map.gz / 形式: CCP4 / 大きさ: 381.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CCP5 class#2 focused map, b-factor sharpened | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.245 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
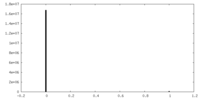
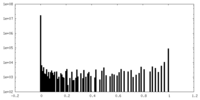
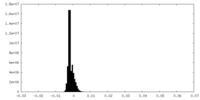
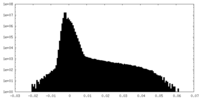
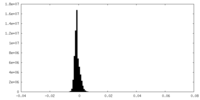
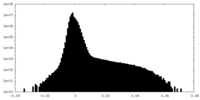
| ファイル |  emd_42951_msk_1.map emd_42951_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: CCP5 class#2 focused map, DeepEMhancer processed
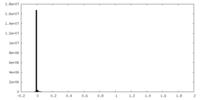
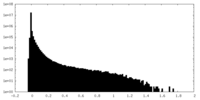
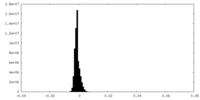
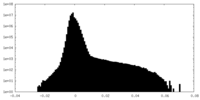
| ファイル | emd_42951_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CCP5 class#2 focused map, DeepEMhancer processed | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: CCP5 class#2 focused map, raw full map
| ファイル | emd_42951_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CCP5 class#2 focused map, raw full map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: CCP5 class#2 focused map, raw half map
| ファイル | emd_42951_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CCP5 class#2 focused map, raw half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: CCP5 class#2 focused map, raw half map
| ファイル | emd_42951_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CCP5 class#2 focused map, raw half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CCP5 in complex with beta tubulin tail
| 全体 | 名称: CCP5 in complex with beta tubulin tail |
|---|---|
| 要素 |
|
-超分子 #1: CCP5 in complex with beta tubulin tail
| 超分子 | 名称: CCP5 in complex with beta tubulin tail / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Focused refinement of CCP5:microtubule class#2 structure |
|---|
-分子 #1: Cytosolic carboxypeptidase-like protein 5
| 分子 | 名称: Cytosolic carboxypeptidase-like protein 5 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 68.229547 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: NELRCGGLLF SSRFDSGNLA HVEKVESLSS DGEGVGGGAS ALTSGIASSP DYEFNVWTRP DCAETEFENG NRSWFYFSVR GGMPGKLIK INIMNMNKQS KLYSQGMAPF VRTLPTRPRW ERIRDRPTFE MTETQFVLSF VHRFVEGRGA TTFFAFCYPF S YSDCQELL ...文字列: NELRCGGLLF SSRFDSGNLA HVEKVESLSS DGEGVGGGAS ALTSGIASSP DYEFNVWTRP DCAETEFENG NRSWFYFSVR GGMPGKLIK INIMNMNKQS KLYSQGMAPF VRTLPTRPRW ERIRDRPTFE MTETQFVLSF VHRFVEGRGA TTFFAFCYPF S YSDCQELL NQLDQRFPEN HPTHSSPLDT IYYHRELLCY SLDGLRVDLL TITSCHGLRE DREPRLEQLF PDTSTPRPFR FA GKRIFFL SSRVHPGETP SSFVFNGFLD FILRPDDPRA QTLRRLFVFK LIPMLNPDGV VRGHYRTDSR GVNLNRQYLK PDA VLHPAI YGAKAVLLYH HVHSRLNSQS SSEHQPSSCL PPDAPVSDLE KANNLQNEAQ CGHSADRHNA EAWKQTEPAE QKLN SVWIM PQQSAGLEES APDTIPPKES GVAYYVDLHG HASKRGCFMY GNSFSDESTQ VENMLYPKLI SLNSAHFDFQ GCNFS EKNM YARDRRDGQS KEGSGRVAIY KASGIIHSYT LACNYNTGRS VNSIPAACHD NGRASPPPPP AFPSRYTVEL FEQVGR AMA IAALDMAECN PWPRIVLSEH SSLTNLRAWM LKHVRNSRGL SS UniProtKB: Cytosolic carboxypeptidase-like protein 5 |
-分子 #2: beta tubulin tail
| 分子 | 名称: beta tubulin tail / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 487.548 Da |
| 配列 | 文字列: (UNK)(UNK)E(UNK)(UNK) |
-分子 #3: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #4: GLUTAMIC ACID
| 分子 | 名称: GLUTAMIC ACID / タイプ: ligand / ID: 4 / コピー数: 1 / 式: GLU |
|---|---|
| 分子量 | 理論値: 147.129 Da |
| Chemical component information |  ChemComp-GLU: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||||
| グリッド | モデル: Au-flat 1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 45 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 303 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均露光時間: 1.651 sec. / 平均電子線量: 53.34 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 135000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: Other / Chain - Initial model type: experimental model / 詳細: crystal structure of apo CCP5 |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: Cross-correlation coefficient |
| 得られたモデル |  PDB-8v3r: |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X