登録情報 データベース : EMDB / ID : EMD-41883タイトル Structure of the HER4/NRG1b Homodimer Extracellular Domain 複合体 : HER4/NRG1b homodimerタンパク質・ペプチド : Receptor tyrosine-protein kinase erbB-4タンパク質・ペプチド : Isoform 6 of Pro-neuregulin-1, membrane-bound isoformリガンド : 2-acetamido-2-deoxy-beta-D-glucopyranose / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
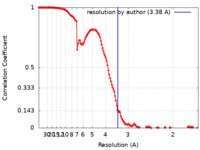
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / 解像度 : 3.38 Å Trenker R / Diwanji D / Bingham T / Verba KA / Jura N 資金援助 Organization Grant number 国 German Research Foundation (DFG) TR 1668/1-1 National Institutes of Health/National Cancer Institute (NIH/NCI) CA274502 National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) GM139636
ジャーナル : Elife / 年 : 2024タイトル : Structural dynamics of the active HER4 and HER2/HER4 complexes is finely tuned by different growth factors and glycosylation.著者 : Raphael Trenker / Devan Diwanji / Tanner Bingham / Kliment A Verba / Natalia Jura / 要旨 : Human Epidermal growth factor Receptor 4 (HER4 or ERBB4) carries out essential functions in the development and maintenance of the cardiovascular and nervous systems. HER4 activation is regulated by ... Human Epidermal growth factor Receptor 4 (HER4 or ERBB4) carries out essential functions in the development and maintenance of the cardiovascular and nervous systems. HER4 activation is regulated by a diverse group of extracellular ligands including the neuregulin (NRG) family and betacellulin (BTC), which promote HER4 homodimerization or heterodimerization with other HER receptors. Important cardiovascular functions of HER4 are exerted via heterodimerization with its close homolog and orphan receptor, HER2. To date structural insights into ligand-mediated HER4 activation have been limited to crystallographic studies of HER4 ectodomain homodimers in complex with NRG1β. Here, we report cryo-EM structures of near full-length HER2/HER4 heterodimers and full-length HER4 homodimers bound to NRG1β and BTC. We show that the structures of the heterodimers bound to either ligand are nearly identical and that in both cases the HER2/HER4 heterodimer interface is less dynamic than those observed in structures of HER2/EGFR and HER2/HER3 heterodimers. In contrast, structures of full-length HER4 homodimers bound to NRG1β and BTC display more large-scale dynamics mirroring states previously reported for EGFR homodimers. Our structures also reveal the presence of multiple glycan modifications within HER4 ectodomains, modeled for the first time in HER receptors, that distinctively contribute to the stabilization of HER4 homodimer interfaces over those of HER2/HER4 heterodimers. 履歴 登録 2023年9月10日 - ヘッダ(付随情報) 公開 2024年3月13日 - マップ公開 2024年3月13日 - 更新 2024年11月20日 - 現状 2024年11月20日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 ドイツ,
ドイツ,  米国, 3件
米国, 3件  引用
引用 ジャーナル: Elife / 年: 2024
ジャーナル: Elife / 年: 2024
 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_41883.map.gz
emd_41883.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-41883-v30.xml
emd-41883-v30.xml emd-41883.xml
emd-41883.xml EMDBヘッダ
EMDBヘッダ emd_41883_fsc.xml
emd_41883_fsc.xml FSCデータファイル
FSCデータファイル emd_41883.png
emd_41883.png emd_41883_msk_1.map
emd_41883_msk_1.map マスクマップ
マスクマップ emd-41883.cif.gz
emd-41883.cif.gz emd_41883_half_map_1.map.gz
emd_41883_half_map_1.map.gz emd_41883_half_map_2.map.gz
emd_41883_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-41883
http://ftp.pdbj.org/pub/emdb/structures/EMD-41883 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41883
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41883 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_41883.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_41883.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_41883_msk_1.map
emd_41883_msk_1.map 試料の構成要素
試料の構成要素 Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト)

 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー



























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)