+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4086 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | RNA polymerase I-Rrn3 complex | |||||||||
 マップデータ マップデータ | Pol I-Rrn3 complex | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.7 Å | |||||||||
 データ登録者 データ登録者 | Torreira E / Louro JA / Gil-Carton D / Gallego O / Calvo O / Fernandez-Tornero C | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2017 ジャーナル: Elife / 年: 2017タイトル: The dynamic assembly of distinct RNA polymerase I complexes modulates rDNA transcription. 著者: Eva Torreira / Jaime Alegrio Louro / Irene Pazos / Noelia González-Polo / David Gil-Carton / Ana Garcia Duran / Sébastien Tosi / Oriol Gallego / Olga Calvo / Carlos Fernández-Tornero /  要旨: Cell growth requires synthesis of ribosomal RNA by RNA polymerase I (Pol I). Binding of initiation factor Rrn3 activates Pol I, fostering recruitment to ribosomal DNA promoters. This fundamental ...Cell growth requires synthesis of ribosomal RNA by RNA polymerase I (Pol I). Binding of initiation factor Rrn3 activates Pol I, fostering recruitment to ribosomal DNA promoters. This fundamental process must be precisely regulated to satisfy cell needs at any time. We present in vivo evidence that, when growth is arrested by nutrient deprivation, cells induce rapid clearance of Pol I-Rrn3 complexes, followed by the assembly of inactive Pol I homodimers. This dual repressive mechanism reverts upon nutrient addition, thus restoring cell growth. Moreover, Pol I dimers also form after inhibition of either ribosome biogenesis or protein synthesis. Our mutational analysis, based on the electron cryomicroscopy structures of monomeric Pol I alone and in complex with Rrn3, underscores the central role of subunits A43 and A14 in the regulation of differential Pol I complexes assembly and subsequent promoter association. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4086.map.gz emd_4086.map.gz | 2.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4086-v30.xml emd-4086-v30.xml emd-4086.xml emd-4086.xml | 13.7 KB 13.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4086_fsc.xml emd_4086_fsc.xml | 5.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4086.png emd_4086.png | 120.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4086 http://ftp.pdbj.org/pub/emdb/structures/EMD-4086 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4086 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4086 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4086_validation.pdf.gz emd_4086_validation.pdf.gz | 246.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4086_full_validation.pdf.gz emd_4086_full_validation.pdf.gz | 245.5 KB | 表示 | |
| XML形式データ |  emd_4086_validation.xml.gz emd_4086_validation.xml.gz | 8.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4086 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4086 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4086 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4086 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4086.map.gz / 形式: CCP4 / 大きさ: 16.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4086.map.gz / 形式: CCP4 / 大きさ: 16.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Pol I-Rrn3 complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.77 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
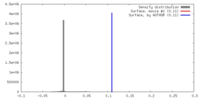
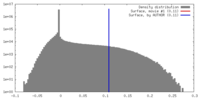
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : S. cerevisiae RNA polymerase I in complex with activating factor Rrn3
| 全体 | 名称: S. cerevisiae RNA polymerase I in complex with activating factor Rrn3 |
|---|---|
| 要素 |
|
-超分子 #1: S. cerevisiae RNA polymerase I in complex with activating factor Rrn3
| 超分子 | 名称: S. cerevisiae RNA polymerase I in complex with activating factor Rrn3 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#14 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 594 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.06 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.8 / 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-68 / 撮影したグリッド数: 1 / 実像数: 1288 / 平均露光時間: 4.0 sec. / 平均電子線量: 68.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 79096 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 4.2 µm / 最小 デフォーカス(公称値): 1.9 µm / 倍率(公称値): 47000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 537 |
|---|
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)