+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3305 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of human TFIID-IIA bound to core promoter DNA (locally-refined BC core) | |||||||||
 マップデータ マップデータ | Locally-refined BC-core of human TFIID-IIA bound to super core promoter DNA | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | TFIID / TFIIA / transcription / RNA polymerase II / general transcription factors / preinitiation complex / core promoter / DNA binding | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of MHC class I biosynthetic process / spermine transport / DNA-templated transcription open complex formation / positive regulation of androgen receptor signaling pathway / TFIIH-class transcription factor complex binding / negative regulation of protein autoubiquitination / negative regulation of MHC class II biosynthetic process / transcription factor TFTC complex / RNA polymerase I general transcription initiation factor activity / regulation of cell cycle G1/S phase transition ...negative regulation of MHC class I biosynthetic process / spermine transport / DNA-templated transcription open complex formation / positive regulation of androgen receptor signaling pathway / TFIIH-class transcription factor complex binding / negative regulation of protein autoubiquitination / negative regulation of MHC class II biosynthetic process / transcription factor TFTC complex / RNA polymerase I general transcription initiation factor activity / regulation of cell cycle G1/S phase transition / RNA polymerase transcription factor SL1 complex / SLIK (SAGA-like) complex / histone H4K16ac reader activity / RNA polymerase III general transcription initiation factor activity / RNA polymerase I core promoter sequence-specific DNA binding / maintenance of protein location in nucleus / RNA Polymerase III Transcription Initiation From Type 1 Promoter / RNA Polymerase III Transcription Initiation From Type 2 Promoter / RNA Polymerase III Transcription Initiation From Type 3 Promoter / transcription factor TFIIA complex / RNA Polymerase III Abortive And Retractive Initiation / female germ cell nucleus / male pronucleus / female pronucleus / RNA polymerase II general transcription initiation factor binding / nuclear vitamin D receptor binding / nuclear thyroid hormone receptor binding / regulation of fat cell differentiation / SAGA complex / RNA Polymerase I Transcription Termination / transcription preinitiation complex / inner cell mass cell proliferation / RNA polymerase II general transcription initiation factor activity / transcription factor TFIID complex / histone acetyltransferase binding / cellular response to ATP / HIV Transcription Initiation / RNA Polymerase II HIV Promoter Escape / Transcription of the HIV genome / RNA Polymerase II Promoter Escape / RNA Polymerase II Transcription Pre-Initiation And Promoter Opening / RNA Polymerase II Transcription Initiation / RNA Polymerase II Transcription Initiation And Promoter Clearance / midbrain development / negative regulation of signal transduction by p53 class mediator / ubiquitin conjugating enzyme activity / transcription initiation at RNA polymerase I promoter / negative regulation of cell cycle / aryl hydrocarbon receptor binding / TFIIB-class transcription factor binding / P-TEFb complex binding / RNA Polymerase I Transcription Initiation / MLL1 complex / transcription by RNA polymerase III / RNA polymerase II transcribes snRNA genes / negative regulation of ubiquitin-dependent protein catabolic process / positive regulation of transcription initiation by RNA polymerase II / histone acetyltransferase activity / RNA polymerase II core promoter sequence-specific DNA binding / regulation of DNA repair / core promoter sequence-specific DNA binding / negative regulation of protein kinase activity / transcription regulator inhibitor activity / RNA polymerase II preinitiation complex assembly / histone acetyltransferase / positive regulation of intrinsic apoptotic signaling pathway / estrogen receptor signaling pathway / RNA Polymerase II Pre-transcription Events / TBP-class protein binding / regulation of signal transduction by p53 class mediator / SIRT1 negatively regulates rRNA expression / nuclear receptor binding / male germ cell nucleus / transcription initiation at RNA polymerase II promoter / DNA-templated transcription initiation / RNA Polymerase I Promoter Escape / mRNA transcription by RNA polymerase II / euchromatin / NoRC negatively regulates rRNA expression / B-WICH complex positively regulates rRNA expression / G2/M transition of mitotic cell cycle / protein polyubiquitination / cellular response to UV / p53 binding / kinase activity / positive regulation of proteasomal ubiquitin-dependent protein catabolic process / chromatin organization / protein autophosphorylation / ubiquitin-dependent protein catabolic process / Regulation of TP53 Activity through Phosphorylation / spermatogenesis / transcription regulator complex / DNA-binding transcription factor binding / Estrogen-dependent gene expression / sequence-specific DNA binding / RNA polymerase II-specific DNA-binding transcription factor binding / transcription by RNA polymerase II / cell differentiation / transcription coactivator activity / transcription cis-regulatory region binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
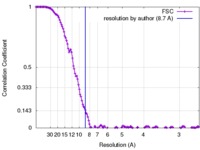
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 8.7 Å | |||||||||
 データ登録者 データ登録者 | Louder RK / He Y / Lopez-Blanco JR / Fang J / Chacon P / Nogales E | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2016 ジャーナル: Nature / 年: 2016タイトル: Structure of promoter-bound TFIID and model of human pre-initiation complex assembly. 著者: Robert K Louder / Yuan He / José Ramón López-Blanco / Jie Fang / Pablo Chacón / Eva Nogales /   要旨: The general transcription factor IID (TFIID) plays a central role in the initiation of RNA polymerase II (Pol II)-dependent transcription by nucleating pre-initiation complex (PIC) assembly at the ...The general transcription factor IID (TFIID) plays a central role in the initiation of RNA polymerase II (Pol II)-dependent transcription by nucleating pre-initiation complex (PIC) assembly at the core promoter. TFIID comprises the TATA-binding protein (TBP) and 13 TBP-associated factors (TAF1-13), which specifically interact with a variety of core promoter DNA sequences. Here we present the structure of human TFIID in complex with TFIIA and core promoter DNA, determined by single-particle cryo-electron microscopy at sub-nanometre resolution. All core promoter elements are contacted by subunits of TFIID, with TAF1 and TAF2 mediating major interactions with the downstream promoter. TFIIA bridges the TBP-TATA complex with lobe B of TFIID. We also present the cryo-electron microscopy reconstruction of a fully assembled human TAF-less PIC. Superposition of common elements between the two structures provides novel insights into the general role of TFIID in promoter recognition, PIC assembly, and transcription initiation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3305.map.gz emd_3305.map.gz | 6.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3305-v30.xml emd-3305-v30.xml emd-3305.xml emd-3305.xml | 12.7 KB 12.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3305_fsc.xml emd_3305_fsc.xml | 16 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3305.png emd_3305.png | 63.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3305 http://ftp.pdbj.org/pub/emdb/structures/EMD-3305 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3305 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3305 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3305_validation.pdf.gz emd_3305_validation.pdf.gz | 223.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3305_full_validation.pdf.gz emd_3305_full_validation.pdf.gz | 222.6 KB | 表示 | |
| XML形式データ |  emd_3305_validation.xml.gz emd_3305_validation.xml.gz | 13.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3305 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3305 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3305 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3305 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3305.map.gz / 形式: CCP4 / 大きさ: 210.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3305.map.gz / 形式: CCP4 / 大きさ: 210.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Locally-refined BC-core of human TFIID-IIA bound to super core promoter DNA | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.32 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human TFIID-TFIIA complex bound to super core promoter DNA
| 全体 | 名称: Human TFIID-TFIIA complex bound to super core promoter DNA |
|---|---|
| 要素 |
|
-超分子 #1000: Human TFIID-TFIIA complex bound to super core promoter DNA
| 超分子 | 名称: Human TFIID-TFIIA complex bound to super core promoter DNA タイプ: sample / ID: 1000 / Number unique components: 3 |
|---|---|
| 分子量 | 理論値: 1.34 MDa |
-分子 #1: General transcription factor IID
| 分子 | 名称: General transcription factor IID / タイプ: protein_or_peptide / ID: 1 / Name.synonym: TFIID / コピー数: 1 / 集合状態: Monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 株: HeLa / 別称: Human / Organelle: Nucleus / 細胞中の位置: Nuclear extract Homo sapiens (ヒト) / 株: HeLa / 別称: Human / Organelle: Nucleus / 細胞中の位置: Nuclear extract |
| 分子量 | 理論値: 1.26 MDa |
-分子 #2: General transcription factor IIA
| 分子 | 名称: General transcription factor IIA / タイプ: protein_or_peptide / ID: 2 / Name.synonym: TFIIA / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 27 KDa |
| 組換発現 | 生物種:  |
-分子 #3: Super core promoter
| 分子 | 名称: Super core promoter / タイプ: dna / ID: 3 / Name.synonym: SCP 詳細: The super core promoter is a composite sequence combining promoter motifs from several strong promoters from humans and D. melanogaster. 分類: DNA / Structure: DOUBLE HELIX / Synthetic?: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 56 KDa |
| 配列 | 文字列: GAAGGGCGCC TATAAAAGGG GGTGGGGGCG CGTTCGTCCT CAGTCGCGAT CGAACACTCG AGCCGAGCAG ACGTGCCTAC GGACCATGG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.05 mg/mL |
|---|---|
| 緩衝液 | pH: 7.9 詳細: 10 mM HEPES, 10 mM MgCl2, 50 mM KCl, 3% trehalose 1 mM DTT, 0.0125% NP-40 |
| グリッド | 詳細: Amorphous continuous carbon over C-flat holey carbon support (4 um holes with 2 um spacing) on 400 mesh copper grid. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV 手法: Incubate 4 ul of sample on grid for 10 minutes, then blot for 4 seconds with force 15. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN |
|---|---|
| 詳細 | The camera was operated in counting mode with a dose rate of 8 electrons/pixel per second, with a total exposure time of 10 seconds fractionated over 20 frames. |
| 日付 | 2014年8月11日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 実像数: 1253 / 平均電子線量: 46 e/Å2 詳細: Whole-micrograph drift correction was performed using MotionCorr before averaging the frames. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 37879 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 2.0 µm |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-5fur: |
-原子モデル構築 2
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-5fur: |
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)