+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-32092 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of amyloid fibril formed by FUS low complexity domain | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | amyloid fibril / PROTEIN FIBRIL | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報membraneless organelle assembly / mRNA stabilization / intracellular membraneless organelle / regulation of RNA splicing / Processing of Capped Intron-Containing Pre-mRNA / postsynaptic cytosol / positive regulation of double-strand break repair via homologous recombination / presynaptic cytosol / mRNA Splicing - Major Pathway / RNA splicing ...membraneless organelle assembly / mRNA stabilization / intracellular membraneless organelle / regulation of RNA splicing / Processing of Capped Intron-Containing Pre-mRNA / postsynaptic cytosol / positive regulation of double-strand break repair via homologous recombination / presynaptic cytosol / mRNA Splicing - Major Pathway / RNA splicing / mRNA 3'-UTR binding / transcription coregulator activity / molecular condensate scaffold activity / protein homooligomerization / GABA-ergic synapse / amyloid fibril formation / transcription coactivator activity / chromatin binding / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II / glutamatergic synapse / DNA binding / RNA binding / zinc ion binding / nucleoplasm / identical protein binding / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Sun YP / Xia WC | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: iScience / 年: 2022 ジャーナル: iScience / 年: 2022タイトル: Molecular structure of an amyloid fibril formed by FUS low-complexity domain. 著者: Yunpeng Sun / Shenqing Zhang / Jiaojiao Hu / Youqi Tao / Wencheng Xia / Jinge Gu / Yichen Li / Qin Cao / Dan Li / Cong Liu /  要旨: FUS is a multifunctional nuclear protein which undergoes liquid-liquid phase separation in response to stress and DNA damage. Dysregulation of FUS dynamic phase separation leads to formation of ...FUS is a multifunctional nuclear protein which undergoes liquid-liquid phase separation in response to stress and DNA damage. Dysregulation of FUS dynamic phase separation leads to formation of pathological fibril closely associated with neurodegenerative diseases such as amyotrophic lateral sclerosis and frontotemporal dementia. In this study, we determined the cryo-EM structure of a cytotoxic fibril formed by the low-complexity (LC) domain of FUS at 2.9 Å resolution. The fibril structure exhibits a new and extensive serpentine fold consisting of three motifs incorporating together via a Tyr triad. FUS LC employs 91 residues to form an enlarged and stable fibril core via hydrophilic interaction and hydrogen bonds, which is distinct from most of previously determined fibrils commonly stabilized by hydrophobic interaction. Our work reveals the structural basis underlying formation of a cytotoxic and thermostable fibril of FUS LC and sheds light on understanding the liquid-to-solid phase transition of FUS in disease. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32092.map.gz emd_32092.map.gz | 59.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32092-v30.xml emd-32092-v30.xml emd-32092.xml emd-32092.xml | 9.3 KB 9.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_32092_fsc.xml emd_32092_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_32092.png emd_32092.png | 33.1 KB | ||
| Filedesc metadata |  emd-32092.cif.gz emd-32092.cif.gz | 5.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32092 http://ftp.pdbj.org/pub/emdb/structures/EMD-32092 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32092 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32092 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32092_validation.pdf.gz emd_32092_validation.pdf.gz | 594.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32092_full_validation.pdf.gz emd_32092_full_validation.pdf.gz | 594.4 KB | 表示 | |
| XML形式データ |  emd_32092_validation.xml.gz emd_32092_validation.xml.gz | 10.9 KB | 表示 | |
| CIF形式データ |  emd_32092_validation.cif.gz emd_32092_validation.cif.gz | 14.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32092 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32092 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32092 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32092 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32092.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32092.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Amyloid fibril formed by FUS low complexity domain
| 全体 | 名称: Amyloid fibril formed by FUS low complexity domain |
|---|---|
| 要素 |
|
-超分子 #1: Amyloid fibril formed by FUS low complexity domain
| 超分子 | 名称: Amyloid fibril formed by FUS low complexity domain / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: fusion protein of mCerulean and FUS LCD
| 分子 | 名称: fusion protein of mCerulean and FUS LCD / タイプ: protein_or_peptide / ID: 1 詳細: 6His-tagged mCerulean:(FPbase ID: J2JWA, Link: https://www.fpbase.org/protein/mcerulean/) コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 51.948352 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSYYHHHHHH DYDIPTTENL YFQGAMVSKG EELFTGVVPI LVELDGDVNG HKFSVSGEGE GDATYGKLTL KFICTTGKLP VPWPTLVTT LTWGVQCFAR YPDHMKQHDF FKSAMPEGYV QERTIFFKDD GNYKTRAEVK FEGDTLVNRI ELKGIDFKED G NILGHKLE ...文字列: MSYYHHHHHH DYDIPTTENL YFQGAMVSKG EELFTGVVPI LVELDGDVNG HKFSVSGEGE GDATYGKLTL KFICTTGKLP VPWPTLVTT LTWGVQCFAR YPDHMKQHDF FKSAMPEGYV QERTIFFKDD GNYKTRAEVK FEGDTLVNRI ELKGIDFKED G NILGHKLE YNAISDNVYI TADKQKNGIK ANFKIRHNIE DGSVQLADHY QQNTPIGDGP VLLPDNHYLS TQSKLSKDPN EK RDHMVLL EFVTAAGITL GMDELYKAGT MDPASNDYTQ QATQSYGAYP TQPGQGYSQQ SSQPYGQQSY SGYSQSTDTS GYG QSSYSS YGQSQNTGYG TQSTPQGYGS TGGYGSSQSS QSSYGQQSSY PGYGQQPAPS STSGSYGSSS QSSSYGQPQS GSYS QQPSY GGQQQSYGQQ QSYNPPQGYG QQNQYNSSSG GGGGGGGGGN YGQDQSSMSS GGGSGGGYGN QDQSGGGGSG GYGQQ DRG UniProtKB: RNA-binding protein FUS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



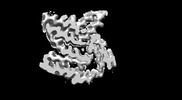
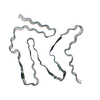













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















