+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
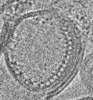
| タイトル | Reconstituted proteoliposomes of TRIM72 in positive curvature #2 | |||||||||
 マップデータ マップデータ | Reconstituted proteoliposomes of TRIM72 in positive curvature #2 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | TRIM / Tripartite motif / Ubiquitin ligase / Coiled coil / B-box / PRY-SPRY / Membrane protein / LIGASE / METAL BINDING PROTEIN | |||||||||
| 生物種 |  | |||||||||
| 手法 | 電子線トモグラフィー法 / クライオ電子顕微鏡法 | |||||||||
 データ登録者 データ登録者 | Park SH / Song HK | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Structure and activation of the RING E3 ubiquitin ligase TRIM72 on the membrane. 著者: Si Hoon Park / Juhyun Han / Byung-Cheon Jeong / Ju Han Song / Se Hwan Jang / Hyeongseop Jeong / Bong Heon Kim / Young-Gyu Ko / Zee-Yong Park / Kyung Eun Lee / Jaekyung Hyun / Hyun Kyu Song /    要旨: Defects in plasma membrane repair can lead to muscle and heart diseases in humans. Tripartite motif-containing protein (TRIM)72 (mitsugumin 53; MG53) has been determined to rapidly nucleate vesicles ...Defects in plasma membrane repair can lead to muscle and heart diseases in humans. Tripartite motif-containing protein (TRIM)72 (mitsugumin 53; MG53) has been determined to rapidly nucleate vesicles at the site of membrane damage, but the underlying molecular mechanisms remain poorly understood. Here we present the structure of Mus musculus TRIM72, a complete model of a TRIM E3 ubiquitin ligase. We demonstrated that the interaction between TRIM72 and phosphatidylserine-enriched membranes is necessary for its oligomeric assembly and ubiquitination activity. Using cryogenic electron tomography and subtomogram averaging, we elucidated a higher-order model of TRIM72 assembly on the phospholipid bilayer. Combining structural and biochemical techniques, we developed a working molecular model of TRIM72, providing insights into the regulation of RING-type E3 ligases through the cooperation of multiple domains in higher-order assemblies. Our findings establish a fundamental basis for the study of TRIM E3 ligases and have therapeutic implications for diseases associated with membrane repair. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31152.map.gz emd_31152.map.gz | 801.3 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31152-v30.xml emd-31152-v30.xml emd-31152.xml emd-31152.xml | 8.3 KB 8.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_31152.png emd_31152.png | 126.7 KB | ||
| Filedesc metadata |  emd-31152.cif.gz emd-31152.cif.gz | 4.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31152 http://ftp.pdbj.org/pub/emdb/structures/EMD-31152 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31152 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31152 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_31152_validation.pdf.gz emd_31152_validation.pdf.gz | 519.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_31152_full_validation.pdf.gz emd_31152_full_validation.pdf.gz | 519.1 KB | 表示 | |
| XML形式データ |  emd_31152_validation.xml.gz emd_31152_validation.xml.gz | 2 KB | 表示 | |
| CIF形式データ |  emd_31152_validation.cif.gz emd_31152_validation.cif.gz | 2.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31152 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31152 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31152 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31152 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31152.map.gz / 形式: CCP4 / 大きさ: 1.7 MB / タイプ: IMAGE STORED AS SIGNED BYTE ダウンロード / ファイル: emd_31152.map.gz / 形式: CCP4 / 大きさ: 1.7 MB / タイプ: IMAGE STORED AS SIGNED BYTE | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstituted proteoliposomes of TRIM72 in positive curvature #2 | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5.6 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : TRIM72 complex with phosphatidylserine-enriched liposomes.
| 全体 | 名称: TRIM72 complex with phosphatidylserine-enriched liposomes. |
|---|---|
| 要素 |
|
-超分子 #1: TRIM72 complex with phosphatidylserine-enriched liposomes.
| 超分子 | 名称: TRIM72 complex with phosphatidylserine-enriched liposomes. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: TRIM72
| 分子 | 名称: TRIM72 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSAAPGLLRQ ELSCPLCLQL FDAPVTAECG HSFCRACLIR VAGEPAADGT VACPCCQAPT RPQALSTNL QLSRLVEGLA QVPQGHCEEH LDPLSIYCEQ DRTLVCGVCA SLGSHRGHRL L PAAEAQAR LKTQLPQQKM QLQEACMRKE KTVAVLEHQL VEVEETVRQF ...文字列: GSAAPGLLRQ ELSCPLCLQL FDAPVTAECG HSFCRACLIR VAGEPAADGT VACPCCQAPT RPQALSTNL QLSRLVEGLA QVPQGHCEEH LDPLSIYCEQ DRTLVCGVCA SLGSHRGHRL L PAAEAQAR LKTQLPQQKM QLQEACMRKE KTVAVLEHQL VEVEETVRQF RGAVGEQLGK MR MFLAALE SSLDREAERV RGDAGVALRR ELSSLNSYLE QLRQMEKVLE EVADKPQTEF LMK FCLVTS RLQKILSESP PPARLDIQLP VISDDFKFQV WKKMFRALMP ALEELTFDPS SAHP SLVVS SSGRRVECSD QKAPPAGEDT RQFDKAVAVV AQQLLSQGEH YWEVEVGDKP RWALG VMAA DASRRGRLHA VPSQGLWLLG LRDGKILEAH VEAKEPRALR TPERPPARIG LYLSFA DGV LAFYDASNPD VLTPIFSFHE RLPGPVYPIF DVCWHDKGKN AQPLLLVGPE QEQA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
| 切片作成 | その他: NO SECTIONING |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 平均電子線量: 3.2 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 使用した粒子像数: 61 |
|---|
 ムービー
ムービー コントローラー
コントローラー