+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | B. subtilis GS(14)-Q-GlnR peptide | ||||||||||||
 マップデータ マップデータ | Sharpened (B factor 74.9 A^2) | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | glutamine synthetase repressor tetradecamer / BIOSYNTHETIC PROTEIN / LIGASE | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報glutamine synthetase / glutamine biosynthetic process / glutamine synthetase activity / DNA-binding transcription factor activity / negative regulation of DNA-templated transcription / DNA binding / ATP binding / metal ion binding / identical protein binding / cytoplasm 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 1.96 Å | ||||||||||||
 データ登録者 データ登録者 | Travis BA / Peck J | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Molecular dissection of the glutamine synthetase-GlnR nitrogen regulatory circuitry in Gram-positive bacteria. 著者: Brady A Travis / Jared V Peck / Raul Salinas / Brandon Dopkins / Nicholas Lent / Viet D Nguyen / Mario J Borgnia / Richard G Brennan / Maria A Schumacher /  要旨: How bacteria sense and respond to nitrogen levels are central questions in microbial physiology. In Gram-positive bacteria, nitrogen homeostasis is controlled by an operon encoding glutamine ...How bacteria sense and respond to nitrogen levels are central questions in microbial physiology. In Gram-positive bacteria, nitrogen homeostasis is controlled by an operon encoding glutamine synthetase (GS), a dodecameric machine that assimilates ammonium into glutamine, and the GlnR repressor. GlnR detects nitrogen excess indirectly by binding glutamine-feedback-inhibited-GS (FBI-GS), which activates its transcription-repression function. The molecular mechanisms behind this regulatory circuitry, however, are unknown. Here we describe biochemical and structural analyses of GS and FBI-GS-GlnR complexes from pathogenic and non-pathogenic Gram-positive bacteria. The structures show FBI-GS binds the GlnR C-terminal domain within its active-site cavity, juxtaposing two GlnR monomers to form a DNA-binding-competent GlnR dimer. The FBI-GS-GlnR interaction stabilizes the inactive GS conformation. Strikingly, this interaction also favors a remarkable dodecamer to tetradecamer transition in some GS, breaking the paradigm that all bacterial GS are dodecamers. These data thus unveil unique structural mechanisms of transcription and enzymatic regulation. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25869.map.gz emd_25869.map.gz | 230.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25869-v30.xml emd-25869-v30.xml emd-25869.xml emd-25869.xml | 14.7 KB 14.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_25869.png emd_25869.png | 126.3 KB | ||
| Filedesc metadata |  emd-25869.cif.gz emd-25869.cif.gz | 5.6 KB | ||
| その他 |  emd_25869_additional_1.map.gz emd_25869_additional_1.map.gz | 122.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25869 http://ftp.pdbj.org/pub/emdb/structures/EMD-25869 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25869 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25869 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25869_validation.pdf.gz emd_25869_validation.pdf.gz | 554.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25869_full_validation.pdf.gz emd_25869_full_validation.pdf.gz | 553.9 KB | 表示 | |
| XML形式データ |  emd_25869_validation.xml.gz emd_25869_validation.xml.gz | 7.1 KB | 表示 | |
| CIF形式データ |  emd_25869_validation.cif.gz emd_25869_validation.cif.gz | 8.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25869 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25869 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25869 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25869 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7tfcMC  7tdpC  7tdvC  7teaC  7tecC  7tenC  7tf6C  7tf7C  7tf9C  7tfaC  7tfbC  7tfdC  7tfeC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25869.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25869.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
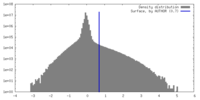
| 注釈 | Sharpened (B factor 74.9 A^2) | ||||||||||||||||||||||||||||||||||||
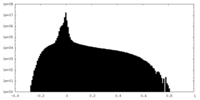
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.88 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unsharpened
| ファイル | emd_25869_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
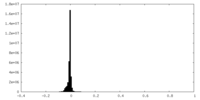
| 注釈 | Unsharpened | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Tetradecameric B. subtilis GS complex with glutamine and GlnR C-t...
| 全体 | 名称: Tetradecameric B. subtilis GS complex with glutamine and GlnR C-tail peptides |
|---|---|
| 要素 |
|
-超分子 #1: Tetradecameric B. subtilis GS complex with glutamine and GlnR C-t...
| 超分子 | 名称: Tetradecameric B. subtilis GS complex with glutamine and GlnR C-tail peptides タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|
-分子 #1: Glutamine synthetase
| 分子 | 名称: Glutamine synthetase / タイプ: protein_or_peptide / ID: 1 / コピー数: 14 / 光学異性体: LEVO / EC番号: glutamine synthetase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 52.509449 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSGLVPRGSH MAKYTREDIE KLVKEENVKY IRLQFTDILG TIKNVEIPVS QLGKALDNKV MFDGSSIEGF VRIEESDMY LYPDLNTFVI FPWTAEKGKV ARFICDIYNP DGTPFEGDPR NNLKRILKEM EDLGFSDFNL GPEPEFFLFK L DEKGEPTL ...文字列: MGSSHHHHHH SSGLVPRGSH MAKYTREDIE KLVKEENVKY IRLQFTDILG TIKNVEIPVS QLGKALDNKV MFDGSSIEGF VRIEESDMY LYPDLNTFVI FPWTAEKGKV ARFICDIYNP DGTPFEGDPR NNLKRILKEM EDLGFSDFNL GPEPEFFLFK L DEKGEPTL ELNDKGGYFD LAPTDLGENC RRDIVLELEE MGFEIEASHH EVAPGQHEID FKYAGAVRSC DDIQTFKLVV KT IARKHGL HATFMPKPLF GVNGSGMHCN LSLFKNGVNA FFDENADLQL SETAKHFIAG IVKHATSFTA VTNPTVNSYK RLV PGYEAP CYVAWSAQNR SPLIRIPASR GISTRVEVRS VDPAANPYLA LSVLLAAGLD GIKNKLEAPA PIDRNIYVMS KEER MENGI VDLPATLAEA LEEFKSNEVM VKALGEHLFE HFIEAKEIEW DMFRTQVHPW EREQYMSQY UniProtKB: Glutamine synthetase |
-分子 #2: GlnR C-tail peptide
| 分子 | 名称: GlnR C-tail peptide / タイプ: protein_or_peptide / ID: 2 / コピー数: 14 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 1.246396 KDa |
| 配列 | 文字列: TFRQGDMSRF UniProtKB: HTH-type transcriptional regulator GlnR |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 28 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: GLUTAMINE
| 分子 | 名称: GLUTAMINE / タイプ: ligand / ID: 4 / コピー数: 14 / 式: GLN |
|---|---|
| 分子量 | 理論値: 146.144 Da |
| Chemical component information |  ChemComp-GLN: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 43.74 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.4 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: D7 (2回x7回 2面回転対称) 解像度のタイプ: BY AUTHOR / 解像度: 1.96 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 3.2) / 使用した粒子像数: 1376404 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3.2) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3.2) |
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-7tfc: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)