+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the C-terminal half of Leucine Rich Repeat Kinase 1 (LRRK1) | |||||||||
 マップデータ マップデータ | Catalytic C-terminus of Leucine Rich Repeat Kinase 1 (LRRK1) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | kinase / gtpase / CYTOSOLIC PROTEIN | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
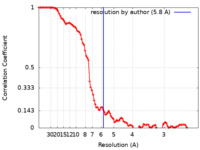
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.8 Å | |||||||||
 データ登録者 データ登録者 | Matyszewski M / Snead DM / Leschziner AE | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2022 ジャーナル: Nat Struct Mol Biol / 年: 2022タイトル: Structural basis for Parkinson's disease-linked LRRK2's binding to microtubules. 著者: David M Snead / Mariusz Matyszewski / Andrea M Dickey / Yu Xuan Lin / Andres E Leschziner / Samara L Reck-Peterson /  要旨: Leucine-rich repeat kinase 2 (LRRK2) is one of the most commonly mutated genes in familial Parkinson's disease (PD). Under some circumstances, LRRK2 co-localizes with microtubules in cells, an ...Leucine-rich repeat kinase 2 (LRRK2) is one of the most commonly mutated genes in familial Parkinson's disease (PD). Under some circumstances, LRRK2 co-localizes with microtubules in cells, an association enhanced by PD mutations. We report a cryo-EM structure of the catalytic half of LRRK2, containing its kinase, in a closed conformation, and GTPase domains, bound to microtubules. We also report a structure of the catalytic half of LRRK1, which is closely related to LRRK2 but is not linked to PD. Although LRRK1's structure is similar to that of LRRK2, we find that LRRK1 does not interact with microtubules. Guided by these structures, we identify amino acids in LRRK2's GTPase that mediate microtubule binding; mutating them disrupts microtubule binding in vitro and in cells, without affecting LRRK2's kinase activity. Our results have implications for the design of therapeutic LRRK2 kinase inhibitors. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25672.map.gz emd_25672.map.gz | 85.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25672-v30.xml emd-25672-v30.xml emd-25672.xml emd-25672.xml | 20.1 KB 20.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25672_fsc.xml emd_25672_fsc.xml | 10 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25672.png emd_25672.png | 43.2 KB | ||
| マスクデータ |  emd_25672_msk_1.map emd_25672_msk_1.map | 91.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-25672.cif.gz emd-25672.cif.gz | 6.3 KB | ||
| その他 |  emd_25672_additional_1.map.gz emd_25672_additional_1.map.gz emd_25672_half_map_1.map.gz emd_25672_half_map_1.map.gz emd_25672_half_map_2.map.gz emd_25672_half_map_2.map.gz | 45.3 MB 84.5 MB 84.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25672 http://ftp.pdbj.org/pub/emdb/structures/EMD-25672 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25672 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25672 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25672_validation.pdf.gz emd_25672_validation.pdf.gz | 864.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25672_full_validation.pdf.gz emd_25672_full_validation.pdf.gz | 864.3 KB | 表示 | |
| XML形式データ |  emd_25672_validation.xml.gz emd_25672_validation.xml.gz | 17.3 KB | 表示 | |
| CIF形式データ |  emd_25672_validation.cif.gz emd_25672_validation.cif.gz | 21.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25672 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25672 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25672 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25672 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25672.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25672.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Catalytic C-terminus of Leucine Rich Repeat Kinase 1 (LRRK1) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.16 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_25672_msk_1.map emd_25672_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
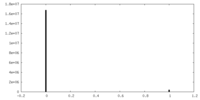
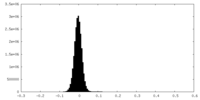
| 密度ヒストグラム |
-追加マップ: Non-sharpened map of LRRK1
| ファイル | emd_25672_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Non-sharpened map of LRRK1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
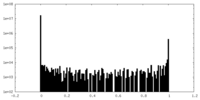
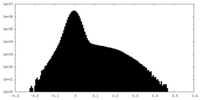
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_25672_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
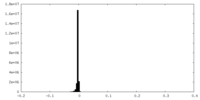
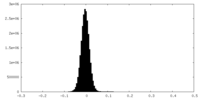
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_25672_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
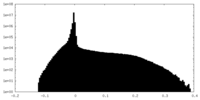
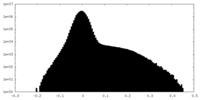
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Catalytic C-terminus of Leucine Rich Repeat Kinase 1
| 全体 | 名称: Catalytic C-terminus of Leucine Rich Repeat Kinase 1 |
|---|---|
| 要素 |
|
-超分子 #1: Catalytic C-terminus of Leucine Rich Repeat Kinase 1
| 超分子 | 名称: Catalytic C-terminus of Leucine Rich Repeat Kinase 1 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 155 KDa |
-分子 #1: C-terminal half of Leucine Rich Repeat Kinase 1
| 分子 | 名称: C-terminal half of Leucine Rich Repeat Kinase 1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: RKAEKCKLMK MIIVGPPRQG KSTLLEILQT GRAPQVVHGE ATIRTTKWEL QRPAGSRAKV ESVEFNVWD IGGPASMATV NQCFFTDKAL YVVVWNLALG EEAVANLQFW LLNIEAKAPN A VVLVVGTH LDLIEAKFRV ERIATLRAYV LALCRSPSGS RATGFPDITF ...文字列: RKAEKCKLMK MIIVGPPRQG KSTLLEILQT GRAPQVVHGE ATIRTTKWEL QRPAGSRAKV ESVEFNVWD IGGPASMATV NQCFFTDKAL YVVVWNLALG EEAVANLQFW LLNIEAKAPN A VVLVVGTH LDLIEAKFRV ERIATLRAYV LALCRSPSGS RATGFPDITF KHLHEISCKS LE GQEGLRQ LIFHVTCSMK DVGSTIGCQR LAGRLIPRSY LSLQEAVLAE QQRRSRDDDV QYL TDRQLE QLVEQTPDND IKDYEDLQSA ISFLIETGTL LHFPDTSHGL RNLYFLDPIW LSEC LQRIF NIKGSRSVAK NGVIRAEDLR MLLVGTGFTQ QTEEQYFQFL AKFEIALPVA NDSYL LPHL LPSKPGLDTH GMRHPTANTI QRVFKMSFVP VGFWQRFIAR MLISLAEMDL QLFENK KNT KSRNRKVTIY SFTGNQRNRC STFRVKRNQT IYWQEGLLVT FDGGYLSVES SDVNWKK KK SGGMKIVCQS EVRDFSAMAF ITDHVNSLID QWFPALTATE SDGTPLMEQY VPCPVCET A WAQHTDPSEK SEDVQYFDME DCVLTAIERD FISCPRHPDL PVPLQELVPE LFMTDFPAR LFLENSKLEH SEDEGSVLGQ GGSGTVIYRA RYQGQPVAVK RFHIKKFKNF ANVPADTMLR HLRATDAMK NFSEFRQEAS MLHALQHPCI VALIGISIHP LCFALELAPL SSLNTVLSEN A RDSSFIPL GHMLTQKIAY QIASGLAYLH KKNIIFCDLK SDNILVWSLD VKEHINIKLS DY GISRQSF HEGALGVEGT PGYQAPEIRP RIVYDEKVDM FSYGMVLYEL LSGQRPALGH HQL QIAKKL SKGIRPVLGQ PEEVQFRRLQ ALMMECWDTK PEKRPLALSV VSQMKDPTFA TFMY ELCCG KQTAFFSSQG QEYTVVFWDG KEESRNYTVV NTEKGLMEVQ RMCCPGMKVS CQLQV QRSL WTATEDQKIY IYTLKGMCPL NTPQQALDTP AVVTCFLAVP VIKKNSYLVL AGLADG LVA VFPVVRGTPK DSCSYLCSHT ANRSKFSIAD EDARQNPYPV KAMEVVNSGS EVWYSNG PG LLVIDCASLE ICRRLEPYMA PSMVTSVVCS SEGRGEEVVW CLDDKANSLV MYHSTTYQ L CARYFCGVPS PLRDMFPVRP LDTEPPAASH TANPKVPEGD SIADVSIMYS EELGTQILI HQESLTDYCS MSSYSSSPPR QAARSPSSLP SSPASSSSVP FSTDCEDSDM LHTPGAASDR SEHDLTPMD GETFSQHLQA VKILAVRDLI WVPRRGGDVI VIGLEKDSGA QRGRVIAVLK A RELTPHGV LVDAAVVAKD TVVCTFENEN TEWCLAVWRG WGAREFDIFY QSYEELGRLE AC TRKRR |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 10 sec. | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV | ||||||||||||||||||
| 詳細 | Concentrations varied from 2 to 6 uM depending on the grid. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 詳細 | 1 out of the 4 datasets collected was tilted by 20 degrees |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 4 / 平均電子線量: 55.0 e/Å2 詳細: 1 dataset was tilted by 20 degrees. 250 ms frames used |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 36000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)