+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2494 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Substrate Recruitment Pathways in the Yeast Exosome by Electron Microscopy | |||||||||
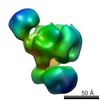
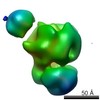
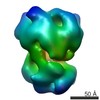
 マップデータ マップデータ | Exosome incubated with ssRNA(14 nucleotides in length) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | exosome / RNA degradation route | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 23.0 Å | |||||||||
 データ登録者 データ登録者 | Liu J-J / Bratkowski MA / Liu XQ / Niu C-Y / Ke AL / Wang H-W | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2014 ジャーナル: Nat Struct Mol Biol / 年: 2014タイトル: Visualization of distinct substrate-recruitment pathways in the yeast exosome by EM. 著者: Jun-Jie Liu / Matthew A Bratkowski / Xueqi Liu / Chu-Ya Niu / Ailong Ke / Hong-Wei Wang /   要旨: The eukaryotic exosome is a multisubunit complex typically composed of a catalytically inactive core and the Rrp44 protein, which contains 3'-to-5' exo- and endo-RNase activities. RNA substrates have ...The eukaryotic exosome is a multisubunit complex typically composed of a catalytically inactive core and the Rrp44 protein, which contains 3'-to-5' exo- and endo-RNase activities. RNA substrates have been shown to be recruited through the core to reach Rrp44's exo-RNase (EXO) site. Using single-particle EM and biochemical analysis, we provide visual evidence that two distinct substrate-recruitment pathways exist. In the through-core route, channeling of the single-stranded substrates from the core to Rrp44 induces a characteristic conformational change in Rrp44. In the alternative direct-access route, this conformational change does not take place, and the RNA substrate is visualized to avoid the core and enter Rrp44's EXO site directly. Our results provide mechanistic explanations for several RNA processing scenarios by the eukaryotic exosome and indicate substrate-specific modes of degradation by this complex. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2494.map.gz emd_2494.map.gz | 2.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2494-v30.xml emd-2494-v30.xml emd-2494.xml emd-2494.xml | 16.7 KB 16.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  2494-RNA14-RE.png 2494-RNA14-RE.png | 65.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2494 http://ftp.pdbj.org/pub/emdb/structures/EMD-2494 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2494 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2494 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2494_validation.pdf.gz emd_2494_validation.pdf.gz | 202.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2494_full_validation.pdf.gz emd_2494_full_validation.pdf.gz | 201.3 KB | 表示 | |
| XML形式データ |  emd_2494_validation.xml.gz emd_2494_validation.xml.gz | 5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2494 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2494 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2494 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2494 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2494.map.gz / 形式: CCP4 / 大きさ: 2.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2494.map.gz / 形式: CCP4 / 大きさ: 2.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Exosome incubated with ssRNA(14 nucleotides in length) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.4 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : Rrp44-Exosome incubated with RNA14
+超分子 #1000: Rrp44-Exosome incubated with RNA14
+分子 #1: Rrp44
+分子 #2: Rrp43
+分子 #3: Rrp4
+分子 #4: Csl4
+分子 #5: Rrp45
+分子 #6: Rrp46-TAP
+分子 #7: Rrp41
+分子 #8: Rrp42
+分子 #9: Mtr3
+分子 #10: Rrp40
+分子 #11: RNA14
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 150mM NaCl, 50mM Tris-HCL,1mM DTT |
| 染色 | タイプ: NEGATIVE 詳細: All samples were diluted at a final concentration of ~80 nM of the exosome in the non-digestive buffer and negatively stained in 2% (w/v) uranyl acetate solution following the standard deep ...詳細: All samples were diluted at a final concentration of ~80 nM of the exosome in the non-digestive buffer and negatively stained in 2% (w/v) uranyl acetate solution following the standard deep stain procedure on holey-carbon coated EM copper grids covered with a thin layer of continuous carbon |
| グリッド | 詳細: 300 mesh continus carbon |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 日付 | 2012年10月10日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GENERIC GATAN (4k x 4k) 実像数: 300 / 平均電子線量: 30 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: OTHER |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 23.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Spider / 使用した粒子像数: 26050 |
|---|---|
| 最終 角度割当 | 詳細: SPIDER: theta 90 degrees, phi 359.9 degrees |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B / Chain - #2 - Chain ID: C / Chain - #3 - Chain ID: D / Chain - #4 - Chain ID: E / Chain - #5 - Chain ID: F / Chain - #6 - Chain ID: J / Chain - #7 - Chain ID: H / Chain - #8 - Chain ID: I / Chain - #9 - Chain ID: J |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















