+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23358 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of E. coli 70S ribosome from EMPIAR-10304 at 4.8 Angstrom Resolution | |||||||||
 マップデータ マップデータ | Structure of E. coli 70S ribosome from EMPIAR-10304 at 4.8 Angstrom Resolution | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
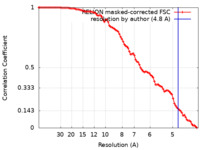
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 4.8 Å | |||||||||
 データ登録者 データ登録者 | Bouvette J / Liu HF / Du X / Zhou Y / Sikkema AP / Mello JFR / Klemm B / Huang R / Schaaper RM / Borgnia MJ / Bartesaghi A | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Beam image-shift accelerated data acquisition for near-atomic resolution single-particle cryo-electron tomography. 著者: Jonathan Bouvette / Hsuan-Fu Liu / Xiaochen Du / Ye Zhou / Andrew P Sikkema / Juliana da Fonseca Rezende E Mello / Bradley P Klemm / Rick Huang / Roel M Schaaper / Mario J Borgnia / Alberto Bartesaghi /  要旨: Tomographic reconstruction of cryopreserved specimens imaged in an electron microscope followed by extraction and averaging of sub-volumes has been successfully used to derive atomic models of ...Tomographic reconstruction of cryopreserved specimens imaged in an electron microscope followed by extraction and averaging of sub-volumes has been successfully used to derive atomic models of macromolecules in their biological environment. Eliminating biochemical isolation steps required by other techniques, this method opens up the cell to in-situ structural studies. However, the need to compensate for errors in targeting introduced during mechanical navigation of the specimen significantly slows down tomographic data collection thus limiting its practical value. Here, we introduce protocols for tilt-series acquisition and processing that accelerate data collection speed by up to an order of magnitude and improve map resolution compared to existing approaches. We achieve this by using beam-image shift to multiply the number of areas imaged at each stage position, by integrating geometrical constraints during imaging to achieve high precision targeting, and by performing per-tilt astigmatic CTF estimation and data-driven exposure weighting to improve final map resolution. We validated our beam image-shift electron cryo-tomography (BISECT) approach by determining the structure of a low molecular weight target (~300 kDa) at 3.6 Å resolution where density for individual side chains is clearly resolved. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23358.map.gz emd_23358.map.gz | 8.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23358-v30.xml emd-23358-v30.xml emd-23358.xml emd-23358.xml | 15.5 KB 15.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23358_fsc.xml emd_23358_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23358.png emd_23358.png | 76.3 KB | ||
| マスクデータ |  emd_23358_msk_1.map emd_23358_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_23358_additional_1.map.gz emd_23358_additional_1.map.gz emd_23358_half_map_1.map.gz emd_23358_half_map_1.map.gz emd_23358_half_map_2.map.gz emd_23358_half_map_2.map.gz | 54.8 MB 32.7 MB 32.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23358 http://ftp.pdbj.org/pub/emdb/structures/EMD-23358 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23358 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23358 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23358_validation.pdf.gz emd_23358_validation.pdf.gz | 546.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23358_full_validation.pdf.gz emd_23358_full_validation.pdf.gz | 545.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23358 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23358 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23358 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23358 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23358.map.gz / 形式: CCP4 / 大きさ: 9.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23358.map.gz / 形式: CCP4 / 大きさ: 9.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of E. coli 70S ribosome from EMPIAR-10304 at 4.8 Angstrom Resolution | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_23358_msk_1.map emd_23358_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
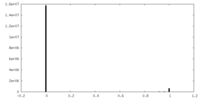
| 密度ヒストグラム |
-追加マップ: Structure of E. coli 70S ribosome from EMPIAR-10304...
| ファイル | emd_23358_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of E. coli 70S ribosome from EMPIAR-10304 at 4.8 Angstrom Resolution | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
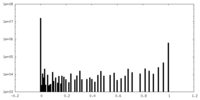
| 密度ヒストグラム |
-ハーフマップ: Structure of E. coli 70S ribosome from EMPIAR-10304...
| ファイル | emd_23358_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of E. coli 70S ribosome from EMPIAR-10304 at 4.8 Angstrom Resolution | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
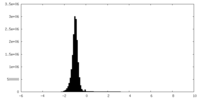
| 密度ヒストグラム |
-ハーフマップ: Structure of E. coli 70S ribosome from EMPIAR-10304...
| ファイル | emd_23358_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of E. coli 70S ribosome from EMPIAR-10304 at 4.8 Angstrom Resolution | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
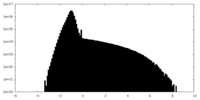
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 70S ribosome
| 全体 | 名称: 70S ribosome |
|---|---|
| 要素 |
|
-超分子 #1: 70S ribosome
| 超分子 | 名称: 70S ribosome / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 200 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 4092 pixel / デジタル化 - サイズ - 縦: 5760 pixel / 平均電子線量: 3.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)