+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23200 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
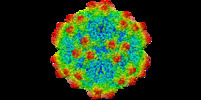
| タイトル | The genome-containing AAV12 capsid | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Icosahedral Capsid / AAV12 / Adeno-associated virus / Parvovirus / Gene Therapy / VIRUS | |||||||||
| 機能・相同性 | Phospholipase A2-like domain / Phospholipase A2-like domain / Parvovirus coat protein VP2 / Parvovirus coat protein VP1/VP2 / Parvovirus coat protein VP2 / Capsid/spike protein, ssDNA virus / T=1 icosahedral viral capsid / structural molecule activity / VP1 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Adeno-associated virus 12 (アデノ随伴ウイルス) Adeno-associated virus 12 (アデノ随伴ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.67 Å | |||||||||
 データ登録者 データ登録者 | Mietzsch M / Agbandje-McKenna M | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Viruses / 年: 2021 ジャーナル: Viruses / 年: 2021タイトル: Completion of the AAV Structural Atlas: Serotype Capsid Structures Reveals Clade-Specific Features. 著者: Mario Mietzsch / Ariana Jose / Paul Chipman / Nilakshee Bhattacharya / Nadia Daneshparvar / Robert McKenna / Mavis Agbandje-McKenna /  要旨: The capsid structures of most Adeno-associated virus (AAV) serotypes, already assigned to an antigenic clade, have been previously determined. This study reports the remaining capsid structures of ...The capsid structures of most Adeno-associated virus (AAV) serotypes, already assigned to an antigenic clade, have been previously determined. This study reports the remaining capsid structures of AAV7, AAV11, AAV12, and AAV13 determined by cryo-electron microscopy and three-dimensional image reconstruction to 2.96, 2.86, 2.54, and 2.76 Å resolution, respectively. These structures complete the structural atlas of the AAV serotype capsids. AAV7 represents the first clade D capsid structure; AAV11 and AAV12 are of a currently unassigned clade that would include AAV4; and AAV13 represents the first AAV2-AAV3 hybrid clade C capsid structure. These newly determined capsid structures all exhibit the AAV capsid features including 5-fold channels, 3-fold protrusions, 2-fold depressions, and a nucleotide binding pocket with an ordered nucleotide in genome-containing capsids. However, these structures have viral proteins that display clade-specific loop conformations. This structural characterization completes our three-dimensional library of the current AAV serotypes to provide an atlas of surface loop configurations compatible with capsid assembly and amenable for future vector engineering efforts. Derived vectors could improve gene delivery success with respect to specific tissue targeting, transduction efficiency, antigenicity or receptor retargeting. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23200.map.gz emd_23200.map.gz | 262.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23200-v30.xml emd-23200-v30.xml emd-23200.xml emd-23200.xml | 14.7 KB 14.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23200.png emd_23200.png | 323.8 KB | ||
| Filedesc metadata |  emd-23200.cif.gz emd-23200.cif.gz | 6.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23200 http://ftp.pdbj.org/pub/emdb/structures/EMD-23200 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23200 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23200 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23200_validation.pdf.gz emd_23200_validation.pdf.gz | 703.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23200_full_validation.pdf.gz emd_23200_full_validation.pdf.gz | 703 KB | 表示 | |
| XML形式データ |  emd_23200_validation.xml.gz emd_23200_validation.xml.gz | 7.6 KB | 表示 | |
| CIF形式データ |  emd_23200_validation.cif.gz emd_23200_validation.cif.gz | 8.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23200 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23200 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23200 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23200 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23200.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23200.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.082 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Adeno-associated virus 12
| 全体 | 名称:  Adeno-associated virus 12 (アデノ随伴ウイルス) Adeno-associated virus 12 (アデノ随伴ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Adeno-associated virus 12
| 超分子 | 名称: Adeno-associated virus 12 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1 / NCBI-ID: 235458 / 生物種: Adeno-associated virus 12 / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: VP1
| 分子 | 名称: VP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 60 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Adeno-associated virus 12 (アデノ随伴ウイルス) Adeno-associated virus 12 (アデノ随伴ウイルス) |
| 分子量 | 理論値: 58.549543 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DGVGNASGDW HCDSTWSEGR VTTTSTRTWV LPTYNNHLYL RIGTTANSNT YNGFSTPWGY FDFNRFHCHF SPRDWQRLIN NNWGLRPKS MRVKIFNIQV KEVTTSNGET TVANNLTSTV QIFADSTYEL PYVMDAGQEG SFPPFPNDVF MVPQYGYCGV V TGKNQNQT ...文字列: DGVGNASGDW HCDSTWSEGR VTTTSTRTWV LPTYNNHLYL RIGTTANSNT YNGFSTPWGY FDFNRFHCHF SPRDWQRLIN NNWGLRPKS MRVKIFNIQV KEVTTSNGET TVANNLTSTV QIFADSTYEL PYVMDAGQEG SFPPFPNDVF MVPQYGYCGV V TGKNQNQT DRNAFYCLEY FPSQMLRTGN NFEVSYQFEK VPFHSMYAHS QSLDRMMNPL LDQYLWHLQS TTTGNSLNQG TA TTTYGKI TTGDFAYYRK NWLPGACIKQ QKFSKNANQN YKIPASGGDA LLKYDTHTTL NGRWSNMAPG PPMATAGAGD SDF SNSQLI FAGPNPSGNT TTSSNNLLFT SEEEIATTNP RDTDMFGQIA DNNQNATTAP HIANLDAMGI VPGMVWQNRD IYYQ GPIWA KVPHTDGHFH PSPLMGGFGL KHPPPQIFIK NTPVPANPNT TFSAARINSF LTQYSTGQVA VQIDWEIQKE HSKRW NPEV QFTSNYGTQN SMLWAPDNAG NYHELRAIGS RFLTHHL UniProtKB: VP1 |
-分子 #2: 2'-DEOXYADENOSINE-5'-MONOPHOSPHATE
| 分子 | 名称: 2'-DEOXYADENOSINE-5'-MONOPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 60 / 式: D5M |
|---|---|
| 分子量 | 理論値: 331.222 Da |
| Chemical component information | 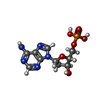 ChemComp-D5M: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






























 Z (Sec.)
Z (Sec.) X (Row.)
X (Row.) Y (Col.)
Y (Col.)





















